结核病是危害人类健康的重要传染病之一。2015年世界范围内新发结核病病例估计有1 040万,其中死亡140万,耐药结核病新发病例约48万[1]。有效的药物治疗是防治结核病的关键,如利福平、异烟肼、吡嗪酰胺等一线药物联合使用的直接督导下短程化疗(directly observed treatment short course,DOTS)方案,使人类在与结核病抗争中取得了暂时性胜利。然而,耐多药结核(multidrug-resistant tuberculosis,MDR-TB)、广泛耐药结核(extensively drug-resistant tuberculosis,XDR-TB)及利福平耐药结核(rifampicin-resistant tuberculosis,RR-TB)的大量出现,表明研发具有新作用机制的抗结核药物仍很迫切。
贝塔喹啉(TMC-207)是一个具有良好抗结核活性的新药,通过抑制结核分枝杆菌的ATP合成酶而发挥活性[2-4],全新的作用靶标及良好的抗结核活性使其成为抗耐药结核的希望。本实验室前期对容量为5万个小分子化合物的化合物库进行了抗结核活性初步筛选,并对具有抗结核活性的小分子化合物进行了系列优化改造,最终获得数十个具有抗耐药结核活性的新型小分子化合物[5]。HY-152E是其中一个具有良好抗结核活性的化合物(MIC≤0.09 μg/mL)[6],为进一步研究其抗结核机制,本研究拟对其作用靶标进行分析。
目前,药物作用靶蛋白的研究方法包括蛋白质芯片(protein chip)测定[7]、药物亲和反应靶点稳定性(drug affinity responsive target stability,DARTS)检测[8]、基于氧化速率的蛋白稳定性(stability of proteins from rates of oxidation,SPROX)检测[9]、热力学蛋白质组分析(thermal proteome profiling,TPP)[10]等技术。其中DARTS技术利用结合药物分子后的靶蛋白对链霉菌蛋白酶的稳定性相对提高而抵抗链霉菌蛋白酶消化的原理,筛选可能与药物分子结合的潜在靶标分子(图 1)。其不需对药物分子进行修饰,且系统中蛋白保有天然活性,是近年来较好的一种研究药物靶标的方法。本文即采用DARTS技术并结合蛋白质谱学技术和生物信息学方法来探索分析活性化合物HY-152E可能的抗结核靶标分子。
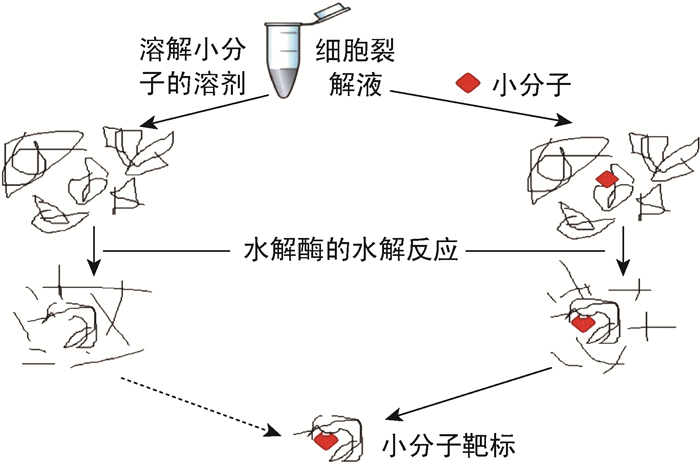
|
| 图 1 药物亲和反应靶点稳定性的作用原理 Fig. 1 The principle of drug affinity responsive target stability (DARTS) assay |
化合物HY-152E委托上海皓元化学科技有限公司合成,溶解于二甲亚砜(dimethyl sulfoxide,DMSO)中。Middlebrook 7H9和7H10为BD公司产品;BCA蛋白定量试剂盒为北京艾德莱生物科技有限公司产品;蛋白酶抑制剂购自Roche Diagnostics公司,取cOmpleteTMMini一片,用500 μL无菌水溶解配制为10 mg/mL的溶液,-20 ℃保存;链霉菌蛋白酶为Roche Diagnostics产品,用TNC溶液(50 mmol/L Tris-HCl pH 8.0、50 mmol/L NaCl、10 mmol/L CaCl2)溶解,母液浓度为10 mg/mL,-20 ℃保存,使用时按所需浓度进行稀释。其他试剂均为实验室常用分析纯试剂。
1.2 方法 1.2.1 菌株及培养H37Ra菌株为本实验室保存,用接种环划线接种于7H10-10% ADC中,37 ℃培养箱中静置培养2~3周,再用接种环刮取菌落至灭菌的EP管中,-30 ℃保存待用。
1.2.2 H37Ra裂解液的制备细菌裂解液体系:0.4% Triton X-100、400 mmol/L NaCl、100 mmol/L Tris-HCl (pH 7.5)和20%甘油。将300 μL细菌裂解液加入收集的菌体中,为防止破壁不充分,轻轻吹打至无结块菌体;加入200~300 μL玻璃砂、150 μL磷酸酶抑制剂(40 mmol/L Na4P2O7、200 mmol/L NaF、0.4 mmol/L Na3VO4,-20 ℃保存)、30 μL蛋白酶抑制剂,补无菌水至总体积为600 μL;用FastPrep 24匀浆机破碎1 min(速率6 m/s,45 s/次),冰上放置1 min,循环12~14次;4 ℃ 13 000~15 000 r/min充分离心10~15 min,确保上清液中无玻璃砂或蛋白沉淀物,收集上清液于冰上备用。用BCA蛋白定量试剂盒对上清液总蛋白进行定量(按试剂盒说明书进行)。
1.2.3 不同浓度HY-152E的亲和反应靶点稳定性检测将细菌蛋白裂解液样品用去离子水稀释至浓度为4~6 μg/μL,分装于1.5 mL EP管中,每管50 μL。每管加入不同浓度HY-152E(终浓度分别为0.6、1.2、3.0、4.8、6.0、9.0 mg/mL),阴性对照为DMSO。轻轻颠倒混匀,避免剧烈操作影响蛋白的活性结构,室温孵育1 h。加入链霉菌蛋白酶1.25 μg,轻轻混匀,室温消化45 min,加入上样缓冲液,沸水煮10 min终止酶切反应,进行10%十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)。比较HY-152E组与对照组SDS-PAGE结果,寻找差异蛋白条带。上述实验重复3次,寻找重复性高的差异条带。
1.2.4 链霉菌蛋白酶的浓度及作用时间对亲和反应靶点稳定性的影响选择终浓度为9.0 mg/mL的HY-152E,与菌体蛋白(250 μg)室温孵育1 h。加入不同浓度的链霉菌蛋白酶(含量分别为2.5、1.25、0.625、0.312 5、0.25 μg),使最终链霉菌蛋白酶(μg):菌体蛋白(μg)为1:100、1:200、1:400、1:800、1:1 000。轻轻混匀,分别于室温消化30、45、60 min,加入SDS-PAGE上样缓冲液,沸水煮10 min终止反应,进行12% SDS-PAGE。比较不同链霉菌蛋白酶浓度、不同水解时间下HY-152E组与DMSO对照组的SDS-PAGE结果,寻找差异蛋白条带。
1.2.5 差异条带质谱分析将蛋白水解后SDS-PAGE显示重复性高的差异蛋白进行切胶回收,用超高压纳升液相-高分辨率质谱联用仪进行蛋白质谱分析,比较分析蛋白质谱数据结果中HY-152E组与阴性对照组的差异多肽片段。
2 结果 2.1 不同浓度HY-152E亲和反应靶点稳定性的检测选择不同浓度的HY-152E(终浓度分别为0.6、1.2、3.0、4.8、6.0、9.0 mg/mL)及DMSO,与结核分枝杆菌菌体蛋白孵育。在链霉菌蛋白酶作用浓度为1:200(1.25 μg)时,室温消化45 min后电泳结果(图 2)显示,在HY-152E作用下,相对分子质量70 000左右及40 000~55 000处分别有一条明显的差异蛋白。在HY-152E作用下,差异蛋白条带更清晰,且随着浓度降低,蛋白条带变弱,而DMSO对照组中蛋白条带完全被水解,表明HY-152E可与该差异蛋白结合并保护蛋白耐受链霉菌蛋白酶的作用。
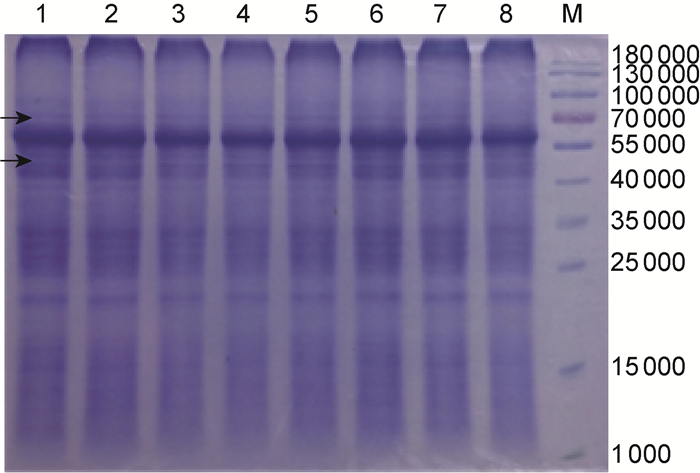
|
| Lanes 1-7 corresponding to the final concentrations of compound HY-152E: 9.0, 6.0, 4.8, 3.0, 1.2, 0.6 mg/mL. Hydrolysis time: 45 min; streptomycin protease concentration: 1:200 (1.25 μg). Lane 8: DMSO control without compound HY-152E. 图 2 DARTS检测HY-152E与结核分枝杆菌菌体蛋白结合 Fig. 2 DARTS experiment for analysis of compound HY-152E binding to protein targets of Mycobacterium tuberculosis |
为确定HY-152E对上述差异蛋白耐链霉菌蛋白酶保护作用的真实性,进一步分析不同浓度链霉菌蛋白酶及不同水解时间对HY-152E与蛋白结合并保护差异蛋白耐受链霉菌蛋白酶作用的影响。同时,为使目标差异条带更清晰,将10%蛋白胶浓度(1.2.3)改为12%(1.2.4)。图 3显示了菌体蛋白与终浓度为9.0 mg/mL的HY-152E互作后,在不同浓度链霉菌蛋白酶作用下分别水解30、45和60 min的结果。与图 1结果相同,在相对分子质量70 000左右和45 000~55 000处分别有一条明显的差异条带,且随着链霉菌蛋白酶浓度变化,差异条带呈现明显的浓度梯度差异。作用时间相同时,随着链霉菌蛋白酶浓度降低,蛋白水解程度也降低;链霉菌蛋白酶被稀释至1:400时,水解程度已不明显(图 3B及3C第3泳道)。链霉菌蛋白酶浓度相同时,随着水解时间增加,靶蛋白消化程度加深(图 3A、3B及3C第4泳道),但60 min时靶蛋白经HY-152E稳定后消化程度基本不再变化。基于DARTS作用原理,蛋白-分子耦合物的稳定性比单独蛋白高,可抵抗蛋白酶的消化作用。上述结果推测,具有抗结核活性的HY-152E可能是与差异蛋白结合,从而保护差异蛋白耐受链霉菌蛋白酶的水解作用。

|
| The final concentration of compound HY-152E is 9.0 mg/mL. Lanes 1-5 corresponding to streptomycin protease concentrations of 1:1 000, 1:800, 1:400, 1:200, 1:100; no streptomycin protease in lane 6. Hydrolysis time: 30 min in A, 45 min in B, 60 min in C. 图 3 链霉菌蛋白酶浓度及作用时间对亲和反应靶点稳定性的影响 Fig. 3 Effects of concentration and digestion time of streptomycin protease on DARTS results |
比较HY-152E组与DMSO对照组的SDS-PAGE差异蛋白,将相同位置的胶切割回收。DARTS技术原理表明,相比于可结合蛋白,如果蛋白无法与小分子结合,其稳定性低,水解过程中会发生降解。因此,通过比较蛋白质谱的数据,去除HY-152E组和对照组中共有的多肽片段,保留只在HY-152E组出现的肽段信息,并根据蛋白相对分子质量筛选70 000左右及45 000~55 000处的蛋白。质谱数据来源于两次独立实验样品,通过UniProt蛋白数据库搜索,显示70 000左右的蛋白共计52个,45 000~55 000处的蛋白共计34个。
结合两次蛋白质谱测定结果,利用TB Database基因数据库,分析对应蛋白的编码基因在结核分枝杆菌生长中的必需性及基因生理功能信息,并结合蛋白相对分子质量、蛋白肽段可信度的综合评分,筛选出9个可能是HY-152E抗结核作用的潜在靶蛋白(表 1),供后续进一步研究。
| Molecular weight (×1 000) |
Gene | Protein |
| 109.070 5 | glnE | Glutamate-ammonia ligase adenylyltransferase |
| 98.411 6 | polA | DNA polymerase Ⅰ |
| 96.856 1 | dnaB | Replicative DNA helicase |
| 71.461 4 | glfT | Galactofuranosyl transferase GlfT2 |
| 53.418 2 | gltD | Glutamate synthase [NADPH] small chain |
| 50.360 8 | lgt | Prolipoprotein diacylglyceryl transferase |
| 46.753 4 | clpX | ATP-dependent Clp protease ATP-binding subunit |
| 46.273 9 | hisD | Histidinol dehydrogenase |
| 44.035 5 | murA | UDP-N-acetylglucosamine 1-carboxyvinyltransferase |
药物靶标的确定是筛选药物的基础,新的药物靶标有助于筛选出结构和作用机制全新的药物,因而寻找切实可靠的药物靶标一直是药物研究的关键,而基于活性药物或化合物筛选的药物靶标具有切实的可靠性。本研究在前期筛选的具有良好抗结核活性化合物的基础上开展药物靶标研究,对筛选新型抗结核药物和药物靶标具有重要价值。
98%以上的药物靶标属于蛋白。从理论上说,作为药物靶标的蛋白必须能以适当的化学特性和亲和力结合小分子化合物[11]。近年来,大量分子生物学技术的出现,尤其是基因组学、生物信息学、蛋白质组学、质谱联用技术及生物大分子相互作用分析(biospecific interaction analysis,BIA)等推动了从纷繁复杂的细胞内生物大分子中发现特异性药物作用靶标分子的进程[12]。靶标的寻找通常在基因数据库、转录水平及蛋白水平进行。Lomenick等[8]提出的DARTS技术是在蛋白水平开展的靶标研究,不仅能避免因标记或修饰导致小分子丧失活性的问题,保持蛋白的天然活性,还能避免基因数据库大量信息处理过程,比较简单易行。
本研究采用未进行分子修饰的DARTS技术确定靶标,研究的蛋白来自结核分枝杆菌总蛋白。查阅已发表的文献资料发现,利用DARTS技术筛选药物作用靶标的对象全部为真核细胞[13-17]。因此,本研究以原核生物为研究对象,尤其是对细胞壁和脂质层较厚的结核分枝杆菌而言,菌体蛋白的前期处理及蛋白使用浓度是较重要的环节。DARTS实验中蛋白浓度要求为4~6 μg/μL[8],蛋白浓度太低或太高均影响实验结果,且蛋白浓度不准确不利于寻找差异蛋白条带。经摸索,本研究按3~4环(刮菌环)菌量,用300 μL Triton X-100细胞裂解液提取相应蛋白,所得蛋白浓度基本符合要求。DARTS实验所用裂解液必须新鲜,需避免剧烈振摇,以免引起蛋白变性,进而影响其与药物的结合,而HY-152E溶解于DMSO。因此,为防止DMSO在低温下凝结,室温下进行化合物与菌体蛋白孵育有助于实验的良好重复性。本研究发现,HY-152E的最佳浓度>6.0 μg/μL,链霉菌蛋白酶的最佳用量为蛋白酶:菌体蛋白=1:100或1:200,链霉菌蛋白酶水解时间要控制在45~60 min。此外,在菌体蛋白处理过程中使用了磷酸酶和蛋白酶抑制剂,这两种抑制剂对链霉菌蛋白酶(来源于灰色链霉菌)的消化功能无明显影响[8]。
HY-152E潜在靶标蛋白在结核分枝杆菌的生理功能方面发挥重要作用。如ClpX为Clp蛋白酶ATP结合亚基部分,参与结核分枝杆菌蛋白的正确折叠、降解错误蛋白等[18-19];DnaB和PolA分别是DNA复制过程中的解旋酶和DNA聚合酶Ⅰ,控制细菌DNA的解旋和染色体的起始及延伸,是细菌生长繁殖的关键[20-21];与肽聚糖生物合成相关的UDP-N-乙酰葡糖胺-羧基乙烯基转移酶(MurA)及组氨酸生物合成途径关键酶——组氨醇脱氢酶(HisD)均为结核分枝杆菌物质合成代谢的关键蛋白[22-23]。Gene Ontology分析发现,这些蛋白主要富集于结核分枝杆菌的细胞壁和胞质中。目前,很多化合物对结核分枝杆菌的弱杀菌或无杀菌活性缘其很难透过结核分枝杆菌较厚的细胞壁,而本研究中筛选的HY-152E潜在靶蛋白主要集中于细胞壁,这对筛选具有良好活性的抗结核新药的研究具有十分重要的意义。
本研究中,仅以DARTS技术通过比较实验组与对照组之间蛋白质谱数据的差异肽段来确定潜在靶标存在一定的局限性,未能实现定量比较。这主要是由于结核分枝杆菌在培养过程中没有采用标记培养基,如果能利用SILAC或稳定同位素标记培养基来培养细菌,后续只需分别将等量实验组和对照组的蛋白混合进行DARTS技术处理,从而在蛋白质谱分析时可较准确地对差异蛋白进行定量分析。因此,本研究确定的9个潜在的HY-152E作用靶标还需进一步在体外靶向结合能力及靶蛋白生物功能影响方面进行深入验证。
| [1] |
WHO. Global tuberculosis report 2016 [R/OL]. http://www.who.int/tb/publications/global_report/archive/en.
|
| [2] |
Andries K, Verhasselt P, Guillemont J, Göhlmann HW, Neefs JM, Winkler H, Van Gestel J, Timmerman P, Zhu M, Lee E, Williams P, de Chaffoy D, Huitric E, Hoffner S, Cambau E, Truffot-Pernot C, Lounis N, Jarlier V. A diarylquinoline drug active on the ATP synthase of Mycobacterium tuberculosis[J]. Science, 2005, 307(5707): 223-227.
[DOI]
|
| [3] |
Diacon AH, Pym A, Grobusch M, Patientia R, Rustomjee R, Page-Shipp L, Pistorius C, Krause R, Bogoshi M, Churchyard G, Venter A, Allen J, Palomino JC, De Marez T, van Heeswijk RP, Lounis N, Meyvisch P, Verbeeck J, Parys W, de Beule K, Andries K, Mc Neeley DF. The diarylquinoline TMC207 for multidrug-resistant tuberculosis[J]. N Engl J Med, 2009, 360(23): 2397-2405.
[DOI]
|
| [4] |
Huitric E, Verhasselt P, Koul A, Andries K, Hoffner S, Andersson DI. Rates and mechanisms of resistance development in Mycobacterium tuberculosis to a novel diarylquinoline ATP synthase inhibitor[J]. Antimicrob Agents Chemother, 2010, 54(3): 1022-1028.
[DOI]
|
| [5] |
Luo R, Laitinen T, Teng L, Nevalainen T, Lahtela-Kakkonen M, Zheng B, Wang H, Poso A, Zhang X. Synthesis and biological evaluation of arylthiourea derivatives with antitubercular activity[J]. Lett Drug Des Discov, 2013, 10(7): 640-650.
[DOI]
|
| [6] |
滕丽艳, 刘霞, 张雪莲, 王洪海. 新型先导化合物E体外抗结核分枝杆菌活性的研究[J]. 微生物与感染, 2012, 7(4): 208-212. [URI]
|
| [7] |
Parasido EM, Silvestri A, Canzonieri V, Belluco C, Diodoro MG, Milione M, Melotti F, De Maria R, Liotta L, Petricoin EF, Pierobon M. Protein drug target activation homogeneity in the face of intra-tumor heterogeneity: implications for precision medicine[J]. Oncotarget, 2017, 8(30): 48534-48544.
[URI]
|
| [8] |
Lomenick B, Hao R, Jonai N, Chin RM, Aghajan M, Warburton S, Wang J, Wu RP, Gomez F, Loo JA, Wohlschlegel JA, Vondriska TM, Pelletier J, Herschman HR, Clardy J, Clarke CF, Huang J. Target identification using drug affinity responsive target stability (DARTS)[J]. Proc Natl Acad Sci USA, 2009, 106(51): 21984-21989.
[DOI]
|
| [9] |
Strickland EC, Geer MA, Tran DT, Adhikari J, West GM, Dearmond PD, Xu Y, Fitzgerald MC. Thermodynamic analysis of protein-ligand binding interactions in complex biological mixtures using the stability of proteins from rates of oxidation[J]. Nat Protoc, 2013, 8(1): 148-161.
[URI]
|
| [10] |
Franken H, Mathieson T, Childs D, Sweetman GM, Werner T, Tögel I, Doce C, Gade S, Bantscheff M, Drewes G, Reinhard FB, Huber W, Savitski MM. Thermal proteome profiling for unbiased identification of direct and indirect drug targets using multiplexed quantitative mass spectrometry[J]. Nat Protoc, 2015, 10(10): 1567-1593.
[DOI]
|
| [11] |
周喆, 王升启. 药物靶标搜寻和验证方法的研究进展[J]. 国外医学:药学分册, 2005, 32(3): 145-149. [CNKI]
|
| [12] |
杨建雄, 刘志辉. 药物靶标在新药研发中的作用[J]. 时珍国医国药, 2009, 20(3): 750-751. [CNKI]
|
| [13] |
Gotsbacher MP, Cho S, Kwon HJ, Karuso P. Daptomycin, a last-resort antibiotic, binds ribosomal protein S19 in humans[J]. Proteome Sci, 2017, 15: 16.
[DOI]
|
| [14] |
Kim D, Hwang HY, Kim JY, Lee JY, Yoo JS, Marko-Varga G, Kwon HJ. FK506, an immunosuppressive drug, induces autophagy by binding to the V-ATPase catalytic subunit A in neuronal cells[J]. J Proteome Res, 2017, 16(1): 55-64.
[DOI]
|
| [15] |
Kost GC, Yang MY, Li L, Zhang Y, Liu CY, Kim DJ, Ahn CH, Lee YB, Liu ZR. A novel anti-cancer agent, 1-(3, 5-dimethoxyphenyl)-4-[(6-fluoro-2-methoxyquinoxalin-3-yl)aminocarbonyl] piperazine (RX-5902), interferes with β-catenin function through Y593 phospho-p68 RNA helicase[J]. J Cell Biochem, 2015, 116(8): 1595-1601.
[DOI]
|
| [16] |
Park YD, Sun W, Salas A, Antia A, Carvajal C, Wang A, Xu X, Meng Z, Zhou M, Tawa GJ, Dehdashti J, Zheng W, Henderson CM, Zelazny AM, Williamson PR. Identification of multiple cryptococcal fungicidal drug targets by combined gene dosing and drug affinity responsive target stability screening[J]. MBio, 2016, 7(4): e01073-16.
[DOI]
|
| [17] |
Qu Y, Gharbi N, Yuan X, Olsen JR, Blicher P, Dalhus B, Brokstad KA, Lin B, Øyan AM, Zhang W, Kalland KH, Ke X. Axitinib blocks Wnt/β-catenin signaling and directs asymmetric cell division in cancer[J]. Proc Natl Acad Sci USA, 2016, 113(33): 9339-9344.
[DOI]
|
| [18] |
Ingvarsson H, Maté MJ, Högbom M, Portnoï D, Benaroudj N, Alzari PM, Ortiz-Lombardía M, Unge T. Insights into the inter-ring plasticity of caseinolytic proteases from the X-ray structure of Mycobacterium tuberculosis ClpP1[J]. Acta Crystallogr D Biol Crystallogr, 2007, 63(Pt 2): 249-259.
[URI]
|
| [19] |
Sherrid AM, Rustad TR, Cangelosi GA, Sherman DR. Characterization of a Clp protease gene regulator and the reaeration response in Mycobacterium tuberculosis[J]. PLoS One, 2010, 5(7): e11622.
[DOI]
|
| [20] |
Xie Y, He ZG. Characterization of physical interaction between replication initiator protein DnaA and replicative helicase from Mycobacterium tuberculosis H37Rv[J]. Biochemistry (Mosc), 2009, 74(12): 1320-1327.
[DOI]
|
| [21] |
Arrigo CJ, Singh K, Modak MJ. DNA polymerase Ⅰ of Mycobacterium tuberculosis: functional role of a conserved aspartate in the hinge joining the M and N helices[J]. J Biol Chem, 2002, 277(3): 1653-1661.
[DOI]
|
| [22] |
Babajan B, Chaitanya M, Rajsekhar C, Gowsia D, Madhusudhana P, Naveen M, Chitta SK, Anuradha CM. Comprehensive structural and functional characterization of Mycobacterium tuberculosis UDP-NAG enolpyruvyl transferase (Mtb-MurA) and prediction of its accurate binding affinities with inhibitors[J]. Interdiscip Sci, 2011, 3(3): 204-216.
[DOI]
|
| [23] |
Lunardi J, Martinelli LKB, Raupp AS, Nunes JES, Rostirolla DC, Timmers LFSM, Villela AD, Pissinate K, Limberger J, de Souza ON, Basso LA, Santos DS, Machado P. Mycobacterium tuberculosis histidinol dehydrogenase: biochemical characterization and inhibition studies[J]. RSC Adv, 2016, 6(34): 28406-28418.
[DOI]
|
 2018, Vol. 13
2018, Vol. 13


