结核病是由结核分枝杆菌引起的传染性疾病。相比于敏感株,结核分枝杆菌耐药株的传播力是研究热点之一[1]。本课题组前期收集了55株2003—2009年于重庆市肺科医院诊断的泛耐药结核分枝杆菌(extensively drug-resistant Mycobacterium tuberculosis,XDR-TB)菌株,但由于技术上的限制,2014年仅用传统的多位点数目可变串联重复序列(variable-number tandem-repeat,VNTR)分型对其传播及成簇特征进行了分析,发现该地区XDR-TB的传播率较高[2]。
VNTR分型方法因简便易行、结果可读性强、实验室间可比性高,广泛应用于结核分枝杆菌的传播研究[3-4]。近年来,随着技术的进步、测序成本的降低,全基因组测序(whole-genome sequencing,WGS)技术开始用于研究结核分枝杆菌的传播,已有多项研究证明WGS较VNTR分型具有更高的分辨率[5-6]。但目前应用WGS鉴定结核病传播的研究主要集中于敏感菌株及欧美菌株[7-9],我国研究较少[10],且尚未在XDR-TB中进行相关研究。因此,本研究应用WGS对上述菌株进行重新分析,并评价VNTR分型判断XDR基因型分布及成簇特征的准确性。
1 材料与方法 1.1 研究对象收集2003—2009年重庆市肺科医院诊断的55株XDR-TB菌株。该医院于2003—2009年共诊断2 727例结核病,其中624例为多重耐药结核分枝杆菌(multidrug-resistant Mycobacterium tuberculosis,MDR-TB),85例为XDR-TB(已去重)。2015年共回溯、成功复苏、完成药敏试验和VNTR分型的菌株有95株[2]。本次成功复苏并完成WGS的XDR-TB菌株有55株。
1.2 研究方法 1.2.1 菌株选取对55株XDR-TB菌株进行复苏,并进行DNA抽提。
1.2.2 VNTR分型对成功复苏并完成WGS的55株菌株进行9+3个位点的VNTR分型,纳入的9个位点为QUB-11b、QUB-18、QUB-26、MIRU26、Mtub21、Mtub04、MIRU31、MIRU40、VNTR2372,3个高变位点为VNTR3232、VNTR3820、VNTR4120[11]。聚合酶链反应(polymerase chain reaction,PCR)产物用2%琼脂糖凝胶分析。
计算每个位点重复序列的拷贝数,在VNTRplus在线分析平台(http://www.VNTRplus.org/MIRU/index.faces)上,用UPGMA算法构建系统发生树。具有完全相同MIRU-VNTR基因型的结核分枝杆菌分离株≥2株即被认为成簇。
1.2.3 WGS分型对所有菌株DNA进行文库构建,基于Illumina X10平台进行测序,深度至少为200×,用Bowtie 2软件将测序reads比对到H37Rv (NC_000962.3)参考序列,覆盖率为99.21%~99.89%。用SAMtools进行SNP检测,若某位点的突变型比例在80%以上即认为是固定突变。分析菌株间的遗传距离时,去除PE、PPE等高度重复区域,GC富集序列及耐药相关基因SNP,且SNP需测得至少10个reads无正负链偏差。基于SNP结果,用MEGA 7软件以最大似然法构建系统发育树,以H37Rv为根。根据现有研究,规定遗传距离相差不超过12个SNP为成簇,即有近期传播关系[7, 10]。
2 结果 2.1 菌株信息本研究最终纳入55株2003—2009年重庆市肺科医院诊断为XDR-TB的菌株。当时患者基本资料如下:平均年龄15~82(47±18)岁,其中男性21例(38.2%)。均有4个一线抗结核药物[异烟肼(H)、利福平(R)、链霉素(S)、乙胺丁醇(E)]和3个二线抗结核药物[氧氟沙星(O)、卡那霉素(K)、卷曲霉素(C)]的表型药敏试验结果,耐药分布特征为HRSOK 4例、HRSOC 2例、HRSOKC 5例、HRSEOK 10例、HRSEOC 6例、HRSEOKC 28例。
2.2 VNTR分型结果55株结核分枝杆菌通过VNTR分型方法可分为45个基因型。39株为单一基因型,16株(29.1%)分别归入6个基因簇,即C1、C2、C3、C4-1、C4-2、C4-3,每个簇分别含有5、2、3、2、2、2株分离株。用UPGMA算法构建系统发育树(图 1)。
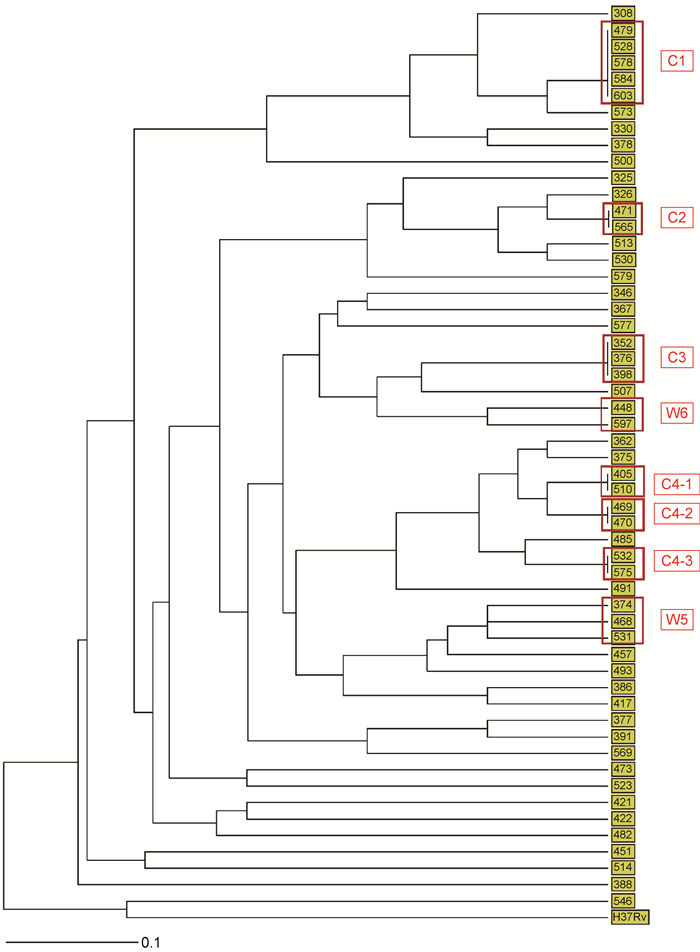
|
| 注:红色框内是用VNTR、WGS分型方法鉴定的簇,其中C簇是VNTR分型鉴定出的簇,W簇是WGS分型鉴定出的簇。 图 1 基于VNTR分型方法构建进化树 Fig. 1 Phylogenetic tree based on VNTR typing |
根据SNP,将55株菌株分为2个谱系[12],其中53株为Lineage 2(East-Asian),2株(Q546、Q388)为Lineage 4(Euro-American)。
根据现有研究,规定遗传距离相差不超过12个SNP为成簇,可将55株菌株中的20株(36.4%)分为5个簇,即W1、W2、W4、W5、W6,分别含有6、4、5、3、2株菌株,并用MEGA 7软件以最大似然法构建系统发育树(图 2)。
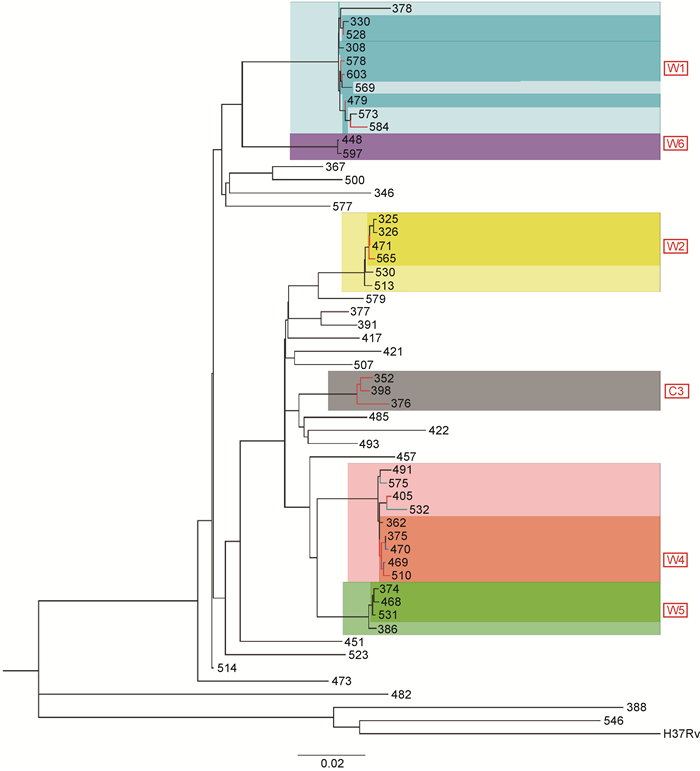
|
| 注:红色框内是用VNTR、WGS分型方法鉴定的簇,其中C簇是VNTR分型鉴定出的簇,W簇是WGS分型鉴定出的簇。 图 2 基于WGS分型方法构建进化树 Fig. 2 Phylogenetic tree based on WGS |
簇内菌株间SNP差异个数为4~21个,平均相差(12.89±3.62)个SNP。5个簇簇内SNP差异个数分别为7~21(平均13.9±3.6)、9~19(平均11.3±3.9)、4~21(平均13.2±4.7)、12、7个。进一步比较簇内关系最接近的菌株,其两两之间相差的SNP个数为4~12个,平均(9.8±2.4)个。5个簇簇内菌株与簇外其他菌株的SNP差异个数分别为(341.9±120.1)、(288.9±120.3)、(290.0±138.5)、(285.2±123.3)、(351.0±82.5)个(表 1)。
| WGS簇 | 菌株个数 | 簇内SNP差异最小值 | 簇内SNP差异最大值 | 簇内最近菌株间SNP差异个数 | 簇内SNP个数差异平均值 | 与簇外菌株SNP差异个数 | 组成 |
| W1 | 6 | 7 | 21 | 7~12 | 13.9±3.6 | 341.9±120.1 | 308、330、479、528、578、603 |
| W2 | 4 | 9 | 19 | 9 | 11.3±3.9 | 288.9±120.3 | 325、326、471、565 |
| W4 | 5 | 4 | 21 | 4~12 | 13.2±4.7 | 290.0±138.5 | 362、375、469、470、510 |
| W5 | 3 | 12 | 12 | 12 | 12 | 285.2±123.3 | 374、468、531 |
| W6 | 2 | 7 | 7 | 7 | 7 | 351.0±82.5 | 448、597 |
| 注:加粗的编号是WGS特有的菌株,未加粗的编号是两种分型方法共有的菌株。 | |||||||
VNTR将55株菌分成6个簇,WGS分成5个簇,其中C1与W1、C2与W2、C4-1~C4-3与W4有对应关系。
VNTR分型鉴定C1中的5株为一簇,WGS分型鉴定W1中的6株为一簇,两种方法均将Q479、Q528、Q578、Q603这4株分为同一簇。但Q584仅被VNTR鉴定为C1簇,WGS提示其不成簇,且与W1中其他菌株至少相差30个SNP;Q308、Q330仅被WGS鉴定为W1,VNTR提示其不成簇,且与C1中其他菌株分别相差2和3个VNTR位点。
VNTR分型鉴定C2中的2株为一簇,WGS分型鉴定W2中的4株为一簇。W2包含了VNTR分型鉴定的C2中的Q471、Q565两株,并在此基础上多鉴定出Q325、Q326与其成簇,Q325、Q326与C2簇分别相差1和3个VNTR位点。
VNTR分型鉴定Q405、Q510为一簇,即C4-1;鉴定Q469、Q470为一簇,即C4-2;鉴定Q532、Q575为一簇,即C4-3。其中C4-1与C4-2相差1个VNTR位点,C4-3与其他两个簇相差2个VNTR位点。WGS分型鉴定W4中的5株为一簇,W4中包含了C4-2的全部菌株(Q469、Q470)和C4-1中的Q510。此外,WGS还多鉴定出Q362、Q375与其成簇,这两株与其他C4菌株相差1个VNTR位点。VNTR鉴定为一簇,但WGS提示不成簇的菌株中,C4-3中的Q532与Q575相差45个SNP;C4-1中的Q405与Q510相差29个SNP。
VNTR分型鉴定C3中的3株为一簇(Q352、Q376、Q398),WGS提示其两两之间相差29、59、64个SNP,因此不成簇。此外,WGS新鉴定了2个簇,W5包含3株(Q374、Q468、Q531),其两两之间相差12个SNP、2~3个VNTR位点;W6包含2株(Q448、Q597),相差7个SNP、2个VNTR位点(表 1~2)。
| VNTR簇 | 菌株个数 | 簇内SNP差异最小值 | 簇内SNP差异最大值 | 簇内最近菌株间SNP差异个数 | 簇内SNP差异个数平均值 | 组成 |
| C1 | 5 | 10 | 40 | 10~30 | 22.4±12.9 | 479、528、578、584、603 |
| C2 | 2 | 9 | 9 | 9 | 9 | 471、565 |
| C3 | 3 | 29 | 64 | 29~59 | 50.6±18.9 | 352、376、398 |
| C4-1 | 2 | 29 | 29 | 29 | 29 | 405、510 |
| C4-2 | 2 | 12 | 12 | 12 | 12 | 469、470 |
| C4-3 | 2 | 45 | 45 | 45 | 45 | 532、575 |
| 注:加粗的编号是VNTR分型特有的菌株,未加粗的编号是两种分型方法共有的菌株。 | ||||||
共有8株同时被两种方法鉴定为成簇,8株仅VNTR鉴定其成簇,12株仅WGS鉴定其成簇,其余27株两种方法均提示不成簇。VNTR与WGS分型的一致性为63.6%(35/55)。与WGS相比,VNTR分型的特异度为77.1%,灵敏度为40.0%,阳性预测值为50.0%,阴性预测值为69.2%(表 3)。
| WGS | ||||
| 成簇 | 非成簇 | 总计 | ||
| VNTR | 成簇 | 8 | 8 | 16 |
| 非成簇 | 12 | 27 | 39 | |
| 总计 | 20 | 35 | 55 | |
VNTR分型与WGS分型相比,有20株成簇判断结果不一致,其中8株仅VNTR鉴定其成簇,12株仅WGS鉴定其成簇。8株仅VNTR鉴定成簇的菌株中,错误地将C3中的3个菌株分为一簇(Q352、Q376、Q398),WGS提示Q398、Q352分别与Q376相差59、64个SNP。这一现象已被多项研究报道,即VNTR分型一致的簇内SNP差异很大[6, 13],因此公认在鉴定传播时WGS比VNTR分辨率高。
12株仅WGS鉴定成簇的菌株中,W1中的Q308和Q330、W2中的Q326与C1簇、C2簇的其他菌株相差1或2个VNTR位点。WGS新鉴定了2个簇,共5个菌株(Q374、Q468、Q531、Q448、Q597),其簇内菌株间相差2个VNTR位点。这可能与VNTR多变位点的进化有关,同时也提示不能因为菌株间仅相差1~2个VNTR位点就排除近期传播[14]。现有研究多是在VNTR分型一致的菌株中进行WGS分型,很少在VNTR不一致的菌株中直接使用WGS分型并评估其效能,因此没有全面估计VNTR分型的灵敏度。
以往研究通过将WGS数据与流行病学调查结果结合,发现有流行病学联系的MDR菌株之间最多相差12个SNP,因此定义菌株间差异小于12个SNP为近期感染[7, 10, 15]。但是,本研究中纳入的均为XDR菌株,超过一半的菌株同时对异烟肼、利福平、乙胺丁醇、链霉素、二线注射类药物、氟喹诺酮类药物、丙硫异烟胺、对氨基水杨酸8类药物耐药,且对乙硫异烟胺和对氨基水杨酸这两种药物来说,目前已知的耐药基因仅能解释不到60%的表型耐药[16-18],其具体耐药机制及是否有补偿突变并不明确,导致在药物压力下筛选出的耐药相关基因及其补偿突变无法被过滤,可能因此造成菌株间SNP差异个数较多,即遗传距离较远。采用一个恒定的差异SNP阈值是适用于所有情况,还是仅限于某些特定情境,还有待进一步研究[14]。本研究尝试将55株菌株进行两两比对,构建SNP差异个数的矩阵,并绘制散点图(图 3);但是由于缺乏流行病学数据,无法通过此矩阵定义判断近期传播的SNP阈值。
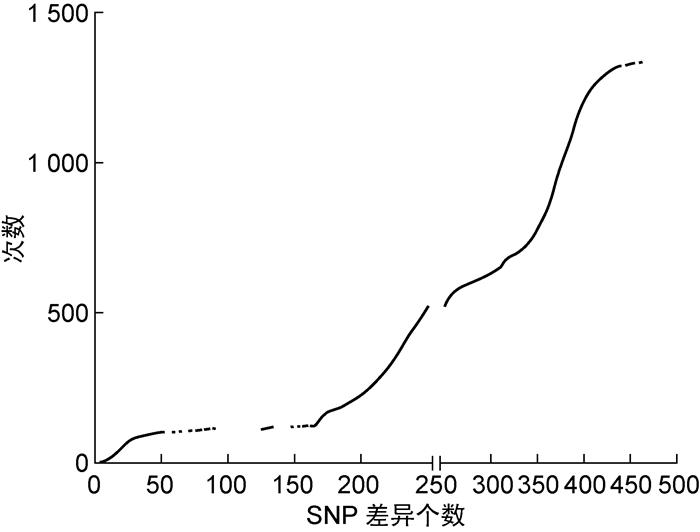
|
| 图 3 所有菌株间两两比较SNP差异个数 Fig. 3 Pairwise SNP differences among 55 strains |
由于结核分枝杆菌的遗传学特性,其可随机发生自发突变,且回复突变极少见。假设A同时将其菌株传给B、C两人,A菌在B、C两人体内分别进化,根据现有研究,则B与A、C与A的遗传距离小于12个SNP;但若直接比较B、C两菌,其差距最大可能达24个SNP。由于本研究中纳入的是单中心取样的菌株,并非疾病预防控制中心所获得的某地区的全部菌株,很有可能存在上述A病例丢失的可能性,造成具有近期传播关系的菌株间的遗传距离较大。因此,本研究中以菌株间差异小于12个SNP为近期感染这一标准,可能过于严苛。若定义菌株间差异小于24个SNP为近期感染,则可将55株菌株中的28株(50.91%)分入5簇。放宽标准后,与W1很接近的菌株Q569、Q573,分别与W1中的菌株最小相差16、23个SNP;与W2很接近的菌株Q513、Q530,分别与W2中的菌株最小相差17个SNP;与W4很接近的菌株Q491、Q575、Q405,分别与W4中的菌株最小相差23、15、20个SNP,且菌株Q405与C4-1中的Q510有一致的VNTR分型。
本研究存在以下不足。2003—2009年诊断为XDR的患者痰标本培养物于-80 ℃冰箱中保存至今,时间久远,仅有55株复苏成功。本研究纳入的是单中心收集的菌株,并非疾病预防控制中心所获得的某地区的全部菌株,且没有流行病学调查结果,因此缺失较多病例,即缺失传播的中间环节,无法构建传播链。成簇菌株采样时间跨度较大,最长时间间隔为42个月,可能造成簇内菌株遗传距离增加,这一现象在以往大型研究中也有提及[8]。
本研究采用基于SNP的分型方法评价VNTR分型在XDR-TB传播研究中的应用,发现VNTR分型具有较高的特异度,但仅用此方法可能会错误估计XDR的传播性。与VNTR分型相比,WGS可更全面地鉴定出传播簇。现有研究认为MDR-TB中菌株间相差不超过12个SNP即有近期传播关系,这一临界值是否适用于XDR-TB有待进一步研究。
| [1] |
Kendall EA, Fofana MO, Dowdy DW. Burden of transmitted multidrug resistance in epidemics of tuberculosis: a transmission modelling analysis[J]. Lancet Respir Med, 2015, 3(12): 963-972.
[DOI]
|
| [2] |
Wang F, Shao L, Fan X, Shen Y, Diao N, Jin J, Sun F, Wu J, Chen J, Weng X, Cheng X, Zhang Y, Zhang W. Evolution and transmission patterns of extensively drug-resistant tuberculosis in China[J]. Antimicrob Agents Chemother, 2015, 59(2): 818-825.
[DOI]
|
| [3] |
罗丹, 蓝兰, 赵锦明, 张影坤, 蓝如束. 结核分枝杆菌可变数目串联重复序列基因型成簇特征分析[J]. 中华传染病杂志, 2016, 34(8): 490-493. [DOI]
|
| [4] |
刘梅, 罗涛, 杨崇广, 柳清云, 高谦. 不同可变数目串联重复序列组合鉴定结核分枝杆菌临床菌株分型的效果[J]. 中华结核和呼吸杂志, 2015, 38(10): 746-750. [DOI]
|
| [5] |
Niemann S, Köser CU, Gagneux S, Plinke C, Homolka S, Bignell H, Carter RJ, Cheetham RK, Cox A, Gormley NA, Kokko-Gonzales P, Murray LJ, Rigatti R, Smith VP, Arends FP, Cox HS, Smith G, Archer JA. Genomic diversity among drug sensitive and multidrug resistant isolates of Mycobacterium tuberculosis with identical DNA fingerprints[J]. PLoS One, 2009, 4(10): e7407.
[DOI]
|
| [6] |
Gardy JL, Johnston JC, Ho Sui SJ, Cook VJ, Shah L, Brodkin E, Rempel S, Moore R, Zhao Y, Holt R, Varhol R, Birol I, Lem M, Sharma MK, Elwood K, Jones SJ, Brinkman FS, Brunham RC, Tang P. Whole-genome sequencing and social-network analysis of a tuberculosis outbreak[J]. N Engl J Med, 2011, 364(8): 730-739.
[DOI]
|
| [7] |
Walker TM, Ip CL, Harrell RH, Evans JT, Kapatai G, Dedicoat MJ, Eyre DW, Wilson DJ, Hawkey PM, Crook DW, Parkhill J, Harris D, Walker AS, Bowden R, Monk P, Smith EG, Peto TE. Whole-genome sequencing to delineate Mycobacterium tuberculosis outbreaks: a retrospective observational study[J]. Lancet Infect Dis, 2013, 13(2): 137-146.
[DOI]
|
| [8] |
Roetzer A, Diel R, Kohl TA, Ruckert C, Nubel U, Blom J, Wirth T, Jaenicke S, Schuback S, Rüsch-Gerdes S, Supply P, Kalinowski J, Niemann S. Whole genome sequencing versus traditional genotyping for investigation of a Mycobacterium tuberculosis outbreak: a longitudinal molecular epidemiological study[J]. PLoS Med, 2013, 10(2): e1001387.
[DOI]
|
| [9] |
Guerra-Assunção JA, Crampin AC, Houben RM, Mzembe T, Mallard K, Coll F, Khan P, Banda L, Chiwaya A, Pereira RP, McNerney R, Fine PE, Parkhill J, Clark TG, Glynn JR. Large-scale whole genome sequencing of M. tuberculosis provides insights into transmission in a high prevalence area[J]. Elife, 2015.
[DOI]
|
| [10] |
Yang C, Luo T, Shen X, Wu J, Gan M, Xu P, Wu Z, Lin S, Tian J, Liu Q, Yuan Z, Mei J, DeRiemer K, Gao Q. Transmission of multidrug-resistant Mycobacterium tuberculosis in Shanghai, China: a retrospective observational study using whole-genome sequencing and epidemiological investigation[J]. Lancet Infect Dis, 2017, 17(3): 275-284.
[DOI]
|
| [11] |
Luo T, Yang C, Pang Y, Zhao Y, Mei J, Gao Q. Development of a hierarchical variable-number tandem repeat typing scheme for Mycobacterium tuberculosis in China[J]. PLoS One, 2014, 9(2): e89726.
[DOI]
|
| [12] |
Coll F, McNerney R, Guerra-Assunção JA, Glynn JR, Perdigão J, Viveiros M, Portugal I, Pain A, Martin N, Clark TG. A robust SNP barcode for typing Mycobacterium tuberculosis complex strains[J]. Nat Commun, 2014, 5: 4812.
[DOI]
|
| [13] |
Luo T, Yang C, Peng Y, Lu L, Sun G, Wu J, Jin X, Hong J, Li F, Mei J, DeRiemer K, Gao Q. Whole-genome sequencing to detect recent transmission of Mycobacterium tuberculosis in settings with a high burden of tuberculosis[J]. Tuberculosis (Edinb), 2014, 94(4): 434-440.
[DOI]
|
| [14] |
Pérez-Lago L, Comas I, Navarro Y, González-Candelas F, Herranz M, Bouza E, García-de-Viedma D. Whole genome sequencing analysis of intrapatient microevolution in Mycobacterium tuberculosis: potential impact on the inference of tuberculosis transmission[J]. J Infect Dis, 2014, 209(1): 98-108.
[DOI]
|
| [15] |
Walker TM, Lalor MK, Broda A, Ortega LS, Morgan M, Parker L, Churchill S, Bennett K, Golubchik T, Giess AP, Del Ojo Elias C, Jeffery KJ, Bowler ICJW, Laurenson IF, Barrett A, Drobniewski F, McCarthy ND, Anderson LF, Abubakar I, Thomas HL, Monk P, Smith EG, Walker AS, Crook DW, Peto TEA, Conlon CP. Assessment of Mycobacterium tuberculosis transmission in Oxfordshire, UK, 2007-12, with whole pathogen genome sequences: an observational study[J]. Lancet Respir Med, 2014, 2(4): 285-292.
[DOI]
|
| [16] |
Morlock GP, Metchock B, Sikes D, Crawford JT, Cooksey RC. ethA, inhA, and katG loci of ethionamide-resistant clinical Mycobacterium tuberculosis isolates[J]. Antimicrob Agents Chemother, 2003, 47(12): 3799-3805.
[DOI]
|
| [17] |
Niehaus AJ, Mlisana K, Gandhi NR, Mathema B, Brust JC. High prevalence of inhA promoter mutations among patients with drug-resistant tuberculosis in KwaZulu-Natal, South Africa[J]. PLoS One, 2015, 10(9): e0135003.
[DOI]
|
| [18] |
Zhang X, Liu L, Zhang Y, Dai G, Huang H, Jin Q. Genetic determinants involved in p-aminosalicylic acid resistance in clinical isolates from tuberculosis patients in northern China from 2006 to 2012[J]. Antimicrob Agents Chemother, 2015, 59(2): 1320-1324.
[DOI]
|
 2018, Vol. 13
2018, Vol. 13


