皮肤作为人体的第一道物理屏障,对防御外来病原菌的攻击有重要作用[1]。皮肤上有大量微生物,包括细菌、真菌、病毒和螨虫等[2]。根据物理结构及化学环境等差异,皮肤主要分为3类:背部及额头等油脂分泌较多的皮脂溢出部位,前臂等干燥部位,以及鼻孔及指缝等湿润部位[3]。这3类皮肤上的微生物群落结构大不相同。已有研究表明,皮肤脂溢部位的微生物多样性显著低于干燥和湿润部位[4]。
婴儿皮肤表面的微生物对其免疫系统的建立有一定影响。研究显示,新生儿出生后10 min内已能检测到皮肤表面的微生物,表明出生后皮肤表面迅速被各种微生物占据[5-6]。因此,研究婴儿皮肤表面的微生物对维护人类健康和疾病防治非常重要[7-8]。新生儿皮肤的理化性质与成人有显著差异。相比成人,新生儿皮肤更柔软,水分及pH更高,皮脂含量更低[9-12]。同时,新生儿皮肤表面的微生态与成人也有差异,在出生不到1年的新生儿皮肤表面葡萄球菌属(Staphylococcus)和链球菌属(Streptococcus)的丰度高达60%[13]。
目前,皮肤微生物相关研究主要针对成人,母亲与婴儿之间皮肤细菌群落相关性的文献较少。有学者对出生1 d以内的新生儿及其母亲相应部位的微生物进行取样,证明新生儿皮肤表面的微生物与分娩方式有关[5, 14]。母亲是婴儿接触最为频繁的人,两者皮肤微生物相互传播的机会较多[8, 14-15]。婴儿出生后免疫系统需要一定的时间才能逐步健全,在此期间自身微生物与免疫系统尚未达到平衡状态,成人机体上的细菌在婴儿皮肤微生态的塑造中发挥着何种作用尚不可知[16]。另一方面,自然分娩出生的婴儿首先接触的有菌环境是母亲的产道,并从中获取部分细菌,而剖宫产出生的婴儿则首先接触母亲的皮肤和外部环境,两类婴儿之间细菌群落结构差异有统计学意义[5, 15]。有研究显示,剖宫产可能与婴儿乳糜泻、哮喘等疾病有关[17-20],但尚不清楚生产方式不同导致的微生物差异如何给婴儿带来潜在疾病风险及这种差异会在何时消失。
本研究选取8名2~4个月的婴儿及其母亲作为受试者,采用焦磷酸测序分析皮肤细菌多样性,意在阐明年龄为2~4个月的婴儿皮肤表面细菌群落的特征、母婴皮肤细菌多样性的相关性、自然分娩出生的婴儿与剖宫产出生的婴儿之间皮肤微生物群落的差异。
1 材料与方法 1.1 材料 1.1.1 试剂和仪器仪器包括恒科DK 80水浴锅、TaKaRa聚合酶链反应(polymerase chain reaction,PCR)仪、Eppendorf台式高速离心机5415D和Nanodrop 3300。Mobio PowerSoil DNA提取试剂盒、Mobio UltraClean PCR纯化试剂盒购自深圳市青珊瑚科技有限公司,Ex Taq酶混合液和无酶无菌去离子水购自天根科技(北京)有限公司,小牛血清蛋白购自生工生物工程(上海)股份有限公司,PicoGreen dsDNA Reagent(Invitrogen)购自赛默飞世尔科技(中国)有限公司,AxyPrep DNA Gel Extraction Kit(Axygen)购自上海因帕仪器有限公司。
1.1.2 引物采用细菌通用引物27FYM(5 ′ -AGAGTTTGATYMTGGCTCAG-3 ′)和536RK(5 ′ -GTATTACCGCGGCKGCTGG-3 ′)扩增细菌16S rRNA基因V1~V3区域。引物AdaA-27FYM(5 ′ -CCATCTCATCCCTGCGTGTCTCCGACGA-CTNNNNNNNNTCAGAGTTTGATYMTGGCT-CAG-3 ′)包含454测序接头A、8-nt[用于区分不同样品的标签序列(NNNNNNNN)]、2-nt连接碱基(TC)和27FYM。引物AdaB-536RK(5 ′ -CCTA-TCCCCTGTGTGCCTTGGCAGTCGACTCAGT-ATTACCGCGGCKGCTGG-3 ′)包含454测序接头B、2-nt连接碱基(CA)和536RK[21]。
1.1.3 在线工具和数据库使用细菌16S rRNA基因数据库Silva(http://www.arb-silva.de/)、UniFrac距离矩阵在线构建工具Fast UniFrac(http://bmf2.colorado.edu/fastunifrac/index.psp)。
1.2 方法 1.2.1 采样本研究通过了复旦大学生命科学学院伦理委员会的审核。有8对年龄相近的母婴参与本研究,其中4名婴儿为剖宫产,4名婴儿为自然分娩(表 1)。受试者均由医师在取样前检查并确认没有皮肤病。所有母亲均被详细告知本研究的目的和采样后可能出现的不适反应,并签署了书面知情同意书。所有受试者在采样前7 d内未使用抗生素或甾醇类药物,采样位点12 h内未有洗漱。母亲皮肤表面的采样部位共有7处,采样顺序为手背(hand back,Hb)、手指指缝(interdigital space,Is;在中指与无名指之间进行样本采集)、前臂掌侧(volar forearm,Vf)、肘窝(antecubital fossa,Af)、鼻孔(nares,Na)、额头(glabella,Gb)和背部(back,Ba)。Na部位的样品采集可能对婴儿造成伤害,因此婴儿采集了除Na之外其余6个部位的样品,先后顺序与母亲相同。采样使用0.15 mol/L NaCl和0.1%吐温20溶液浸湿的聚酯纤维头拭子,采样面积为2 cm×2 cm(Is部位为1 cm×1 cm)。采样过程中,拭子在规定区域内至少完成50次擦拭,持续时间约30 s。然后用灭菌镊子将拭子头取出,小心放入Mobio PowerSoil DNA提取试剂盒内含有石英砂的PowerBead Tube中。将一个湿润的、未经擦拭的拭子头放入PowerBead Tube中,作为空白对照。每个受试者接受隔天3次重复采样,最终获取母婴样品共计312份。采样时室温维持在24 ℃左右。
| Mothers | Infants | |||||
| No. | Age (year) | No. | Way of parturition | Age (month) | Sex | |
| M01 | 31 | I01 | Caesarean | 3 | Male | |
| M02 | 30 | I02 | Caesarean | 4 | Female | |
| M03 | 28 | I03 | Caesarean | 4 | Female | |
| M04 | 26 | I04 | Caesarean | 2 | Female | |
| M05 | 29 | I05 | Vaginally born | 2 | Male | |
| M06 | 31 | I06 | Vaginally born | 3 | Female | |
| M07 | 33 | I07 | Vaginally born | 3 | Male | |
| M08 | 32 | I08 | Vaginally born | 2 | Female | |
所有样品均在采样后12 h内完成DNA提取。为提高DNA提取效率,优化了DNA提取步骤:将加入60 μL C1溶液的PowerBead Tube密封并于65 ℃恒温水浴10 min,随后以最大速度在涡旋振荡仪上水平振动5 min,其余步骤按试剂盒提供的指导说明逐步进行,最终将DNA重悬于50 μL洗脱液中,-20 ℃保存。
1.2.3 PCR使用两轮PCR扩增细菌16S rRNA基因序列。第1轮反应使用细菌通用引物27FYM和536RK。25 μL反应体系包括:正反向引物(10 μmol/L)各0.25 μL、DNA 6 μL、小牛血清蛋白溶液(20 mg/mL)1 μL、Ex Taq酶混合液12.5 μL。扩增反应参数:94 ℃预变性5 min;94 ℃ 45 s、53 ℃ 30 s、72 ℃ 90 s,20次循环;72 ℃最终延伸10 min。第2轮反应使用引物AdaA-27FYM和AdaB-536RK,将焦磷酸测序接头连接到扩增产物的两端。100 μL反应体系包括:正反向引物(10 μmol/L)各1 μL、第1轮PCR产物10 μL、小牛血清蛋白溶液(20 mg/mL)4 μL、Ex Taq酶混合液50 μL。反应参数中循环数由20下调至10,其余参数与第1轮相同。最终获得含有测序接头的长约500 bp的片段。
1.2.4 PCR产物纯化和测序采用Mobio UltraClean PCR试剂盒纯化第2轮PCR扩增产物,随后用50 μL洗脱液将纯化后的产物重悬。用PicoGreen dsDNA Reagent染色并于Nanodrop 3300测定产物浓度,根据浓度将所有纯化产物混合,用AxyPrep DNA Gel Extraction Kit进行切胶回收,完成进一步纯化。最后于Roche 454 GS-FLX平台进行焦磷酸测序。
1.2.5 微生物分类和多样性分析测序产生196 790条16S rRNA基因序列。采用Mothur软件对测序数据进行初步处理[22],质控(Q score>25)分库后保留138 375条;在Bioedit软件内删除前端引物序列后,根据97%相似度划分可操作分类单元(operational taxonomy unit,OTU),产生2 239个OTU;使用UCHIME比对Silva gold database(http://www.mothur.org/wiki/Silva_reference_files),识别并去除数据中的嵌合体;移除序列数少于5条的OTU以避免焦磷酸测序错误产生的影响,最终获得1 078个OTU;比对Silva数据库,获取各OTU代表序列的分类信息。若某细菌在超过50%的样品中被发现,则认为是皮肤常驻细菌(general bacterium),若同时丰度>1%,则被认为是皮肤主要细菌(core bacterium)。对皮肤上较为常见且在本研究中丰度较高的几个细菌种属进行Local Blast,比对数据库为各种属典型菌株的16S rRNA基因序列[23-27]。对数据进行标准化(深度为74)后计算α多样性指数(包括Sobs、ACE、Shannon、Chao I、Jackknife指数)和β多样性(使用Fast UniFrac计算加权和非加权UniFrac距离矩阵)。
1.2.6 聚类分析和统计学检验使用R软件中ape()进行主坐标分析(principal coordinates analysis,PCoA),ggplot()进行绘图,pairwise.t.test()进行双尾T检验,并使用Bonferroni方法进行多重检验矫正。距离矩阵热图绘制采用MeV(MultiExperiment Viewer)软件,聚类分析采用HCL(hierarchical clustering)方法。
2 结果 2.1 母亲和婴儿皮肤细菌多样性分析Shannon等α多样性指数结果如表 2所示。母亲不同部位细菌群落的5个多样性指数变化基本相同:Gb、Ba和Na部位细菌多样性低于Af、Is、Vf和Hb部位。在婴儿样本中,Is和Hb部位多样性相对较低,Ba部位多样性较高,且不同婴儿同一部位的多样性指数变化较大。婴儿群体在Ba部位的多样性指数显著高于母亲群体(P < 0.001),母亲群体手部皮肤(Is和Hb)较婴儿群体拥有更高的细菌多样性(均P < 0.05)。
| Gb | Ba** | Na | |||||
| Mothers | Infants | Mothers | Infants | Mothers | |||
| Sobs | 19.8±4.7 | 26.5±3.0 | 22.8±3.2 | 36.6±5.0 | 19.1±2.5 | ||
| Chao I | 44.0±11.4 | 49.1±7.8 | 50.1±9.9 | 91.2±17.9 | 41.2±10.9 | ||
| ACE | 88.1±41.2 | 81.3±24.6 | 96.4±15.3 | 147.5±40.6 | 63.3±15.7 | ||
| Jackknife | 46.8±11.1 | 50.2±8.4 | 56.6±12.4 | 100.0±23.0 | 42.4±10.7 | ||
| Shannon | 1.80±0.41 | 2.67±0.31 | 2.17±0.37 | 3.22±0.25 | 2.31±0.12 | ||
| Simpson | 0.348±0.117 | 0.122±0.049 | 0.245±0.106 | 0.048±0.015 | 0.132±0.013 | ||
| Af | Is** | ||||||
| Mothers | Infants | Mothers | Infants | ||||
| Sobs | 33.8±2.8 | 32.8±2.7 | 36.3±3.7 | 20.1±0.9 | |||
| Chao I | 73.4±14.9 | 75.0±16.2 | 80.6±19.5 | 40.7±8.0 | |||
| ACE | 102.1±24.6 | 100.3±18.0 | 129.3±38.6 | 60.5±14.9 | |||
| Jackknife | 79.1±18.0 | 80.9±18.8 | 89.4±27.7 | 40.7±7.5 | |||
| Shannon | 3.15±0.13 | 3.07±0.11 | 3.19±0.22 | 2.37±0.17 | |||
| Simpson | 0.053±0.010 | 0.061±0.011 | 0.054±0.020 | 0.134±0.026 | |||
| Vf | Hb* | ||||||
| Mothers | Infants | Mothers | Infants | ||||
| Sobs | 37.8±2.5 | 33.6±1.9 | 36.8±2.3 | 29.7±5.3 | |||
| Chao I | 81.1±10.3 | 74.5±11.8 | 80.2±14.2 | 59.6±14.1 | |||
| ACE | 111.1±14.1 | 99.0±16.4 | 124.8±29.6 | 98.4±20.7 | |||
| Jackknife | 87.6±14.6 | 76.6±11.4 | 83.4±17.8 | 61.2±15.5 | |||
| Shannon | 3.23±0.16 | 2.99±0.12 | 3.23±0.12 | 2.82±0.27 | |||
| Simpson | 0.050±0.017 | 0.077±0.018 | 0.051±0.011 | 0.091±0.019 | |||
| *P < 0.05, * *P < 0.001. | |||||||
将加权UniFrac数据按M_same_Site(母亲相同部位所有样品之间的差异)、M_different_Sites(母亲某部位所有样品与其他部位样品之间的差异)、M_I(母亲与婴儿相同部位所有样品之间的差异)、I_same_Site(婴儿相同部位所有样品之间的差异)和I_different_Sites(婴儿某部位所有样品与其他部位样品之间的差异)分组,并在每两组之间进行T检验(图 1)。结果显示,M_same_Site显著低于M_different_Sites(P < 0.05),而I_same_Site显著高于M_same_Site(P < 0.05)。I_same_Site与I_different_Sites之间差异较小,但仍有统计学差异(P < 0.05)。上述结果表明,婴儿皮肤细菌群落在个体间的变异程度比母亲高。从M_I组可看出,母亲与婴儿之间同一部位细菌群落结构差异较大,尤其是Gb、Is和Hb;而在Af部位,母亲与婴儿的菌群结构相似。此外,将出生方式、婴儿性别与细菌群落结构的关系也纳入检验,但没有观察到显著差异。
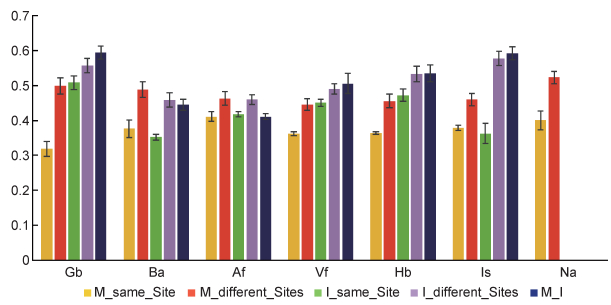
|
| M_same_Site: weighted UniFrac distance between the same site of mothers; M_different_Sites: weighted UniFrac distance between different sites of mothers; I_same_Site: weighted UniFrac distance between the same site of infants; I_different_Sites: weighted UniFrac distance between different sites of infants; M_I: weighted UniFrac distance between the same site from mothers and infants. 图 1 样本之间加权UniFrac距离比较 Fig. 1 Weighted UniFrac distance of samples |
观察非加权和加权UniFrac PCoA(图 2)及距离矩阵热图(图 3)发现,非加权结果中,母亲Na部位与其他部位差异较大,但与婴儿Is部位相近(图 2A、图 3A);加权结果中,母婴皮肤部位之间的细菌群落各自聚类(图 2B、图 3B),婴儿的Is、Vf、Hb和Gb部位与所有母亲样品分开。婴儿Gb与Hb部位细菌群落结构相似,Ba与Af部位相似。母亲Is、Af部位分别与婴儿Ba、Af部位细菌群落结构相似。
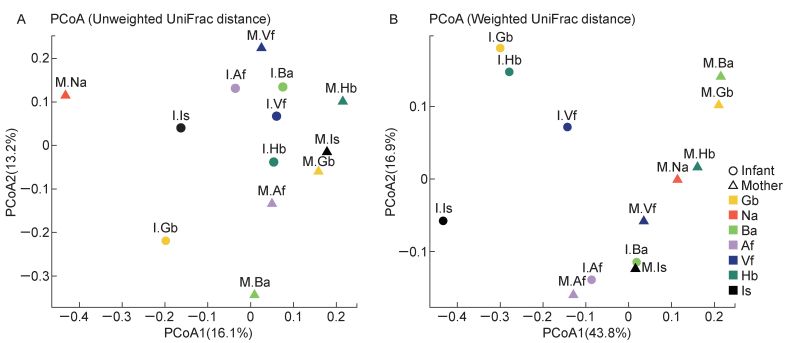
|
| Samples were grouped according to the identity and skin site. M means mothers, I means infants. For example, M.Na means Na samples from mothers. 图 2 母婴各皮肤部位细菌群落非加权(A)和加权(B)PCoA分析 Fig. 2 Unweighted (A) and weighted (B) PCoA analysis of bacterial communities on skins of mothers and infants |
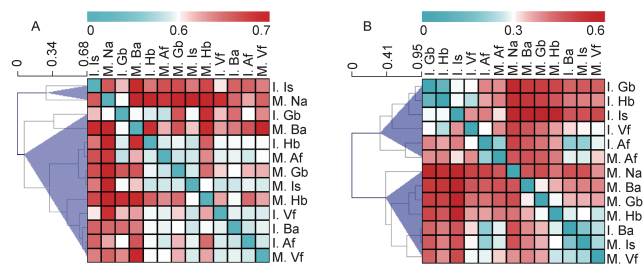
|
| Hierarchical Clustering method was used in sample clusters. 图 3 母婴各皮肤部位之间细菌群落非加权(A)和加权(B)距离矩阵热图 Fig. 3 Unweighted (A) and weighted (B) UniFrac distance heat map of bacterial communities between skin sites of mothers and infants |
母婴皮肤微生物主要有4个门:拟杆菌门(Bacteroidetes;母婴皮肤表面平均含量分别为2.3%和2.0%)、放线菌门(Actinobacteria;分别为29.0%和10.5%)、厚壁菌门(Firmicutes;分别为30.4%和64.4%)及变形菌门(Proteobacteria;分别为28.0%和19.1%)。后三者在母亲与婴儿样本中含量差异显著(P < 0.001),其余门的比例均 < 1%。
母亲与婴儿皮肤的主要细菌相似(图 4、表 3),包括棒状杆菌属(Corynebacterium)、丙酸杆菌属(Propionibacterium)、葡萄球菌属和链球菌属。母亲与婴儿相同的皮肤常驻细菌包括不动杆菌属(Acinetobacter)、厌氧球菌属(Anaerococcus)、脱硫微菌属(Desulfomicrobium)、水栖菌属(Enhydrobacter)、盐单胞菌属(Halomonas)、嗜糖假单胞菌属(Pelomonas)、假单胞菌属(Pseudomonas)和鞘氨醇单胞菌属(Sphingomonas)。短波单胞菌属(Brevundimonas)和副球菌属(Paracoccus)是母亲独有的常驻细菌;孪生球菌属(Gemella)、普雷沃菌属(Prevotella)、罗思菌属(Rothia)和韦荣球菌属(Veillonella)是婴儿独有的常驻细菌。该结果与PCoA和距离矩阵热图(母婴样本在非加权时聚类在一起,而在加权时呈现各自的聚类)结果可表明,婴儿与母亲皮肤表面细菌种类组成相似,但丰度存在差异。

|
| 图 4 母婴皮肤各部位主要细菌属(>0.5%)的分布状况 Fig. 4 Main bacterial genera (>0.5%) on skin sites of mothers and infants |
| Mothers | Infants | |
| Core bacteria | Propionibacterium, Staphylococcus, Corynebacterium, Streptococcus, Escherichia |
Streptococcus, Staphylococcus, Corynebacterium, Propionibacterium |
| General bacteria | Enhydrobacter, Acinetobacter, Pseudomonas, Pelomonas, Desulfomicrobium, Sphingomonas, Halomonas, Anaerococcus, Paracoccus, Brevundimonas |
Enhydrobacter, Escherichia, Veillonella, Acinetobacter, Pseudomonas, Gemella, Pelomonas, Desulfomicrobium, Anaerococcus, Halomonas, Sphingomonas, Rothia, Prevotella |
不动杆菌属在母亲Is和Hb部位比例较高,主要包含约氏不动杆菌(Acinetobacter johnsonii;35.5%)、琼氏不动杆菌(Acinetobacter junii;30.2%)和鲍曼不动杆菌(Acinetobacter baumannii;11.8%)。棒状杆菌属主要分布在Na和其他非脂溢部位,包括假生殖道棒状杆菌(Corynebacterium pseudogenitalium)、结核硬脂酸棒状杆菌(Corynebacterium tuberculostearicum)和拥挤棒杆菌(Corynebacterium accolens),三者占棒状杆菌属总量的57.8%;其他还包括Corynebacterium camporealensis(27.3%)和麦氏棒杆菌(Corynebacterium macginleyi;6.8%)。丙酸杆菌属在母亲的Gb、Ba和Na部位含量丰富,Local Blast结果显示仅存在一个种:痤疮丙酸杆菌(Propionibacterium acnes)。痤疮丙酸杆菌多分布在皮肤脂溢部位,在潮湿和干燥部位的丰度低于皮肤脂溢部位。
比较自然分娩出生的婴儿与剖宫产出生的婴儿(图 5),Gb部位链球菌属(自然分娩出生的婴儿和剖宫产出生的婴儿的含量65.2% vs. 48.5%)和葡萄球菌属(1.7% vs. 4.5%)含量差异显著;Ba部位脱硫微菌属(0.2% vs. 3.3%)、葡萄球菌属(3.9% vs. 13.2%)和不动杆菌属(2.3% vs. 0.4%)含量差异显著;Af部位假单胞菌属(1.8% vs. 0.4%)和葡萄球菌属(5.0% vs. 13.0%)含量差异显著(均P < 0.05);Is和Hb部位微生物组成无显著差异。葡萄球菌属在Is部位最多,其次是Af和Ba部位;剖宫产出生的婴儿Gb、Ba和Af部位葡萄球菌属的含量较自然分娩出生的婴儿高(P < 0.05)。Local Blast结果显示,葡萄球菌属包括溶血性葡萄球菌(Staphylococcus hominis;64.2%)、表皮葡萄球菌(Staphylococcus epidermis;28.3%)和金黄色葡萄球菌(Staphylococcus aureus;5.7%)。链球菌属在婴儿Ba部位含量较低(0~14.1%)。8名婴儿中仅有1名为非母乳喂养,其样本中链球菌平均含量为40.4%。Local Blast结果显示,链球菌属主要为假肺炎链球菌(Streptococcus pseudopneumoniae;72.1%)、唾液链球菌(Streptococcus salivarius;15.3%)和口腔链球菌(Streptococcus oralis;10.0%)。
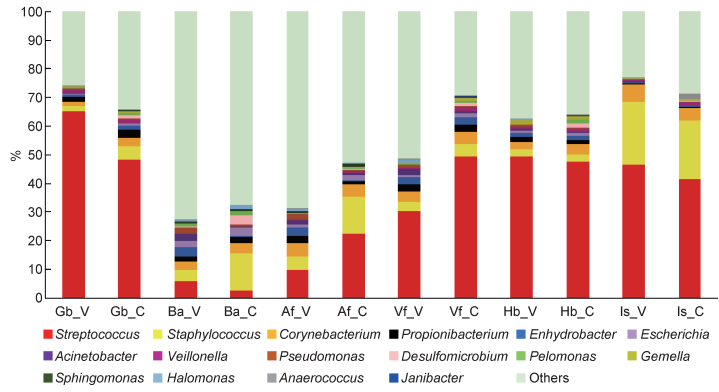
|
| V: vaginally born infants; C: caesarian-born infants. 图 5 自然分娩出生的婴儿与剖宫产出生的婴儿各部位主要细菌属(>0.5%)分布比较 Fig. 5 Main bacterial genera (>0.5%) on skin sites of vaginally born and caesarian-born infants |
皮肤是人体重要的防御器官,了解皮肤表面微生物有助于疾病预防和治疗。皮肤细菌对环境选择有一定的偏好,例如放线菌门(主要为丙酸杆菌)在母亲的皮脂溢出部位(Gb和Ba)较高,而在婴儿相应部位较低。除Na部位外,厚壁菌门(主要为链球菌和葡萄球菌)的丰度在其他采样点高于放线菌门。由此推断,放线菌门倾向于分布在皮脂溢出部位,而厚壁菌门更倾向于分布在非皮脂溢出部位[4]。出生4个月以内的婴儿皮脂分泌量少,角质层水分含量很高,有利于链球菌和葡萄球菌的生长[13]。成人Gb和Ba是皮脂溢出部位,而丙酸杆菌与皮肤皮脂分泌密切相关,因此丙酸杆菌在母亲Gb和Ba部位有很高的丰度[4]。
母亲的微生物群落已成为独立的生态位,个体间相同部位细菌群落结构差异较小,呈现相对稳定性。相反,婴儿个体间的细菌群落结构差异大,表现出相对高的变异性。儿童Hb和Is部位的微生物多样性指数低于母亲,链球菌和葡萄球菌的含量较高可能是原因之一。研究显示,人类皮肤细菌多样性指数在12岁之前呈上升趋势,成人的皮肤细菌多样性显著高于青少年和老年人[28-29],与本研究结果一致。母亲的细菌多样性高于婴儿,婴儿与自己母亲在细菌种类构成上又相似,表明母亲是影响婴儿皮肤微生物群落塑造的重要外环境之一。婴儿之间皮肤细菌群落结构更相近,表明皮肤微生物的丰度与皮肤的年龄、理化状态相关,而环境对皮肤微生物的影响主要为微生物种类[28, 30]。
不同分娩方式出生的婴儿皮肤表面微生物从出生开始呈现不同的结构。葡萄球菌在环境中常见,出生时剖宫产出生的婴儿中其含量比自然分娩出生的婴儿更高。原因可能是自然分娩出生的婴儿在出生时经过产道并获取部分微生物,且母亲产道菌群分泌的抗菌肽能帮助其抵抗环境微生物的定植[31]。而剖宫产出生的婴儿首先接触到的有菌环境是母亲的皮肤和空气,因此首先从外部环境中获取部分微生物。换言之,剖宫产出生的婴儿更易受到环境微生物入侵,这对婴儿皮肤免疫系统的建立具有重要意义[8]。
出生2~3个月以后,两种分娩方式出生的婴儿皮肤表面微生物呈现相似状态,群落结构差异不显著[13]。婴儿皮肤表面存在大量链球菌和葡萄球菌。链球菌是婴儿所有皮肤部位的优势菌,但在婴儿出生时并不占主导地位[13],出生数月后链球菌优势逐渐呈现。链球菌可帮助其他微生物形成生物膜并定植于牙齿表面[32-34]。研究显示,婴儿皮肤上的链球菌可能源自母亲的口腔和母乳[16, 35]。本研究中,婴儿皮肤表面的链球菌主要为假肺炎链球菌、唾液链球菌和口腔链球菌,后两者通常在口腔中被检测到[36-37],此结果也支持婴儿皮肤上链球菌可能源自母亲口腔。此外,4种婴儿独有的皮肤常驻细菌(孪生球菌属、普雷沃菌属、罗思菌属和韦荣球菌属)通常可在成年人的口腔中检测到[38]。另有小鼠实验研究证明,母亲的口腔微生物是胎儿宫内感染的潜在来源[39]。这些结果表明,母亲的口腔细菌对婴儿早期皮肤细菌群落的塑造可能发挥一定的作用[31-33]。
综上所述,婴儿与母亲的皮肤表面细菌群落结构存在差异,又有联系。出生后2~4个月,婴儿皮肤常驻细菌不仅受不同分娩方式的影响,还受母亲皮肤、口腔细菌的影响。这些结果对了解婴儿皮肤微生态构造和皮肤病防治有重要意义。
| [1] |
Williams ML, Hanley K, Elias PM, Feingold KR. Ontogeny of the epidermal permeability barrier[J]. J Investig Dermatol Symp Proc, 1998, 3(2): 75-79.
[DOI]
|
| [2] |
Grice EA, Kong HH, Renaud G, Young AC; NISC Comparative Sequencing Program; Bouffard GG, Blakesley RW, Wolfsberg TG, Turner ML, Segre JA. A diversity profile of the human skin microbiota[J]. Genome Res, 2008, 18(7): 1043-1050.
[DOI]
|
| [3] |
Kong HH. Skin microbiome: genomics-based insights into the diversity and role of skin microbes[J]. Trends Mol Med, 2011, 17(6): 320-328.
[DOI]
|
| [4] |
Grice EA, Kong HH, Conlan S, Deming CB, Davis J, Young AC; NISC Comparative Sequencing Program; Bouffard GG, Blakesley RW, Murray PR, Green ED, Turner ML, Segre JA. Topographical and temporal diversity of the human skin microbiome[J]. Science, 2009, 324(5931): 1190-1192.
[DOI]
|
| [5] |
Dominguez-Bello MG, Costello EK, Contreras M, Magris M, Hidalgo G, Fierer N, Knight R. Delivery mode shapes the acquisition and structure of the initial microbiota across multiple body habitats in newborns[J]. Proc Natl Acad Sci USA, 2010, 107(26): 11971-11975.
[DOI]
|
| [6] |
Costello EK, Carlisle EM, Bik EM, Morowitz MJ, Relman DA. Microbiome assembly across multiple body sites in low-birthweight infants[J]. MBio, 2013, 4(6): e00782-13.
[URI]
|
| [7] |
Naik S, Bouladoux N, Linehan JL, Han SJ, Harrison OJ, Wilhelm C, Conlan S, Himmelfarb S, Byrd AL, Deming C, Quinones M, Brenchley JM, Kong HH, Tussiwand R, Murphy KM, Merad M, Segre JA, Belkaid Y. Commensal-dendritic-cell interaction specifies a unique protective skin immune signature[J]. Nature, 2015, 520(7545): 104-108.
[DOI]
|
| [8] |
Gensollen T, Iyer SS, Kasper DL, Blumberg RS. How colonization by microbiota in early life shapes the immune system[J]. Science, 2016, 352(6285): 539-544.
[DOI]
|
| [9] |
Kong F, Galzote C, Duan Y. Change in skin properties over the first 10 years of life: a cross-sectional study[J]. Arch Dermatol Res, 2017, 309(8): 653-658.
[DOI]
|
| [10] |
Fluhr J, de Belilovsky C, Lachmann N, Msika P. What is different in skin physiology in neonates and young children of different age groups compared to adults? A randomized in vivo study[J/OL]. J Am Acad Dermatol, 2011, 64(2): AB129. https://www.sciencedirect.com/science/article/pii/S0190962210016129.
|
| [11] |
Luebberding S, Krueger N, Kerscher M. Skin physiology in men and women: in vivo evaluation of 300 people including TEWL, SC hydration, sebum content and skin surface pH[J]. Int J Cosmet Sci, 2013, 35(5): 477-483.
[DOI]
|
| [12] |
Oranges T, Dini V, Romanelli M. Skin physiology of the neonate and infant: clinical implications[J]. Adv Wound Care (New Rochelle), 2015, 4(10): 587-595.
[DOI]
|
| [13] |
Capone KA, Dowd SE, Stamatas GN, Nikolovski J. Diversity of the human skin microbiome early in life[J]. J Invest Dermatol, 2011, 131(10): 2026-2032.
[DOI]
|
| [14] |
Si J, Lee S, Park JM, Sung J, Ko G. Genetic associations and shared environmental effects on the skin microbiome of Korean twins[J]. BMC Genomics, 2015.
[DOI]
|
| [15] |
Funkhouser LJ, Bordenstein SR. Mom knows best: the universality of maternal microbial transmission[J]. PLoS Biol, 2013, 11(8): e1001631.
[DOI]
|
| [16] |
Mueller NT, Bakacs E, Combellick J, Grigoryan Z, Dominguez-Bello MG. The infant microbiome development: mom matters[J]. Trends Mol Med, 2015, 21(2): 109-117.
[DOI]
|
| [17] |
Roduit C, Scholtens S, de Jongste JC, Wijga AH, Gerritsen J, Postma DS, Brunekreef B, Hoekstra MO, Aalberse R, Smit HA. Asthma at 8 years of age in children born by caesarean section[J]. Thorax, 2009, 64(2): 107-113.
[URI]
|
| [18] |
Decker E, Engelmann G, Findeisen A, Gerner P, Laass M, Ney D, Posovszky C, Hoy L, Hornef MW. Cesarean delivery is associated with celiac disease but not inflammatory bowel disease in children[J]. Pediatrics, 2010, 125(6): e1433-1440.
[DOI]
|
| [19] |
Kero J, Gissler M, Grönlund MM, Kero P, Koskinen P, Hemminki E, Isolauri E. Mode of delivery and asthma—is there a connection?[J]. Pediatr Res, 2002, 52(1): 6-11.
[URI]
|
| [20] |
Huh SY, Rifas-Shiman SL, Zera CA, Edwards JW, Oken E, Weiss ST, Gillman MW. Delivery by caesarean section and risk of obesity in preschool age children: a prospective cohort study[J]. Arch Dis Child, 2012, 97(7): 610-616.
[DOI]
|
| [21] |
Fierer N, Hamady M, Lauber CL, Knight R. The influence of sex, handedness, and washing on the diversity of hand surface bacteria[J]. Proc Natl Acad Sci USA, 2008, 105(46): 17994-17999.
[DOI]
|
| [22] |
Schloss PD, Westcott SL, Ryabin T, Hall JR, Hartmann M, Hollister EB, Lesniewski RA, Oakley BB, Parks DH, Robinson CJ, Sahl JW, Stres B, Thallinger GG, Van Horn DJ, Weber CF. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Appl Environ Microbiol, 2009, 75(23): 7537-7541.
[DOI]
|
| [23] |
Aravena-Roman M, Sproer C, Straubler B, Inglis T, Yassin AF. Corynebacterium pilbarense sp. nov., a non-lipophilic Corynebacterium isolated from a human ankle aspirate[J]. Int J Syst Evol Microbiol,, 2010, 60(Pt 7): 1484-1487.
[URI]
|
| [24] |
Supré K, De Vliegher S, Cleenwerck I, Engelbeen K, Van Trappen S, Piepers S, Sampimon OC, Zadoks RN, De Vos P, Haesebrouck F. Staphylococcus devriesei sp. nov., isolated from teat apices and milk of dairy cows[J]. Int J Syst Evol Microbiol, 2010, 60(Pt 12): 2739-2744.
[URI]
|
| [25] |
Takahashi T, Satoh I, Kikuchi N. Phylogenetic relationships of 38 taxa of the genus Staphylococcus based on 16S rRNA gene sequence analysis[J]. Int J Syst Bacteriol, 1999, 49(Pt 2): 725-728.
[URI]
|
| [26] |
Brüggemann H, Henne A, Hoster F, Liesegang H, Wiezer A, Strittmatter A, Hujer S, Dürre P, Gottschalk G. The complete genome sequence of Propionibacterium acnes, a commensal of human skin[J]. Science, 2004, 305(5684): 671-673.
[DOI]
|
| [27] |
Dasen G, Smutny J, Teuber M, Meile L. Classification and identification of Propionibacteria based on ribosomal RNA genes and PCR[J]. Syst Appl Microbiol, 1998, 21(2): 251-259.
[DOI]
|
| [28] |
Ying S, Zeng DN, Chi L, Tan Y, Galzote C, Cardona C, Lax S, Gilbert J, Quan ZX. The influence of age and gender on skin-associated microbial communities in urban and rural human populations[J]. PLoS One, 2015, 10(10): e0141842.
[DOI]
|
| [29] |
Lehtimäki J, Karkman A, Laatikainen T, Paalanen L, von Hertzen L, Haahtela T, Hanski I, Ruokolainen L. Patterns in the skin microbiota differ in children and teenagers between rural and urban environments[J]. Sci Rep, 2017, 7: 45651.
[DOI]
|
| [30] |
Human Microbiome Project Consortium. Structure, function and diversity of the healthy human microbiome[J]. Nature, 2012, 486(7402): 207-214.
[DOI]
|
| [31] |
Mitra A, Macintyre DA, Marchesi JR, Lee YS, Bennett PR, Kyrgiou M. The vaginal microbiota, human papillomavirus infection and cervical intraepithelial neoplasia: what do we know and where are we going next?[J]. Microbiome, 2016, 4(1): 58.
[DOI]
|
| [32] |
Kolenbrander PE, Palmer RJ Jr, Periasamy S, Jakubovics NS. Oral multispecies biofilm development and the key role of cell-cell distance[J]. Nat Rev Microbiol, 2010, 8(7): 471-480.
[DOI]
|
| [33] |
Periasamy S, Kolenbrander PE. Central role of the early colonizer Veillonella sp. in establishing multispecies biofilm communities with initial, middle, and late colonizers of enamel[J]. J Bacteriol, 2010, 192(12): 2965-2972.
[DOI]
|
| [34] |
McCabe RM, Donkersloot JA. Adherence of Veillonella species mediated by extracellular glucosyltransferase from Streptococcus salivarius[J]. Infect Immun, 1977, 18(3): 726-734.
[URI]
|
| [35] |
Carletto-Körber FP, González-Ittig RE, Jiménez MG, Cornejo LS. Initial acquisition and genetic identity of Streptococcus mutans of mother-child pairs[J]. Pediatr Dent, 2010, 32(3): 205-211.
[URI]
|
| [36] |
Wescombe PA, Heng NC, Burton JP, Chilcott CN, Tagg JR. Streptococcal bacteriocins and the case for Streptococcus salivarius as model oral probiotics[J]. Future Microbiol, 2009, 4(7): 819-835.
[DOI]
|
| [37] |
Johnsborg O, Eldholm V, Bjørnstad ML, Håvarstein LS. A predatory mechanism dramatically increases the efficiency of lateral gene transfer in Streptococcus pneumoniae and related commensal species[J]. Mol Microbiol, 2008, 69(1): 245-253.
[DOI]
|
| [38] |
Ling Z, Liu X, Wang Y, Li L, Xiang C. Pyrosequencing analysis of the salivary microbiota of healthy Chinese children and adults[J]. Microb Ecol, 2013, 65(2): 487-495.
[DOI]
|
| [39] |
Fardini Y, Chung P, Dumm R, Joshi N, Han YW. Transmission of diverse oral bacteria to murine placenta: evidence for the oral microbiome as a potential source of intrauterine infection[J]. Infect Immun, 2010, 78(4): 1789-1796.
[DOI]
|
 2019, Vol. 14
2019, Vol. 14


