蛋白质的翻译过程分为起始、延伸、终止和核糖体再循环4个步骤,每个步骤都需要不同的蛋白质翻译因子参与[1-2],如起始因子(IF1、IF2、IF3),延伸因子(EF-Tu、EF-Ts、EF-G)及肽链释放因子(RF1、RF2、RF3)。然而, 在蛋白质翻译过程中,会因各种原因导致核糖体滞留于mRNA,引起蛋白质翻译停滞,此时需要一些因子如EF4、EF-P、ArfA(YhdL)、ArfB(YaeJ)等的参与来拯救停滞的核糖体或异常延伸,从而保证蛋白质合成的持续完成[3-6]。
EF4也被称作LepA[7],是一个高度保守蛋白[8-10],存在于真核生物的线粒体、叶绿体和几乎所有的原核生物[2]中。有意思的是,EF4在原核生物中具有高度的序列保守性[11],却不是一个生长必需的基因。细菌在正常生长情况下并不需要EF4作为延伸因子,但在特殊情况导致核糖体熄火时是否需要EF4作为延伸因子意见尚不一致。2006年Qin等[11]研究认为,EF4可通过将tRNA从E-和P-位点移动到P-和A-位点来反向转运核糖体。动力学研究表明,EF4诱导的反向移位需要几分钟才能完成[12]。此外,Liu等研究认为,在蛋白质翻译的延伸阶段,EF4的功能类似于EF-G,主要用于竞争性结合PRE核糖体复合物[13],且结合速率与EF-G一致,此特征使其可减缓肽链合成速率,从而促进新生多肽链边翻译边折叠。该研究结果也对EF4是否可反向转运核糖体提出质疑[13]。还有研究表明,真核生物中Guf1(EF4的同源物)的缺失会导致线粒体发生严重缺陷,使呼吸链复合体蛋白合成和活性受到严重影响[14]。大肠埃希菌中EF4是一个在压力条件下发挥作用的蛋白,在不同程度的应激反应中表现为促进细胞存活或细胞死亡两种功能[15-19]。有研究表明,鸟分枝杆菌中EF4编码基因的突变株在巨噬细胞内存活率下降[20],提示EF4可能与细菌毒力相关。总之,EF4是一个比较有争议的重要蛋白,不仅在某些特殊情况下作为延伸因子发挥作用,可能还存在其他生理功能。
结核分枝杆菌(Mycobacterium tuberculosis,M. tuberculosis)是导致人类结核病的重要病原菌,致病机制十分复杂。从富氧环境到在巨噬细胞内生存所面对的缺氧、一氧化氮杀伤、营养缺乏等极端生长条件,结核分枝杆菌需要调节自身去适应,EF4可能在其中发挥重要作用。为探索EF4的功能,需要构建EF4敲除突变株,但由于分枝杆菌体内重组率非常低,分枝杆菌的基因敲除一直是个难题。本研究利用高滴度噬菌体特异性转导基因敲除方法[21]成功构建了结核分枝杆菌H37Ra株lepA基因敲除株,为后续深入开展结核分枝杆菌的EF4功能研究奠定了基础。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒结核分枝杆菌H37Ra、耻垢分枝杆菌mc2155、大肠埃希菌DH5α均为本实验室保存;大肠埃希菌HB101、温敏型穿梭质粒phAE159质粒及p0004S质粒由美国W.R. Jacobs实验室提供。
1.1.2 试剂和培养基PrimeSTAR HS DNA Polymerase购自TaKaRa公司,PacI、Van91I、CIP碱性去磷酸化酶和T4连接酶购自NEB(北京)有限公司,MaxPlax Lambda Packaging Extract购自Epicentre公司,Middlebrook7H9/7H10购自美国BD公司,OADC [5%牛血清白蛋白购自Aamresco公司,0.03% catalase购自Sigma公司,2%葡萄糖、0.85%氯化钠和0.01%油酸购自生工生物工程(上海)股份有限公司],Tryptone/Yeast Extract购自Oxoid,BCA蛋白定量试剂盒购自北京原平皓生物技术有限公司,其他常用生化试剂购自生工生物工程(上海)股份有限公司和国药集团化学试剂有限公司。兔抗EF4、ICL1、EF-P多克隆抗体均由本实验室制备。
1.2 方法 1.2.1 p0004S-ΔlepA质粒的构建以结核分枝杆菌H37Ra全基因组DNA为模板,分别设计两对针对lepA基因的左、右臂引物,即lepA-LS(5′-TTTTTTTTCCATAAATTGGCCGAAATGGGC-GATTTTGCG-3′)、lepA-LA(5′-TTTTTTTTC-CATTTCTTGGTCGGGCGCGTCGAGGTGCCA-CAG-3′)和lepA-RS(5′-TTTTTTTTCCATAGA-TTGGTCCAGGGTATCCGCGTCGGG-3′)、lepA-RA(5′-TTTTTTTTCCATCTTTTGGGCATCGT-TAGCATCGGTGAC-3′)。通过聚合酶链反应(polymerase chain reaction,PCR)扩增lepA基因左、右臂。反应条件:98 ℃ 3 min;98 ℃ 10 s,64 ℃ 15 s,72 ℃ 1 min,29个循环;72 ℃ 5 min。采用Van91Ⅰ酶切lepA基因左、右臂和p0004S质粒,连接酶切后的片段,转化DH5α感受态细胞,涂布于含150 μg/mL潮霉素的LB固体平板,挑取单克隆菌落并抽提质粒,Van91Ⅰ酶切验证确定阳性克隆。
1.2.2 phAE159-ΔlepA穿梭质粒的构建PacⅠ分别酶切p0004S-ΔlepA质粒和phAE159质粒,连接两酶切后的片段,通过噬菌体体外包装试剂盒转化HB101感受态细胞,涂布于含150 μg/mL潮霉素的LB固体平板,挑取单克隆菌落并抽提质粒,PacⅠ酶切验证确定阳性克隆。
1.2.3 噬菌体扩增取2 μg phAE159-ΔlepA质粒电转入耻垢分枝杆菌mc2155感受态细胞,加入1 mL 7H9培养基复苏4 h,将复苏的菌液与top agar混匀,铺于LB平板上,30 ℃培养箱避光培养2~3 d。
挑4~5个噬菌斑到EP管中,加入200 μL MP缓冲液,4 ℃过夜。取上清液50 μL加至300 μL新鲜培养的mc2155菌液中,再与top agar混匀,铺于LB平板上。待噬菌斑紧密相连,将噬菌斑刮到离心管中,加入适量MP缓冲液,4 ℃过夜,用0.22 μm滤器过滤上清液,4 ℃保存。
将噬菌体进行10倍梯度稀释,取各梯度噬菌体加至新鲜培养的mc2155菌液中,再与top agar混匀,铺于LB平板上,培养2~3 d,计算噬菌斑滴度。
1.2.4 lepA基因敲除菌株的构建制备结核分枝杆菌H37Ra侵染感受态[21],以细菌:噬菌体=1:10的比例混匀,避光孵育3~4 h,补加10 mL 7H9+OADC,37 ℃孵育16~20 h,离心并涂布于含75 μg/mL潮霉素的7H10+OADC平板上,37 ℃培养4~6周,挑取生长的阳性克隆。
1.2.5 lepA基因敲除菌株的验证挑选单克隆菌落置于10 mL 7H9+OADC培养基中,静置培养3周,抽提敲除株全基因组DNA,以lepA基因左、右臂远端序列设计引物lepA-S(5′-GAGCAGACGC-AAAATCGCCCAAATTC-3′)和lepA-A(5′-GC-CGGTGCTCGACGGGAAGAAACTG-3′)进行PCR验证。PCR条件:98 ℃ 3 min;98 ℃ 10 s,68 ℃ 15 s,72 ℃ 6 min,29个循环;72 ℃ 5 min。PCR产物经1%琼脂糖凝胶电泳检测,回收目的条带并进行测序。
收集5 mL处于对数期的野生株和敲除株菌液,超声破碎细胞,用BCA蛋白定量试剂盒对上清液全菌蛋白进行浓度测定。取野生株和敲除株样品进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)和蛋白免疫印迹检测。
1.2.6 菌落形态及生长曲线调整野生株和敲除株密度至OD600=0.6,稀释后涂布于7H10+OADC平板上,培养6周,在连续变倍体显微镜下观察菌落形态。将生长密度为OD600=0.6的野生株和敲除株以1:50比例接种到7H9+OADC培养基中,每隔3 d取样测OD600,检测生长曲线。
1.2.7 细菌生物膜收集野生株和敲除株菌液,离心,弃上清液,用磷酸盐缓冲液(phosphate buffered saline,PBS)悬浮细菌,清洗2次。用Sauton培养基悬浮细菌,调整菌液密度至OD600=0.6,稀释50倍,在24孔板中加入1.3 mL菌液,四周孔加入1 mL无菌水,用封口膜封口。37 ℃静置培养4、6周后,在连续变倍体显微镜下观察细菌生物膜形成。
1.2.8 胁迫实验调整野生株和敲除株密度至OD600=0.6,分别以pH 5.0的7H9+OADC培养基、含0.05% SDS的7H9+OADC培养基、含5 mmol H2O2的7H9+OADC培养基于37 ℃处理菌液1.5 h及50 ℃水浴处理菌液1.5 h。对处理前后菌液进行10倍梯度稀释,吸取100 μL稀释液涂布于7H10+OADC平板上,37 ℃培养箱倒置培养4周后,进行细菌菌落形成单位计数。
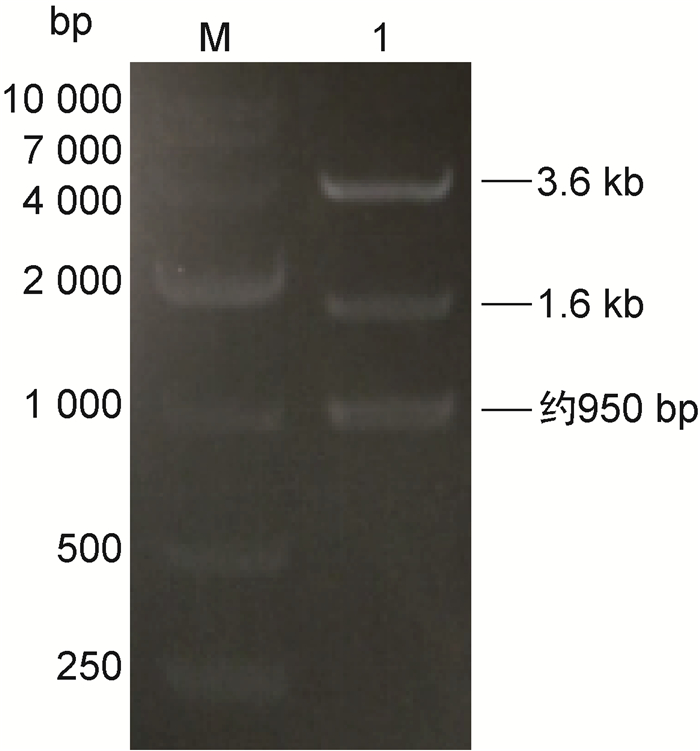
2 结果 2.1 p0004S-ΔlepA质粒的构建以结核分枝杆菌H37Ra全基因组DNA为模板,通过PCR扩增lepA基因左、右臂,大小均约950 bp。用Van91Ⅰ分别酶切p0004S质粒及lepA基因左、右臂PCR产物,胶回收p0004S质粒上3.6、1.6 kb片段,以及lepA基因左、右臂片段。连接4个片段并转化大肠埃希菌DH5α感受态细胞,涂布于含150 μg/mL潮霉素的LB平板上筛选阳性克隆。抽提质粒,经Van91Ⅰ酶切验证,分别获得3.6、1.6 kb和950 bp的片段,与预期目的条带一致(图 1)。进行测序验证,约950 bp的片段对应lepA基因左、右臂,表明成功构建p0004S-ΔlepA质粒。
|
| Lane M: DL10000 marker; lane 1: p0004S-ΔlepA plasmid. 图 1 p0004S-ΔlepA质粒限制性酶切鉴定 Fig. 1 Restriction enzyme digestion identification of p0004S-ΔlepA plasmid |
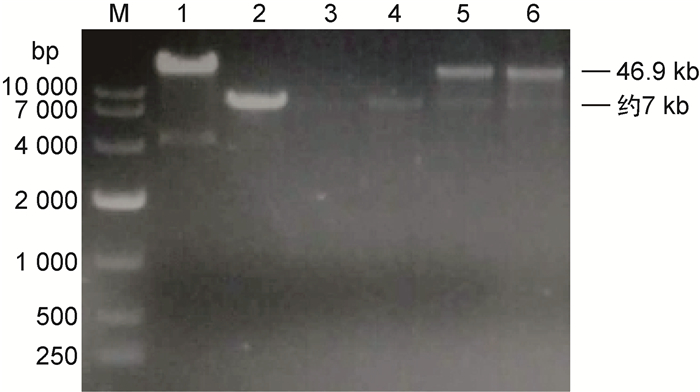
用PacⅠ酶切phAE159质粒(46.9 kb)和p0004S-ΔlepA质粒(约7 kb),连接两酶切后的片段。通过噬菌体体外包装试剂盒,将酶连产物转化HB101感受态,涂布于含150 μg/mL潮霉素的LB平板上筛选阳性克隆。挑取4个单克隆菌落(图 2中3~6泳道),抽提质粒,经PacⅠ酶切验证,有2个重组质粒分别获得约7 kb的小片段和>10 kb的大片段,与预期目的条带一致(图 2中5~6泳道)。同时,将PacⅠ酶切phAE159质粒作为阴性对照(图 2中1泳道),酶切p0004S-ΔlepA质粒作为阳性对照(图 2中2泳道)。经测序验证,两片段分别对应p0004S-ΔlepA质粒和phAE159质粒,表明克隆3与克隆4(图 2中5~6泳道)为成功构建的phAE159-ΔlepA穿梭质粒。
|
| Lane M: DL10000 marker; 1ane 1: phAE159 plasmid; lane 2: p0004S-ΔlepA plasmid; lane 3-6: four phAE159-ΔlepA plasmids respectively. 图 2 phAE159-ΔlepA穿梭质粒限制性酶切鉴定 Fig. 2 Restriction enzyme digestion identification of phAE159-ΔlepA plasmid |
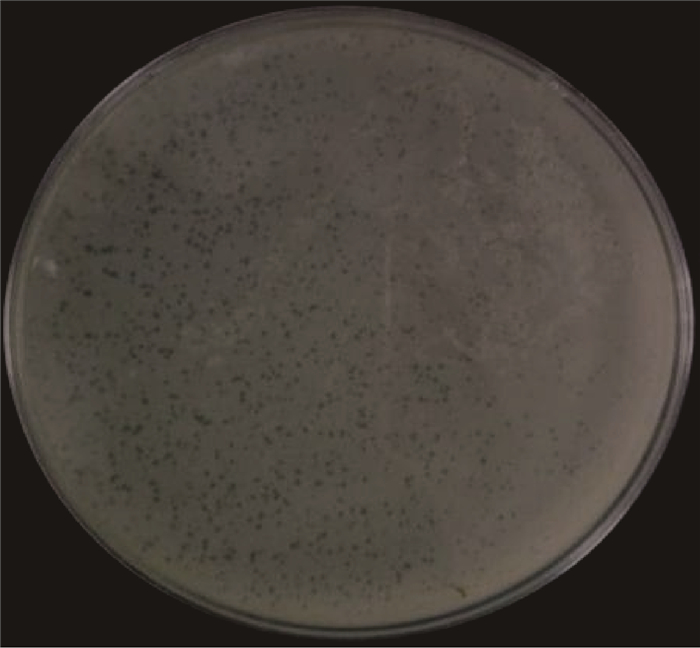
取2 μg phAE159-ΔlepA穿梭质粒电转耻垢分枝杆菌mc2155感受态细胞,37 ℃复苏4 h或过夜,加入top agar混匀,铺于LB平板上,30 ℃培养箱避光培养2~3 d,可观察到生长出的噬菌斑(图 3)。
|
| 图 3 噬菌体phAE159-ΔlepA Fig. 3 Phage of phAE159-ΔlepA |
挑取4~5个噬菌斑到EP管中,加入200 μL MP缓冲液,4 ℃过夜。取上清液50 μL加至300 μL新鲜培养的mc2155菌液中,再与top agar混匀,铺于LB平板上。待噬菌斑紧密相连,将噬菌斑刮到离心管中,加入适量MP缓冲液,4 ℃过夜,用0.22 μm滤器过滤上清液,4 ℃保存。
将噬菌体以10倍梯度进行稀释,取各梯度噬菌体与新鲜培养的mc2155菌液混合。以上述同样方式铺于LB平板上,2~3 d后统计平板上噬菌斑个数,计算噬菌体滴度,达1010 cfμ/mL以上即可。
2.4 lepA基因敲除菌株的构建将噬菌体侵染结核分枝杆菌H37Ra感受态细胞,侵染的感染复数(multiplicity of infection,MOI)为细菌:噬菌体=1:10。在含有75 μg/mL潮霉素的7H10+OADC平板上37 ℃培养4周时,出现一个结核分枝杆菌微小单克隆菌落。继续培养至6周时,挑取该单克隆菌落于液态7H9+OADC培养基中培养3周。抽提菌株基因组DNA为模板,以lepA基因左臂远端序列和右臂远端序列设计正、反向引物(lepA-S/lepA-A),进行PCR鉴定。鉴定原理如图 4A所示。琼脂糖凝胶电泳检测显示,野生株(wild-type,WT)扩增得到包含左臂(950 bp)、lepA全基因(1 962 bp)及右臂(950 bp)共约4 kb的条带,但由于结核分枝杆菌全基因组中的lepA基因被sacB+hyg区域(3 600 bp)置换,lepA基因敲除株扩增得到的条带约5.5 kb,与预期目的条带相符(图 4B)。将目的片段切胶回收后进行序列测定,结果显示结核分枝杆菌全基因组中的lepA基因已被sacB+hyg区域置换,提示该阳性克隆为构建成功的结核分枝杆菌lepA基因敲除株(RaΔlepA,KO)。

|
| A: Schematic diagram of PCR identification. B: Identification of lepA gene deletion mutant by PCR. 图 4 lepA基因敲除株的构建 Fig. 4 Construction of lepA gene deletion mutant |
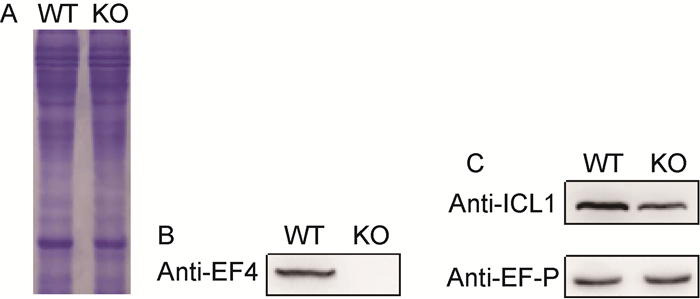
利用兔抗EF4多克隆抗体进一步检测野生株和敲除株中EF4的表达。将生长至对数期的野生株和敲除株超声破碎后,取50 μg菌体蛋白上清液分别进行SDS-PAGE和蛋白免疫印迹检测。SDS-PAGE检测结果表明野生株和敲除株全菌蛋白上样量一致(图 5A)。蛋白免疫印迹检测结果显示,野生株中有EF4表达,敲除株中无EF4表达(图 5B),进一步表明lepA基因已成功从结核分枝杆菌菌株中被敲除。此外,检测EF4敲除株中其他蛋白的表达变化,包括与结核分枝杆菌潜伏感染密切相关的异柠檬酸裂解酶ICL1和翻译过程中的重要延伸因子且是结核分枝杆菌生长必需的EF-P。结果显示,lepA基因敲除后ICL1表达量下调(相对分子质量47×103),EF-P表达量无明显差异(相对分子质量20×103)。
|
| A: SDS-PAGE of wild-type and lepA gene deletion mutant. B: EF4 detected by Western blotting. C: ICL1 and EF-P detected by Western blotting. 图 5 lepA基因敲除株的EF4、ICL1、EF-P蛋白表达水平 Fig. 5 Protein expression of EF4, ICL1, EF-P in lepA gene deletion mutant |
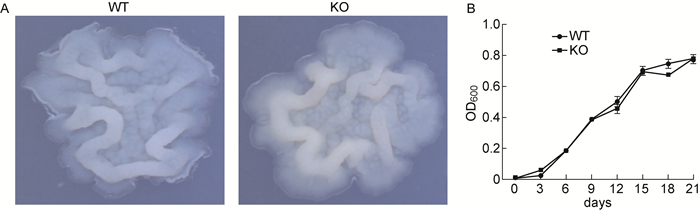
为分析敲除株的菌落形态和生长曲线是否存在变化,取7H10+OADC平板上生长6周的敲除株和野生株的菌落在连续变倍体显微镜下进行菌落形态观察。结果发现两者形态存在差异,相比于野生株,敲除株皱褶更聚集,形态偏厚,颜色发黄(图 6A)。但在正常培养条件下,野生株与敲除株生长趋势一致,无明显差异(图 6B),表明EF4编码基因的敲除对正常条件下结核分枝杆菌的生长无影响。
|
| A: Colony morphology. B: Growth curve. 图 6 野生株和敲除株菌落形态及生长曲线分析 Fig. 6 Analysis of colony morphology and growth curve in wild-type and lepA gene deletion mutant |
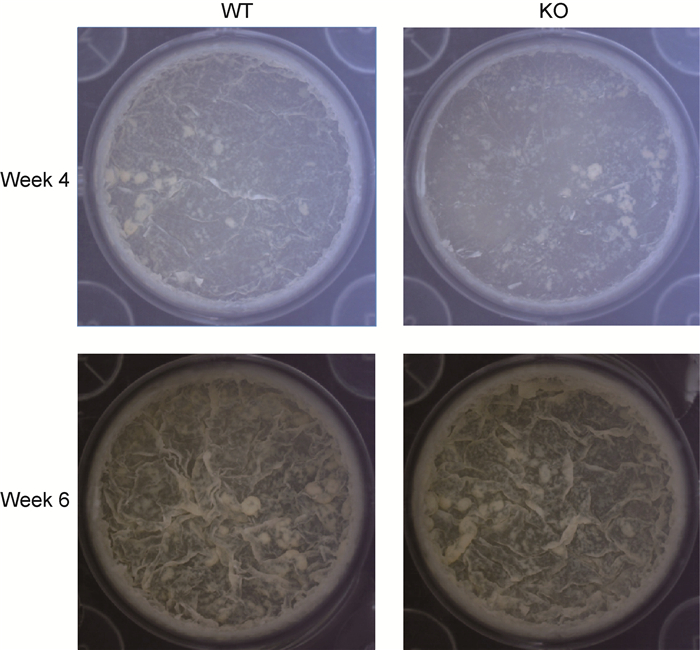
为分析敲除株的生物膜形成能力,采用Sauton培养基在24孔板中分别培养野生株和敲除株4、6周后,利用连续变倍体显微镜观察两者生物膜形成。结果显示,细菌生长4周时,野生株和敲除株均在液面表层形成一层薄的生物膜,且野生株形成少量皱褶,而敲除株几乎没有皱褶形成;6周时,野生株和敲除株均观察到形成大量褶皱,相较于野生株,敲除株生物膜皱褶偏少,且趋于分散(图 7)。
|
| Biofilm formation were analysed after four weeks and seven weeks respectively. 图 7 野生株和敲除株生物膜形成能力分析 Fig. 7 Analysis of biofilm formation in wild-type and lepA gene deletion mutant |
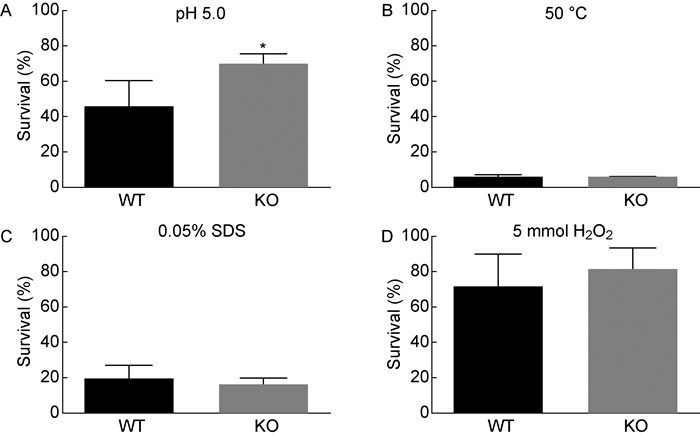
为检测EF4是否与细菌抗逆相关,分别用pH 5.0的7H9+OADC培养基、含0.05% SDS的7H9+OADC培养基、含5 mmol H2O2的7H9+OADC培养基以及50 ℃水浴处理结核分枝杆菌野生株和EF4敲除株,比较野生株与敲除株在耐酸、耐热、抗去垢剂、抗氧化逆境中的存活率。结果显示,相较于野生株,敲除株耐酸能力显著提高(图 8A),耐热、抗去垢剂、抗氧化能力无显著差异(图 8B~D)。
|
| A: pH resistance; B: Heat resistance; C: Detergent resistance; D: Oxidant resistance. 图 8 野生株和敲除株耐胁迫能力分析 Fig. 8 Analysis of stress resistance in wild-type and lepA gene deletion mutant |
结核病是一种慢性致死性疾病,其致死率仅次于艾滋病,严重威胁着人类健康。据统计,全世界约1/3的人口是结核分枝杆菌携带者。据世界卫生组织(World Health Organization,WHO)2018年《全球结核病报告》显示,2017年有1 000万人感染结核分枝杆菌,157万人死亡。结核分枝杆菌的致病机制十分复杂,全基因组4 000个左右的编码序列中有很多基因的功能还未知,对这些未知功能基因进行研究十分必要。
基因敲除是研究基因功能的重要技术,但结核分枝杆菌基因敲除株的构建十分困难,主要原因如下:①结核分枝杆菌细胞壁厚,含有大量分枝菌酸,外源质粒难以进入;②结核分枝杆菌基因组同源重组效率低,传统的利用同源重组原理将含有等位交换底物的自杀性质粒电转到细菌中的敲除方法获得的单交换及假阳性率高;③结核分枝杆菌生长周期长,单克隆生长需要3~4周,故敲除株构建周期长。
本研究利用高滴度温敏型噬菌体特异性侵染分枝杆菌,使靶向目的基因发生同源重组,高效实现了lepA基因敲除株的构建。该方法将同源重组区域插入phAE159质粒基因组中,而phAE159质粒含有分枝杆菌噬菌体的编码信息,利用这一特性,用重组的分枝杆菌噬菌体侵染结核分枝杆菌感受态细胞进行高效率同源重组;同时获得的phAE159-ΔlepA为穿梭质粒,既可在大肠埃希菌又可在分枝杆菌中复制。这一特性使得前期实验可在大肠埃希菌和耻垢分枝杆菌中进行操作,大大缩短了敲除所需的实验周期[22]。
本研究所用噬菌体是温敏型噬菌体,在30 ℃裂解生长,在37 ℃溶原生长[23]。故可在30 ℃感染耻垢分枝杆菌进行噬菌体扩增,在37 ℃侵染结核分枝杆菌,使之溶原生长整合到结核分枝杆菌基因组中,从而提高同源重组和基因敲除的效率。尽管该方法的基因敲除过程比较复杂,但最终筛选成功敲除菌株的概率通常较高[21],本研究中平板上仅出现1个阳性结核分枝杆菌克隆即为成功的敲除株也证实其定位敲除的高效性。
生长曲线分析表明,正常培养条件下结核分枝杆菌野生株与lepA基因敲除株生长趋势一致。在大肠埃希菌lepA敲除株中也发现,lepA基因敲除对细菌正常生长状态没有显著影响[24]。研究显示,大肠埃希菌中EF4可在高Mg2+浓度[15]、低pH[16]和低温[15]压力条件下加速蛋白质合成,并为这些不利生长条件下的细胞提供生存能力方面的优势。另一项研究也表明,EF4有助于大肠埃希菌对亚碲酸盐的抗性[17]。另一方面,EF4编码基因lepA的缺失可在几种致死应激条件下帮助其存活[18]。总之,这些研究结果表明,EF4在体内可能具有多种生理功能。本研究发现,结核分枝杆菌EF4编码基因lepA敲除后,细菌的耐酸特性明显增强,在鸟分枝杆菌EF4突变株中也有类似发现[20]。尽管鸟分枝杆菌EF4突变株研究发现突变株在巨噬细胞内生长能力下降,但此研究中感染巨噬细胞的野生菌株与突变菌株的起始感染量有很大差异,所以认为EF4影响鸟分枝杆菌的毒力还需要进一步斟酌。本研究发现,EF4敲除株菌落颜色发黄,凸起偏厚,通常在结核分枝杆菌脂质合成或细胞壁结构发生变异时会导致菌落表型发生类似变化[25],提示EF4可能与结核分枝杆菌的细胞壁脂质合成有关。结核分枝杆菌的脂质与其毒力和宿主内生存密切相关,且异柠檬酸裂解酶ICL1是结核分枝杆菌细胞内生存的关键基因,EF4敲除后明显影响了其表达;同时考虑到结核分枝杆菌EF4突变株耐酸性增强,提示lepA可能与分枝杆菌的毒力相关,但需要利用动物感染模型来深入探索。
| [1] |
Schmeing TM, Ramakrishnan V. What recent ribosome structures have revealed about the mechanism of translation[J]. Nature, 2009, 461(7268): 1234-1242.
[DOI]
|
| [2] |
Margus T, Remm M, Tenson T. Phylogenetic distribution of translational GTPases in bacteria[J]. BMC Genomics, 2007, 8: 15.
[DOI]
|
| [3] |
Lassak J, Wilson DN, Jung K. Stall no more at polyproline stretches with the translation elongation factors EF-P and IF-5A[J]. Mol Microbiol, 2016, 99(2): 219-235.
[DOI]
|
| [4] |
Huter P, Müller C, Beckert B, Arenz S, Berninghausen O, Beckmann R, Wilson DN. Structural basis for ArfA-RF2-mediated translation termination on mRNAs lacking stop codons[J]. Nature, 2017, 541(7638): 546-549.
[DOI]
|
| [5] |
Ma C, Kurita D, Li N, Chen Y, Himeno H, Gao N. Mechanistic insights into the alternative translation termination by ArfA and RF2[J]. Nature, 2017, 541(7638): 550-553.
[DOI]
|
| [6] |
Chadani Y, Ono K, Kutsukake K, Abo T. Escherichia coli YaeJ protein mediates a novel ribosome-rescue pathway distinct from SsrA-and ArfA-mediated pathways[J]. Mol Microbiol, 2011, 80(3): 772-785.
[DOI]
|
| [7] |
March PE, Inouye M. Characterization of the lep operon of Escherichia coli (Identification of the promoter and the gene upstream of the signal peptidase I gene)[J]. J Biol Chem, 1985, 260(12): 7206-7213.
[PubMed]
|
| [8] |
Caldon CE, Yoong P, March PE. Evolution of a molecular switch:universal bacterial GTPases regulate ribosome function[J]. Mol Microbiol, 2001, 41(2): 289-297.
[DOI]
|
| [9] |
Kiser GL, Weinert TA. GUF1, a gene encoding a novel evolutionarily conserved GTPase in budding yeast[J]. Yeast, 1995, 11(13): 1311-1316.
[DOI]
|
| [10] |
March PE, Inouye M. GTP-binding membrane protein of Escherichia coli with sequence homology to initiation factor 2 and elongation factors Tu and G[J]. Proc Natl Acad Sci USA, 1985, 82(22): 7500-7504.
[DOI]
|
| [11] |
Qin Y, Polacek N, Vesper O, Staub E, Einfeldt E, Wilson DN, Nierhaus KH. The highly conserved LepA is a ribosomal elongation factor that back-translocates the ribosome[J]. Cell, 2006, 127(4): 721-733.
[DOI]
|
| [12] |
Liu H, Pan D, Pech M, Cooperman BS. Interrupted catalysis:The EF4 (LepA) effect on back-translocation[J]. J Mol Biol, 2010, 396(4): 1043-1052.
[DOI]
|
| [13] |
Liu H, Chen C, Zhang H, Kaur J, Goldman YE, Cooperman BS. The conserved protein EF4 (LepA) modulates the elongation cycle of protein synthesis[J]. Proc Natl Acad Sci USA, 2011, 108(39): 16223-16228.
[DOI]
|
| [14] |
Zhu P, Liu Y, Zhang F, Bai X, Chen Z, Shangguan F, Zhang B, Zhang L, Chen Q, Xie D, Lan L, Xue X, Liang XJ, Lu B, Wei T, Qin Y. Human elongation factor 4 regulates cancer bioenergetics by acting as a mitochondrial translation switch[J]. Cancer Res, 2018, 78(11): 2813-2824.
[DOI]
|
| [15] |
Pech M, Karim Z, Yamamoto H, Kitakawa M, Qin Y, Nierhaus KH. Elongation factor 4 (EF4/LepA) accelerates protein synthesis at increased Mg2+ concentrations[J]. Proc Natl Acad Sci USA, 2011, 108(8): 3199-3203.
[DOI]
|
| [16] |
Yang F, Li Z, Hao J, Qin Y. EF4 knockout E.coli cells exhibit lower levels of cellular biosynthesis under acidic stress[J]. Protein Cell, 2014, 5(7): 563-567.
[DOI]
|
| [17] |
Shoji S, Janssen BD, Hayes CS, Fredrick K. Translation factor LepA contributes to tellurite resistance in Escherichia coli but plays no apparent role in the fidelity of protein synthesis[J]. Biochimie, 2010, 92(2): 157-163.
[PubMed]
|
| [18] |
Li L, Hong Y, Luan G, Mosel M, Malik M, Drlica K, Zhao X. Ribosomal elongation factor 4 promotes cell death associated with lethal stress[J]. MBio, 2014, 5(6): e01708.
[PubMed]
|
| [19] |
Badu-Nkansah A, Sello JK. Deletion of the elongation factor 4 gene (lepA) in Streptomyces coelicolor enhances the production of the calcium-dependent antibiotic[J]. FEMS Microbiol Lett, 2010, 311(2): 147-151.
[PubMed]
|
| [20] |
Frigui W, Bottai D, Majlessi L, Monot M, Josselin E, Brodin P, Garnier T, Gicquel B, Martin C, Leclerc C, Cole ST, Brosch R. Control of M. tuberculosis ESAT-6 secretion and specific T cell recognition by PhoP[J]. PLoS Pathog, 2008, 4(2): e33.
[DOI]
|
| [21] |
Jain P, Hsu T, Arai M, Biermann K, Thaler DS, Nquyen A, González PA, Tufariello JM, Kriakov J, Chen B, Larsen MH, Jacobs WR Jr. Specialized transduction designed for precise high-throughput unmarked deletions in Mycobacterium tuberculosis[J]. MBio, 2014, 5(3): e01245-14.
[PubMed]
|
| [22] |
Bardarov S, Bardarov S Jr, Pavelka MS Jr, Sambndamurthy V, Larsen M, Tufariello J, Chan J, Hatfull G, Jacobs WR Jr. Specialized transduction:an efficient method for generating marked and unmarked targeted gene disruptions in Mycobacterium tuberculosis, M. bovis BCG and M. smegmatis[J]. Microbiology, 2002, 148(Pt 10): 3007-3017.
[PubMed]
|
| [23] |
Bhatt A, Fujiwara N, Bhatt K, Gurcha SS, Kremer L, Chen B, Chan J, Porcelli SA, Kobayashi K, Besra GS, Jacobs WR Jr. Deletion of kasB in Mycobacterium tuberculosis causes loss of acid-fastness and subclinical latent tuberculosis in immunocompetent mice[J]. Proc Natl Acad Sci USA, 2007, 104(12): 5157-5162.
[DOI]
|
| [24] |
Dibb NJ, Wolfe PB. lep operon proximal gene is not required for growth or secretion by Escherichia coli[J]. J Bacteriol, 1986, 166(1): 83-87.
[DOI]
|
| [25] |
Glickman MS, Cox JS, Jacobs WR Jr. A novel mycolic acid cyclopropane synthetase is required for cording, persistence, and virulence of Mycobacterium tuberculosis[J]. Mol Cell, 2000, 5(4): 717-727.
[DOI]
|
 2019, Vol. 14
2019, Vol. 14


