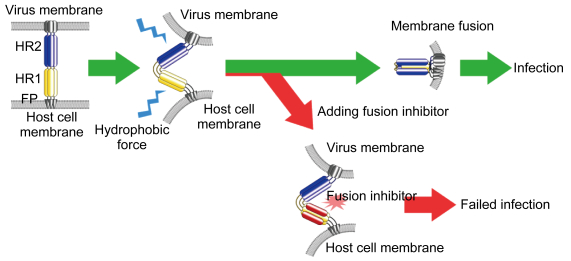
Ⅰ型膜融合蛋白是一类广泛分布于Ⅰ型包膜病毒表面的膜蛋白,可通过形成螺旋结构来介导病毒包膜与宿主细胞膜的融合过程,从而实现病毒感染。Ⅰ型膜融合蛋白的主要功能区域分为N端融膜肽FP、N端螺旋区HR1与C端螺旋区HR2[1]。在早期病毒感染过程中,病毒膜蛋白与宿主细胞膜表面的受体结合,融合蛋白发生结构改变,FP插入宿主细胞膜,同时HR1与HR2暴露于亲水环境中,导致结构不稳定而进一步折叠,形成六螺旋结构(six-helix bundle,6HB),使病毒包膜与宿主细胞膜彼此靠近,发生膜融合(图 1)[2-7]。基于抑制6HB形成的多肽类病毒融合抑制剂是一类多肽片段,通过模拟病毒HR1-HR2区段的氨基酸序列,在病毒6HB形成过程中与病毒蛋白发生竞争性结合,阻碍6HB正常形成,进而抑制病毒融合过程(图 1)。1993年,姜世勃等首次报道了第1个针对人类免疫缺陷病毒1型(human immunodeficiency virus type 1,HIV-1)gp41蛋白的病毒融合抑制多肽——SJ-2179[8-9]。1994年,Wild等报道了另一个多肽——DP-178[10]。2003年,美国食品药品管理局(Food and Drug Administration,FDA)批准恩夫韦肽(T-20)为第1个基于病毒融合抑制原理的抗HIV多肽药物,用于治疗对常用抗HIV药物产生耐受的艾滋病患者。
|
| 图 1 Ⅰ型膜融合蛋白介导的膜融合过程可被多肽类病毒融合抑制剂所抑制 Fig. 1 Class I membrane fusion protein-mediated membrane fusion could be terminated by virus fusion inhibitors |
基于抑制6HB形成的多肽类融合抑制剂的序列来源于病毒原始序列。在大多数情况下,病毒的原始序列完全能明显表现出病毒包膜蛋白介导的膜融合抑制活性。但为追求更好的抑制效果,研究者一般会在融合抑制剂序列中引入突变,从而获得更强的抑制活性。传统融合抑制剂序列的设计与优化,建立在对病毒融合蛋白尤其是HR1-HR2区段三级结构及四级结构的充分认识之上。虽然可通过helical wheel模型[11-14]并根据二级结构建立氨基酸侧链之间的相互作用关系进行优化设计,但这种相互作用关系并不准确,优化设计的成功率往往较低,难以在较短时间内确定有活性的优化位点。对于更高效率的序列优化,则需结合蛋白质晶体结构信息,利用tripartite模型[15]确定精确的氨基酸相互作用。
基于抑制6HB形成的多肽类融合抑制剂不仅可用于治疗慢性感染,还可用于新发突发传染病的应急救治。近年来,随着人类社会的发展,不同地区的人员往来日益增多,造成新发突发传染病频繁暴发,如严重急性呼吸综合征(severe acute respiratory syndrome,SARS)[16]、中东呼吸综合征(Middle East respiratory syndrome, MERS)[17]、埃博拉出血热(Ebola hemorrhagic fever)[18]等,造成严重人员死亡与广泛恐慌。多肽类病毒融合抑制剂的安全性高,活性强,研发流程较简单,是应对突发传染性疾病的候选急救药物之一。然而,人们对导致新发突发传染病的病毒往往缺少蛋白质结构的详细信息,利用传统优化设计手段开发药物较为盲目,准确性较低,且主观性较大,严重影响了开发工作的效率。因此,有必要开发一种新的不依赖病毒蛋白三级结构信息的设计与优化方法,作为多肽类病毒融合抑制剂优化设计的辅助手段,以克服传统设计方法的缺点。
随着计算机技术的发展,机器的运算、学习能力不断增强,利用计算机工具分析蛋白质信息逐渐成为常用的研究手段,尤其是对蛋白质结构的分析与稳定性预测。利用计算机软件开发基于抑制6HB形成的多肽类融合抑制剂能弥补传统设计手段的不足。融合抑制剂与病毒蛋白之间形成的6HB与病毒融合蛋白的天然6HB有相似的三维结构,两者之间也存在相似的氨基酸相互作用和相似的热力学性质,因此融合抑制剂的抑制活性与6HB结构之间的稳定性相关。若某位点的突变能使6HB更加稳定,则该位点对应位置的突变可能导致病毒融合抑制剂与病毒融合蛋白的结合能力升高,即抑制活性增强。I-Mutant2.0是一种在线蛋白质稳定性预测软件,其计算方法建立在对大量蛋白质及其突变后稳定性变化数据库的机器学习[19]。通过分析蛋白质的一级结构信息,该软件可预测出某一点引入某特定突变后蛋白质整体稳定性的变化规律。I-Mutant2.0常用于设计高度稳定的蛋白质结构,或验证序列中某一位置对突变的敏感性等信息。研究者可通过I-Mutant2.0来寻找6HB中的稳定性突变,进而开展针对特定病毒6HB形成的多肽类病毒融合抑制剂的设计工作。
为验证I-Mutant2.0作为多肽类融合抑制剂辅助设计手段的可行性,本研究以中东呼吸综合征冠状病毒(Middle East respiratory syndrome coronavirus,MERS-CoV)作为研究对象,设计基于抑制6HB形成的多肽类病毒融合抑制剂。MERS最早在2012年于中东地区暴发,是一种由MERS-CoV感染引起的传染性疾病[17]。MERS-CoV是Ⅰ型包膜病毒,其膜表面S蛋白三聚体介导病毒膜融合过程[20-23]。MERS-CoV S蛋白的HR2及相邻的不规则螺旋区段表现出较强的病毒抑制活性,是设计病毒融合抑制剂的高效位点。本研究利用I-Mutant2.0作为辅助手段,预测S蛋白的HR2及相邻区段发生特定突变后对MERS-CoV S蛋白6HB整体稳定性的影响,寻找有利于设计MERS-CoV融合抑制剂的突变,并根据预测结果设计4条验证性多肽。圆二色谱(circular dichroism spectrum,CD)测量、细胞-细胞融合抑制及假病毒融合抑制等结果显示,不同多肽的病毒抑制活性受突变影响而变化的规律符合预期,证明I-Mutant2.0可用于辅助设计多肽类病毒膜融合抑制。
1 材料与方法 1.1 材料 1.1.1 细胞系293T细胞由本实验室保存,Huh-7细胞购于中国科学院细胞库,均用含10%胎牛血清(fetal bovine serum,FBS)的DMEM培养基,于37 ℃、5%CO2条件下培养。
1.1.2 质粒pAAV-IRES-EGFP与pAAV-IRES-MERS-EGFP质粒均为本实验室于先期研究工作中构建并保存。pcDNA3.1-MERS-S及pNL4-3.luc.RE由美国血液中心杜兰英博士惠赠。
1.1.3 检测试剂盒荧光素酶检测试剂盒由美国Thermal Scientific公司生产。
1.2 方法 1.2.1 利用I-Mutant2.0预测蛋白质的结构稳定性I-Mutant2.0(http://folding.biofold.org/i-mutant/i-mutant2.0.html)是一款免费的蛋白质稳定性分析软件,可基于蛋白质的PDB ID或序列一级结构信息分析单个突变对蛋白质整体稳定性的影响[19]。向服务器提交希望引入突变的位置与突变种类后,突变后蛋白质稳定性的变化会以吉布斯自由能变化量(ΔΔ G)的形式呈现。ΔΔ G指某一假定的热力学变化过程始末,蛋白分子内能变化量受突变影响而产生差异的值,常用于蛋白质的分子动力学分析[24-27]。如突变使蛋白质趋于稳定,则ΔΔ G>0,反之则表明突变后蛋白质稳定性下降。MERS-CoV S蛋白HR2区段及相邻线性区域序列已被证明具有抗MERS-CoV感染的融合抑制剂活性,因此本研究选择S蛋白6HB氨基酸序列作为研究对象(PDB ID:4njl),提交每个位点的19种天然氨基酸突变。
1.2.2 分析验证性多肽的二级结构性质MERS-CoV S蛋白HR1、HR2区段衍生多肽在溶液中相互作用,能形成稳定的二级结构,可被圆二色谱检测探知[28-29]。根据圆二色谱检测的基本原理,将每种被检测多肽以磷酸盐缓冲液(phosphate buffered saline,PBS)(pH 7.4)稀释至终浓度为10 μmol/L,温度设定为20 ℃。通过J-815圆二色谱检测仪测得溶液平均残基椭偏率[θ],[θ]222为-33 000 deg·cm2·dmol-1时溶液中的多肽完全呈现螺旋状态。多肽复合物的热稳定性通过[θ]222进行测量,将溶液从20 ℃逐步升温至99 ℃,升温幅度5 ℃/min。热变性曲线通过平滑处理后,设定上升斜率最大处为熔解温度(melting temperature,Tm)。
1.2.3 MERS-CoV S蛋白介导细胞-细胞融合抑制实验细胞-细胞融合抑制实验常用于测定药物的抗病毒活性[30-31]。根据细胞-细胞融合抑制实验的基本原理,本研究以表达MERS-CoV S蛋白与增强绿色荧光蛋白(enhanced green fluorescent protein,EGFP)的293T细胞作为效应细胞,以表达DPP4的Huh-7细胞作为靶细胞[23]。将Huh-7细胞预先培养于含10%FBS的DMEM培养基,在96孔板中形成单层细胞,每孔加入1×104个转染了pAAV-IRES-MERS-EGFP质粒的293T细胞。293T细胞与不同浓度的多肽预混(转染了pAAV-IRES-EGFP的293T细胞以相同条件与Huh-7细胞混合,作为阴性对照)并继续培养。由于293T细胞表达绿色荧光蛋白,通过荧光显微镜观察细胞中绿色荧光蛋白的分布即可获知细胞融合情况。将293T细胞与Huh-7细胞共同培养3 h,取出96孔板并在荧光显微镜下拍照。每孔取5个视野(上、中、下、左、右),计算融合细胞数目及未融合细胞数目的平均数。细胞-细胞融合抑制率根据如下公式计算:[1-(E-N)/(P-N)]×100%。其中,E代表实验组中融合细胞占所有细胞的比例;P代表阳性对照组中(多肽浓度为0)融合细胞占所有细胞的比例;N代表阴性对照组中融合细胞占所有细胞的比例。多肽的细胞-细胞融合抑制活性可通过半数抑制浓度(half maximal inhibitory concentration,IC50)比较。
1.2.4 MERS-CoV假病毒抑制实验假病毒包装与感染系统常用于冠状病毒的感染与药物研究[32]。根据假病毒包装与感染的基本原理,本研究利用MERS-CoV S蛋白真核表达质粒pcDNA3.1-MERS-S与HIV骨架质粒pNL4-3.luc.RE共同转染293T细胞,获得表面包装MERS-CoV S蛋白的假病毒颗粒。假病毒骨架中包含荧光素酶报告基因,感染后可利用荧光素酶检测试剂盒测定假病毒感染水平。将pcDNA3.1-MERS-S与pNL4-3.luc.RE共同转染293T细胞,用含10%FBS的DMEM于37 ℃培养48 h,收取上清液。为测定假病毒浓度,将Huh-7细胞培养于96孔板,每孔2×104个。将含假病毒的上清液倍比稀释后加入96孔板,感染Huh-7细胞12 h后弃去含病毒的培养基,更换新鲜的含2%FBS的DMEM继续培养72 h,然后弃去上清液,用荧光素酶检测试剂盒检测Huh-7细胞中荧光素酶表达水平。选取荧光强度为10 000~20 000时的假病毒悬液,进行下一步假病毒感染抑制实验。将假病毒与不同浓度多肽混合后,加入Huh-7细胞,感染12 h,换液后继续培养72 h,检测荧光素酶,过程如前文所述。假病毒感染抑制率根据如下公式计算:[1-(E-N)/(P-N)]×100%。其中,E代表实验组中荧光强度;P代表阳性对照组(不加入多肽)中荧光强度;N代表阴性对照组(不加入假病毒)中荧光强度。
1.3 统计学方法所有实验结果均来自至少3次重复独立实验的数据。采用GraphPad Prism 6.0软件分析数据,行未配对双尾t检验,P<0.05为差异有统计学意义。
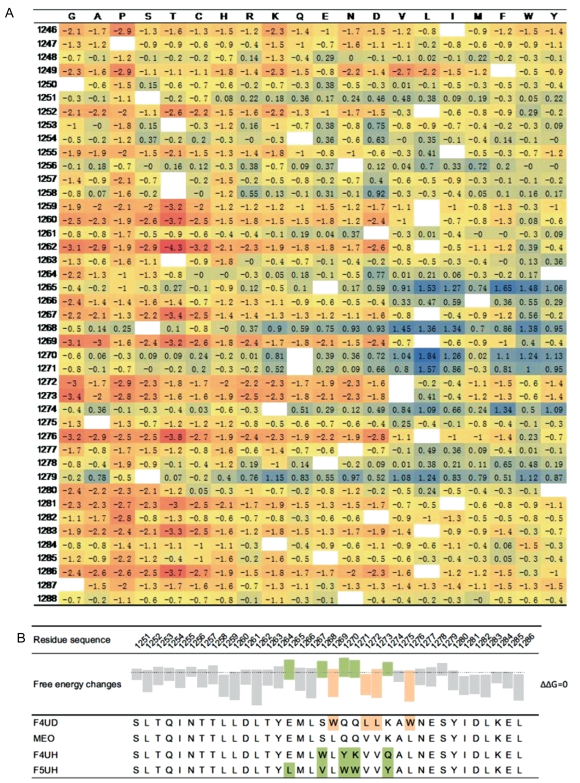
2 结果 2.1 利用I-Mutant2.0设计MERS-CoV多肽类融合抑制剂为寻找促使6HB更加稳定的突变位点,本研究采用I-Mutant2.0预测MERS-CoV S蛋白6HB序列发生突变后对蛋白整体稳定性的影响,分析第1 251~1 288位氨基酸位点中每个位点可能发生的19种天然氨基酸突变,并将结果汇总(图 2A)。结果显示,在分析的所有氨基酸位点中,一些位点的平均ΔΔ G明显高于总体水平,意味着这些位置上稳定性突变较多,优化后可能造成6HB更加稳定,对应的融合抑制剂的抑制活性升高;而有些位点的平均ΔΔ G明显低于总体水平,意味着这些位置上不稳定性突变较多,优化后可能造成6HB不稳定,对应的融合抑制剂的抑制活性下降。根据预测,前者由于适合进行优化,称为“有利位点”;后者不适合优化,称为“不利位点”。在所有位点的计算结果中,选取5个有利位点:E1265、S1268、Q1270、Q1271、K1274。它们的平均ΔΔ G高于整体水平,大多数突变ΔΔ G>0,呈现为稳定性突变,且部分突变ΔΔ G>1,表现出较强的稳定性。同时,选择4个不利位点:L1269、V1272、V1273、L1276。它们绝大多数突变ΔΔ G<0,平均ΔΔ G低于整体水平,表现出明显的不稳定性。针对上述位点,设计4条多肽类融合抑制剂:MEO代表MERS-CoV S蛋白HR2原始序列,F4UH与F5UH分别代表有利位点引入优化突变后的融合抑制剂,而F4UD代表不利位点引入突变后的融合抑制剂(图 2B)。所有位点优化均遵循极性相似且对应ΔΔ G较高的原则,如果该位点上所有突变对应ΔΔ G均<0,则选择对应ΔΔ G最高的突变进行优化。
|
| A: By introducing mutations into 1246-1288 positions in HR2 sequence, a series of ΔΔ G results listed above could be obtained. Positive ΔΔ G is marked in blue and negative ΔΔ G is marked in red. Wild type sequence is marked in blank blocks. B:Four fusion inhibitors were designed according to ΔΔ G results (shown in box-plot). Compatible positions with higher average ΔΔ G are marked in green, and incompatible positions with lower average ΔΔ G are marked in aurantia. 图 2 利用I-Mutant2.0设计针对HR1区段的多肽类融合抑制剂 Fig. 2 I-Mutant2.0 used to design fusion inhibitors against MERS-CoV S protein HR1 region |
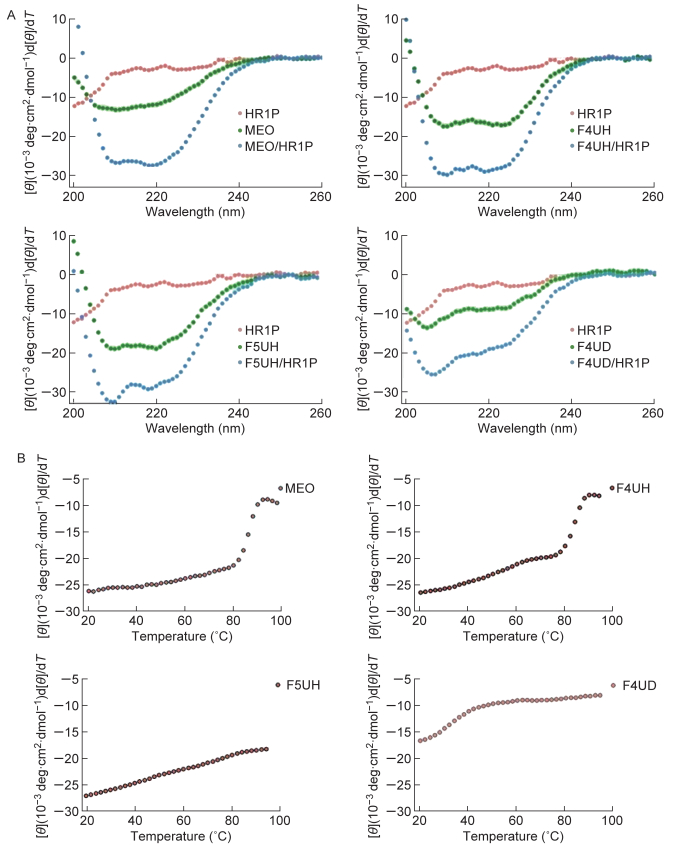
为进一步研究HR1序列与MEO、F4UH、F5UH、F4UD之间的相互作用受突变的影响,本研究采用圆二色谱测量HR1序列多肽HR1P与4条多肽之间相互作用形成的螺旋结构[23]。如图 3A所示,HR1P单独存在于溶液中时,本身不呈现明显的二级结构,曲线中未观察到208 nm与222 nm处明显的负峰值。MEO、F4UH、F5UH与F4UD单独存在于溶液中时,只能形成较少的螺旋结构,208 nm与222 nm处负峰不明显。抑制剂多肽单独存在于溶液中时,222 nm处峰值均未达到-20 000,螺旋度<60%。两者等浓度混合后,HR1P与融合抑制剂相互作用形成6HB,溶液的[θ]明显升高。结果表明,有79%的MEO与HR1P相互作用形成螺旋结构;F4UH、F5UH分别有86%、87%与HR1P结合形成螺旋结构;而F4UD与HR1P结合能力较弱,仅有51%的多肽形成螺旋结构(表 1)。
|
| A: Secondary structure of inhibitors, HR1P and inhibitor/HR1P complex in PBS buffer. CD spectra for inhibitor (10 μmol/L), HR1P (10 μmol/L) and their complex in PBS buffer (pH 7.4) at 20 ℃. The saddle-shaped curve could be observed with double negative peaks at 222 nm and 208 nm. B: CD signal at 222 nm for the inhibitor/HR1P complex as a function of temperature from 20 ℃ to 99 ℃. The thermal melting process could be observed by a rapid leaping of the [θ]222 curve from baseline to around -10. 图 3 突变影响融合抑制剂与HR1序列结合 Fig. 3 Introduced mutations could influence the interaction between fusion inhibitors and HR1 sequence |
| Peptide mixture | Coiling percentage at 20 ℃ | Tm |
| MEO with HR1P | 79% | 87 ℃ |
| F4UH with HR1P | 86% | 87 ℃ |
| F5UH with HR1P | 87% | >99 ℃ |
| F4UD with HR1P | 51% | 35 ℃ |
| The percentage of coiling structure of the inhibitor/HR1P complex presented at 20 ℃ was calculated by the ratio of [θ]222 value to -33 000. Tm value was determined through the climax of the first derivate (d[θ]/dT) against temperature (function not shown). | ||
在此基础上,测量含各种突变的融合抑制剂与HR1P形成复合物的热稳定性。通过测量222 nm处溶液[θ]随温度的变化,绘制复合物热变性曲线(图 3B)。结果显示,MEO、F4UH、F5UH与HR1P相互作用形成的复合物较为稳定。其中,MEO、F4UH与HR1P形成的复合物Tm均位于87 ℃左右,F5UH与HR1P形成的复合物Tm高于99 ℃,而F4UD与HR1P形成的复合物稳定性很低,Tm为35 ℃(表 1)。
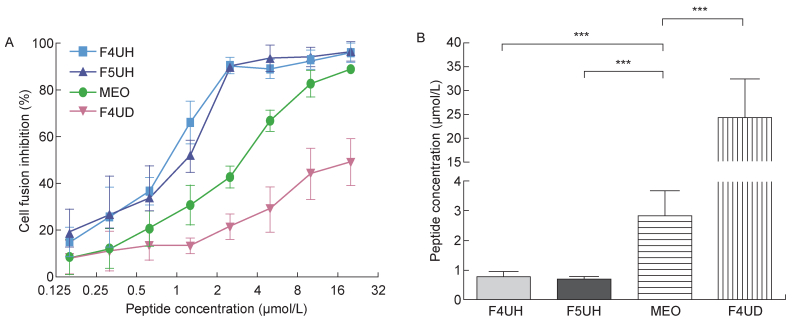
2.3 突变影响融合抑制剂的细胞-细胞融合抑制活性与假病毒抑制活性细胞-细胞融合模型是常用的模拟病毒感染早期过程与评估融合抑制剂抑制病毒包膜蛋白介导的膜融合能力的实验体系[30-31]。本研究利用293T细胞与Huh-7细胞分别表达S蛋白与DPP4蛋白,模拟病毒与宿主细胞早期接触-融合过程。当293T效应细胞与Huh-7靶细胞共同培养3 h后,MEO、F4UH及F5UH均表现出明显的抑制细胞融合的能力,而F4UD则不能有效抑制细胞融合(图 4A)。4条多肽抑制细胞-细胞融合的IC50存在显著性差异:F4UH、F5UH抑制细胞融合的IC50分别为(0.78±0.18) μmol/L、(0.66±0.13) μmol/L,显著低于MEO的(2.79±0.88) μmol/L;而F4UD的IC50为(24.21±8.39) μmol/L,显著高于MEO(图 4B,表 2)。
|
| A:Inhibitory activity of four inhibitors on MERS-CoV S protein-mediated cell-cell fusion as a function of peptide concentration. B: 50% inhibitory concentration was calculated based on median-effect principle. 图 4 突变影响融合抑制剂的细胞-细胞融合抑制活性 Fig. 4 Introduced mutations could influence the inhibition of S protein-mediated cell-cell fusion |
| Peptide | Cell-cell fusion IC50(μmol/L) | Pseudovirus infection IC50(μmol/L) |
| MEO | 2.79±0.88 | 3.31±0.47 |
| F4UH | 0.78±0.18 | 1.71±0.18 |
| F5UH | 0.66±0.13 | 0.73±0.09 |
| F4UD | 24.21±8.39 | 37.35±10.04 |
| The IC50 of cell-cell fusion and pseudovirus infection was calculated based on median-effect principle, according to cell-cell fusion assay and pseudovirus infection assay. | ||
为进一步验证引入突变对融合抑制剂抗病毒活性的影响,本研究采用假病毒感染系统来检测各种突变融合抑制剂的抑制活性。假病毒抑制实验结果与细胞-细胞融合抑制实验相似:F4UH、F5UH与MEO均呈现出较明显的假病毒抑制活性,而F4UD的抑制活性较弱(图 5A)。F4UH、F5UH抑制假病毒的IC50分别为(1.71±0.18) μmol/L、(0.73±0.09) μmol/L,MEO的IC50 为(3.31±0.47) μmol/L,F4UH、F5UH的IC50显著低于MEO;F4UD的IC50为(37.35±10.04) μmol/L,显著高于其他三者(图 5B,表 2)。
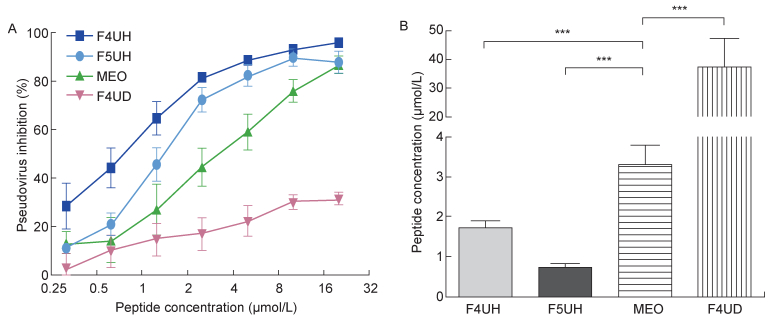
|
| A:Inhibitory activity of four inhibitors on MERS-CoV pseudovirus infection in Huh-7 cells. B: 50% inhibitory concentration was calculated based on median-effect principle. 图 5 突变影响融合抑制剂的假病毒抑制活性 Fig. 5 Introduced mutations could influence the inhibition of pseudovirus infection |
为解决基于抑制6HB形成的多肽类融合抑制剂设计过程中病毒蛋白晶体结构信息缺失对优化设计工作的限制,本研究使用I-Mutant2.0软件作为辅助手段,预测在MERS-CoV S蛋白HR2区段存在若干优化位点,并根据预测结果设计4条多肽。结果表明,与病毒融合抑制剂原始序列MEO相比,在有利位点引入突变,使F4UH、F5UH与HR1P结合的能力提升,其细胞-细胞融合抑制活性与假病毒抑制活性也得到不同程度的提升;而在不利位点引入突变,导致F4UD与HR1P结合能力下降,细胞融合抑制活性、假病毒抑制活性也均下降。这不仅印证了I-Mutant2.0对MERS-CoV S蛋白HR2序列分析和预测的结果,还证明利用I-Mutant2.0可在不依赖病毒蛋白三级结构的情况下更高效、更准确地确定多肽类病毒融合抑制剂中适合优化的位置。
计算机辅助分析手段曾用于设计小分子融合抑制剂和多肽类融合抑制剂[33-37],但传统手段往往依赖对蛋白质分子三级结构详细信息的了解。本研究提供了一种新的思路:对于相关蛋白信息较为缺乏的新发突发传染病,可使用计算机软件作为替代手段,为融合抑制剂的优化设计提供方向,提高研究的目的性与效率。本研究采用的方法是基于蛋白质结构稳定性的原理进行整体分析,并不局限于特定氨基酸之间的相互作用,同样可用于Ⅰ型包膜病毒以外的病毒融合抑制剂研究,在新药开发领域有较好的应用前景。
值得注意的是,I-Mutant2.0基于热力学稳定性做出的预测仅能用于提升融合抑制剂的抑制活性,而融合抑制剂的优化设计往往要追求多种属性的提升,如溶解性、耐储存性、逃逸耐受能力与抗原性等,一味追求抑制活性的提升可能造成融合抑制剂的其他属性受到影响。因此,在实际操作过程中,应谨慎选择所要使用的方法。
| [1] |
White JM, Delos SE, Brecher M, Schornberg K. Structures and mechanisms of viral membrane fusion proteins: multiple variations on a common theme[J]. Crit Rev Biochem Mol Biol, 2008, 43(3): 189-219.
[DOI]
|
| [2] |
Chan DC, Fass D, Berger JM, Kim PS. Core structure of gp41 from the HIV envelope glycoprotein[J]. Cell, 1997, 89(2): 263-273.
[DOI]
|
| [3] |
Colman PM, Lawrence MC. The structural biology of type I viral membrane fusion[J]. Nat Rev Mol Cell Biol, 2003, 4(4): 309-319.
[DOI]
|
| [4] |
Ryser HJ, Flückiger R. Progress in targeting HIV-1 entry[J]. Drug Discov Today, 2005, 10(16): 1085-1094.
[DOI]
|
| [5] |
Buzon V, Natrajan G, Schibli D, Campelo F, Kozlov MM, Weissenhorn W. Crystal structure of HIV-1 gp41 including both fusion peptide and membrane proximal external regions[J]. PLoS Pathog, 2010, 6(5): e1000880.
[DOI]
|
| [6] |
Mao Y, Wang L, Gu C, Herschhorn A, Xiang SH, Haim H, Yang X, Sodroski J. Subunit organization of the membrane-bound HIV-1 envelope glycoprotein trimer[J]. Nat Struct Mol Biol, 2012, 19(9): 893-899.
[DOI]
|
| [7] |
Munro JB, Mothes W. Structure and dynamics of the native HIV-1 Env trimer[J]. J Virol, 2015, 89(11): 5752-5755.
[DOI]
|
| [8] |
Jiang S, Lin K, Strick N, Neurath AR. HIV-1 inhibition by a peptide[J]. Nature, 1993, 365(6442): 113.
[DOI]
|
| [9] |
Jiang S, Lin K, Strick N, Neurath AR. Inhibition of HIV-1 infection by a fusion domain binding peptide from the HIV-1 envelope glycoprotein gp41[J]. Biochem Biophys Res Commun, 1993, 195(2): 533-538.
[DOI]
|
| [10] |
Wild CT, Shugars DC, Greenwell TK, McDanal CB, Matthews TJ. Peptides corresponding to a predictive alpha-helical domain of human immunodeficiency virus type 1 gp41 are potent inhibitors of virus infection[J]. Proc Natl Acad Sci USA, 1994, 91(21): 9770-9774.
[DOI]
|
| [11] |
Nishikawa H, Oishi S, Fujita M, Watanabe K, Tokiwa R, Ohno H, Kodama E, Izumi K, Kajiwara K, Naitoh T, Matsuoka M, Otaka A, Fujii N. Identification of minimal sequence for HIV-1 fusion inhibitors[J]. Bioorg Med Chem, 2008, 16(20): 9184-9187.
[DOI]
|
| [12] |
Zhao L, Hu ZW, Tong P, Chen YX, Zhao YF, Li YM. Addition of artificial salt bridge by Ile646Lys mutation in gp41 coiled-coil domain regulates 6-helical bundle formation[J]. Bioorg Med Chem Lett, 2013, 23(9): 2727-2732.
[DOI]
|
| [13] |
Zhu Y, Su S, Qin L, Wang Q, Shi L, Ma Z, Tang J, Jiang S, Lu L, Ye S, Zhang R. Rational improvement of gp41-targeting HIV-1 fusion inhibitors: an innovatively designed Ile-Asp-Leu tail with alternative conformations[J]. Sci Rep, 2016, 6: 31983.
[DOI]
|
| [14] |
Su S, Zhu Y, Ye S, Qi Q, Xia S, Ma Z, Yu F, Wang Q, Zhang R, Jiang S, Lu L. Creating an artificial tail anchor as a novel strategy to enhance the potency of peptide-based HIV fusion inhibitors[J]. J Virol, 2017, 91(1): e01445-16.
[DOI]
|
| [15] |
Su S, Wang Q, Xu W, Yu F, Hua C, Zhu Y, Jiang S, Lu L. A novel HIV-1 gp41 tripartite model for rational design of HIV-1 fusion inhibitors with improved antiviral activity[J]. AIDS, 2017, 31(7): 885-894.
[DOI]
|
| [16] |
Smith RD. Responding to global infectious disease outbreaks: lessons from SARS on the role of risk perception, communication and management[J]. Soc Sci Med, 2006, 63(12): 3113-3123.
[DOI]
|
| [17] |
Zaki AM, van Boheemen S, Bestebroer TM, Osterhaus AD, Fouchier RA. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia[J]. N Engl J Med, 2012, 367(19): 1814-1820.
[DOI]
|
| [18] |
Baize S, Pannetier D, Oestereich L, Rieger T, Koivogui L, Magassouba N, Soropogui B, Sow MS, Keïta S, De Clerck H, Tiffany A, Dominguez G, Loua M, Traoré A, Kolié M, Malano ER, Heleze E, Bocquin A, Mély S, Raoul H, Caro V, Cadar D, Gabriel M, Pahlmann M, Tappe D, Schmidt-Chanasit J, Impouma B, Diallo AK, Formenty P, Van Herp M, Günther S. Emergence of Zaire Ebola virus disease in Guinea[J]. N Engl J Med, 2014, 371(15): 1418-1425.
[DOI]
|
| [19] |
Capriotti E, Fariselli P, Casadio R. I-Mutant2.0: predicting stability changes upon mutation from the protein sequence or structure[J]. Nucleic Acids Res, 2005, 33: W306-W310.
[DOI]
|
| [20] |
Chen Y, Rajashankar KR, Yang Y, Agnihothram SS, Liu C, Lin YL, Baric RS, Li F. Crystal structure of the receptor-binding domain from newly emerged Middle East respiratory syndrome coronavirus[J]. J Virol, 2013, 87(19): 10777-10783.
[DOI]
|
| [21] |
Du L, Zhao G, Kou Z, Ma C, Sun S, Poon VK, Lu L, Wang L, Debnath AK, Zheng BJ, Zhou Y, Jiang S. Identification of a receptor-binding domain in the S protein of the novel human coronavirus Middle East respiratory syndrome coronavirus as an essential target for vaccine development[J]. J Virol, 2013, 87(17): 9939-9942.
[DOI]
|
| [22] |
Lu G, Hu Y, Wang Q, Qi J, Gao F, Li Y, Zhang Y, Zhang W, Yuan Y, Bao J, Zhang B, Shi Y, Yan J, Gao GF. Molecular basis of binding between novel human coronavirus MERS-CoV and its receptor CD26[J]. Nature, 2013, 500(7461): 227-231.
[DOI]
|
| [23] |
Lu L, Liu Q, Zhu Y, Chan KH, Qin L, Li Y, Wang Q, Chan JF, Du L, Yu F, Ma C, Ye S, Yuen KY, Zhang R, Jiang S. Structure-based discovery of Middle East respiratory syndrome coronavirus fusion inhibitor[J]. Nat Commun, 2014, 5: 3067.
[DOI]
|
| [24] |
Casadio R, Compiani M, Fariselli P, Vivarelli F. Predicting free energy contributions to the conformational stability of folded proteins from the residue sequence with radial basis function networks[J]. Proc Int Conf Intell Syst Mol Biol, 1995, 3: 81-88.
[URI]
|
| [25] |
Gilis D, Rooman M. Predicting protein stability changes upon mutation using database-derived potentials: solvent accessibility determines the importance of local versus non-local interactions along the sequence[J]. J Mol Biol, 1997, 272(2): 276-290.
[DOI]
|
| [26] |
Zhou H, Zhou Y. Distance-scaled, finite ideal-gas reference state improves structure-derived potentials of mean force for structure selection and stability prediction[J]. Protein Sci, 2002, 11(11): 2714-2726.
[URI]
|
| [27] |
Guerois R, Nielsen JE, Serrano L. Predicting changes in the stability of proteins and protein complexes: a study of more than 1 000 mutations[J]. J Mol Biol, 2002, 320(2): 369-387.
[DOI]
|
| [28] |
Lu M, Blacklow SC, Kim PS. A trimeric structural domain of the HIV-1 transmembrane glycoprotein[J]. Nat Struct Biol, 1995, 2(12): 1075-1082.
[DOI]
|
| [29] |
Liu S, Zhao Q, Jiang S. Determination of the HIV-1 gp41 fusogenic core conformation modeled by synthetic peptides: applicable for identification of HIV-1 fusion inhibitors[J]. Peptides, 2003, 24(9): 1303-1313.
[DOI]
|
| [30] |
Lu H, Zhao Q, Xu Z, Jiang S. Automatic quantitation of HIV-1 mediated cell-to-cell fusion with a digital image analysis system (DIAS): application for rapid screening of HIV-1 fusion inhibitors[J]. J Virol Methods, 2003, 107(2): 155-161.
[DOI]
|
| [31] |
Jiang S, Radigan L, Zhang L. A convenient cell fusion assay for rapid screening for HIV entry inhibitors[C/OL]. Progress in Biomedical Optics: Conference on Advances in Nucleic Acid and Protein Analyses, Manipulation, and Sequencing. Bellingham: SPIE-INT SOC Optical Engineering, 2000, 1: 212-219. http://apps.webofknowledge.com/full_record.do?product=UA&search_mode=GeneralSearch&qid=1&SID=6EN1T5jqCGAxUNw3VM2&page=1&doc=1.
|
| [32] |
Zhao G, Du L, Ma C, Li Y, Li L, Poon VK, Wang L, Yu F, Zheng BJ, Jiang S, Zhou Y. A safe and convenient pseudovirus-based inhibition assay to detect neutralizing antibodies and screen for viral entry inhibitors against the novel human coronavirus MERS-CoV[J]. Virol J, 2013, 10: 266.
[DOI]
|
| [33] |
Jiang SB, Debnath AK. Development of HIV entry inhibitors targeted to the coiled-coil regions of gp41[J]. Biochem Biophys Res Commun, 2000, 269(3): 641-646.
[DOI]
|
| [34] |
Martins do Canto AMT, Palace Carvalho AJ, Prates Ramalho JP, Loura LMS. T-20 and T-1249 HIV fusion inhibitors' structure and conformation in solution: a molecular dynamics study[J]. J Pept Sci, 2008, 14(4): 442-447.
[DOI]
|
| [35] |
Martins do Canto AMT, Palace Carvalho AJ, Prates Ramalho JP, Loura LMS. Structure and conformation of HIV fusion inhibitor peptide T-1249 in presence of model membranes: A molecular dynamics study[J]. J Mol Struct (Theochem), 2010, 946(1-3): 119-124.
[DOI]
|
| [36] |
Martins do Canto AMT, Palace Carvalho AJ, Prates Ramalho JP, Loura LMS. Molecular dynamics simulations of T-20 HIV fusion inhibitor interacting with model membranes[J]. Biophys Chem, 2011, 159(2-3): 275-286.
[DOI]
|
| [37] |
Mavioso ICVC, de Andrade VCR, Palace Carvalho AJ, Martins do Canto AMT. Molecular dynamics simulations of T-2410 and T-2429 HIV fusion inhibitors interacting with model membranes: Insight into peptide behavior, structure and dynamics[J]. Biophys Chem, 2017, 228: 69-80.
[DOI]
|
 2019, Vol. 14
2019, Vol. 14


