2. 武汉大学生命科学学院/病毒学国家重点实验室,武汉 430072
2. State Key Laboratory of Virology, College of life science, Wuhan University, Wuhan 43072, China
猪瘟病毒(classical swine fever virus, CSFV)为黄病毒科瘟病毒属成员,是引起猪急性、发热性与接触性传染病——猪瘟的病原体。CSFV基因组为1条长约12.3 kb的单股正链RNA,包括位于两端的非编码区和中间编码多聚蛋白的开放读码框。多聚蛋白经过细胞与病毒的蛋白酶加工后形成4个结构蛋白和8个非结构蛋白。
CSFV 4种结构蛋白分别是核衣壳Core蛋白、包膜蛋白Erns、E1和E2[1]。Core蛋白在基因组编码的多聚蛋白上位于N端蛋白酶Npro与糖蛋白Erns之间。Core蛋白的成熟包括Npro蛋白酶水解多聚蛋白,形成Core蛋白的N端及细胞信号肽酶加工产生Core蛋白的C端[2-4]。通过Core蛋白缺失突变体的CSFV感染性cDNA克隆,研究人员发现Core蛋白的N端与C端在病毒颗粒形成中发挥重要作用[5]。用缺失Core蛋白的CSFV感染性cDNA克隆转染细胞,连续传代后获得的NS3突变以补偿Core蛋白缺失的功能,产生的CSFV毒力降低[6],表明Core蛋白在决定CSFV毒力中有重要作用。研究还发现,对Core蛋白的SUMO化位点或IQGAP1相互作用位点进行突变,可导致CSFV毒力减弱[7-8]。研究表明Core蛋白对病毒的包装及其毒力具有十分重要的作用,但目前关于Core蛋白在细胞中的定位与功能的关系研究报道相对较少。本实验通过研究Core蛋白、截短突变体及氨基酸位点突变体在细胞中的定位,发现Core蛋白的核仁定位序列为PESRKKL,关键氨基酸为R76K77。
1 材料与方法 1.1 材料pEGFP-C1质粒和PK15细胞为本实验室保存,大肠埃希菌(Escherichia coli,E.coli)DH5α感受态细胞为本实验室制备,EcoRⅠ、BamHⅠ限制性内切酶、T4 DNA连接酶、DNA Marker均购自Thermo Fisher公司,质粒提取试剂盒与DNA凝胶回收试剂盒购自Promega公司,LipofectamineTM 2000购自Invitrogen公司,DAPI购自Sigma公司。
1.2 方法 1.2.1 重组质粒pEGFP-Core及Core蛋白突变体的构建根据CSFV石门株基因组序列(NCBI NO.AF092448.2),设计扩增Core基因及其突变体的引物,在引物两端分别引入EcoRⅠ和BamHⅠ酶切位点。以CSFV石门株感染性cDNA克隆为模板,聚合酶链反应(polymerase chain reaction,PCR)扩增Core蛋白基因及其截短突变体。将扩增得到的DNA片段和pEGFP-C1质粒用EcoRⅠ和BamHⅠ双酶切后回收,并用T4连接酶进行连接,获得pEGFP-Core及其截短的重组质粒。利用重叠PCR扩增法获得Core蛋白基因点突变DNA片段,采取上述重组质粒构建方法获得了pEGFP-Core突变体。重组质粒由上海生工生物工程公司进行测序鉴定。
1.2.2 转染提取足量的pEGFP-C1、pEGFP-Core蛋白及其突变体的重组质粒。将培养状态良好的PK15细胞种植于35 mm细胞培养皿,常规培养6 h左右。当细胞汇合度约80%~90%时,用LipofectamineTM 2000进行转染。具体操作:分别配置A液(3 μg质粒、250 μL Opti-MEM培养基)和B液(10 μL LipofectamineTM 2000、250 μL Opti-MEM培养基),室温孵育5分钟后,混匀A液与B液,继续室温孵育20分钟,加到含有细胞和培养液的培养皿中并混匀。
1.2.3 荧光显微镜观察亚细胞定位将重组质粒转染24 h后的PK15细胞用PBS清洗,经甲醇-丙酮固定液(1:1)固定后,DAPI染色。LAS AF Lite 4.0共聚焦荧光显微镜(Leica, Germany)下进行观察。
2 结果 2.1 Core蛋白定位于胞质及核仁为研究CSFV Core蛋白在PK15细胞的定位分布情况,构建重组真核表达质粒pEGFP-Core,转染到PK15细胞后观察Core蛋白与EGFP融合蛋白的表达及细胞定位。结果显示,对照组EGFP均匀分布于整个细胞,而在转染重组质粒pEGFP-Core的PK15细胞的EGFP-Core除胞质定位外还具有明显的核仁定位分布(图 1),这与早期Core蛋白核仁定位的研究结论一致[9]。
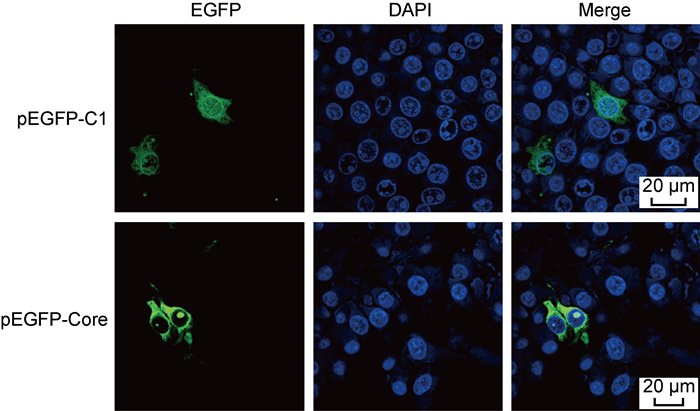
|
| 图 1 CSFV Core蛋白在PK15细胞的定位分析 Fig. 1 Analyze the localization of CSFV Core protein in PK15 cells |
为鉴定Core蛋白核仁定位序列,构建3个pEGFP-Core截短突变体重组质粒(图 2A)。将重组质粒分别转染至PK15细胞,荧光共聚焦显微镜观察。结果显示,EGFP-Core M1与EGFP定位情况类似,表明Core M1不存在定位Core蛋白的特异序列;EGFP-Core M2与EGFP-Core定位情况类似,存在明显的核仁定位,表明Core蛋白的核仁定位序列位于Core蛋白的第36位与第81位氨基酸之间;EGFP-Core M3只定位在胞质内,没有细胞核仁定位(图 2B)。
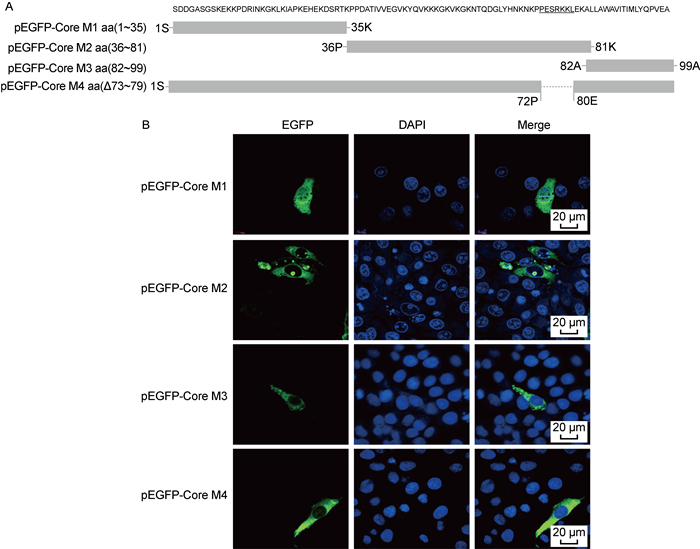
|
| A: Amino acids sequence of CSFV Core and its truncated mutants diagram. B: Immunofluorescence analysis of truncated CSFV Core protein in PK15 cells. 图 2 CSFV Core蛋白、截短突变体的亚细胞定位 Fig. 2 Subcellular localization of truncated CSFV Core mutants |
为进一步确认Core蛋白的核仁定位序列,利用PSORTII计算机程序(https://psort.hgc.jp/form2.html)对Core M2进行核仁定位序列分析。分析结果显示,73~79位氨基酸序列(PESRKKL)可能是潜在的核仁定位序列。为了验证PESRKKL序列是否是Core蛋白核仁定位的关键序列,构建了pEGFP-Core M4(Δ73~79)重组质粒,并将其转染到PK15细胞。荧光显微镜显示,EGFP-Core M4(Δ73~79)没有核仁定位(图 2B),表明PESRKKL序列是CSFV Core蛋白核仁定位的关键序列。
2.3 Core蛋白核仁定位的关键氨基酸为R76K77为进一步定位Core蛋白核仁定位序列的关键氨基酸,将Core蛋白核仁定位序列的氨基酸依次突变为丙氨酸(图 3A和图 3B)。结果发现,PESRKKL序列的单个氨基酸突变并不影响Core蛋白的核仁定位(图 3C)。
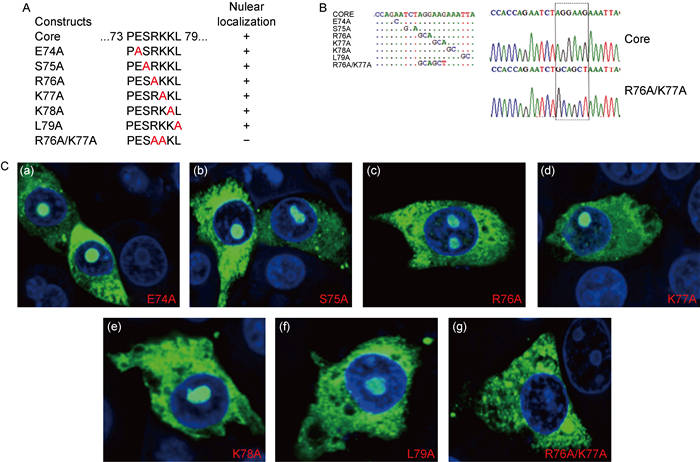
|
| A: Core mutants with indicated amino acids mutation and its localization in PK15 cells. B: The alignment of sequencing results of Core mutants. C: Immunofluorescence analysis of Core mutants localization in PK15 cells. 图 3 CSFV Core蛋白核仁定位的关键氨基酸鉴定 Fig. 3 Identification of critical amino acids for nucleolus localization of CSFV Core |
根据黄病毒科其他病毒Core蛋白的核仁定位序列研究,推测PESRKKL序列的连续2个碱性氨基酸序列可能是该序列核仁定位的关键氨基酸。因此,构建了EGFP-Core的突变体R76A/K77A(图 3A和图 3B)。观察发现,突变体R76A/K77A不存在核仁定位(图 3C)。以上结果表明R76K77是决定Core蛋白核仁定位的关键氨基酸。
3 讨论本研究发现CSFV Core蛋白的核仁定位序列位于73PESRKKL79。已有研究报道,CSFV Core蛋白的一个核仁定位序列位于51KKKGKV56[9]。因此, 我们推测CSFV Core蛋白至少具有两个核仁定位序列,缺失其中任何一个核仁定位序列都将导致Core蛋白的核仁定位功能丧失。
已有的研究表明,黄病毒科成员核心蛋白具有一个或多个核仁定位序列,对这些核仁定位序列进行的突变都严重影响病毒的包装与复制。例如,乙型脑炎病毒Core蛋白的G42P43[10],登革病毒Core蛋白的K73K74A和R85K86[11]以及丙型肝炎病毒Core蛋白第5~13、38~43和112~117位氨基酸[12]都是决定Core蛋白核仁定位的关键氨基酸。那么CSFV Core蛋白的核仁定位对病毒复制具有怎样的意义?有文献报道,缺失Core蛋白第51~57位或第71~78位氨基酸分别导致转染感染性克隆24 h后病毒滴度降低87.5%和93.75%[5]。可见,CSFV Core的核仁定位对病毒颗粒具有一定的影响。而Core蛋白核仁定位序列的缺失对病毒基因组的复制与病毒毒力的影响尚未可知,有待进一步的研究。
此外,研究发现,第82~99位氨基酸序列具有较强的疏水性。研究结果显示,EGFP-Core M3第82~99位氨基酸定位在胞质而没有细胞核仁定位,提示Core蛋白第82~99位氨基酸序列定位于细胞质而发挥作用。在黄病毒科的其他病毒也有类似报道,如丙型肝炎病毒Core蛋白的疏水结构域第109~133位氨基酸是Core蛋白从细胞核转运到细胞质的重要出核信号序列,突变其中的疏水氨基酸都将导致Core蛋白的核仁定位减少[13];登革病毒Core蛋白通过其内部疏水序列第46~66位氨基酸定位到内质网膜上,使得病毒颗粒与内质网膜相关联[14]。登革病毒Core蛋白的内部疏水序列将病毒颗粒牵引到内质网膜形成的脂滴上,促进病毒基因组的复制,从而影响登革病毒的复制[15]。位于CSFV Core蛋白第82~99位氨基酸促进Core蛋白的核仁定位对病毒复制与包装具有怎样的功能还有待进一步研究。
综上所述,本研究通过分析Core蛋白、截短突变体及氨基酸位点突变体与EGFP融合蛋白在PK15细胞中的亚细胞定位,发现了CSFV Core蛋白核仁定位序列和关键氨基酸位点,为进一步研究CSFV Core蛋白结构与功能及其在病毒复制和组装中的作用提供了依据。
| [1] |
Thiel HJ, Stark R, Weiland E, Rümenapf T, Meyers G. Hog cholera virus: molecular composition of virions from a pestivirus[J]. J Virol, 1991, 65(9): 4705-4712.
[URI]
|
| [2] |
Rümenapf T, Stark R, Heimann M, Thiel HJ. N-terminal protease of pestiviruses: identification of putative catalytic residues by site-directed mutagenesis[J]. J Virol, 1998, 72(3): 2544-2547.
[URI]
|
| [3] |
Rümenapf T, Unger G, Strauss JH, Thiel HJ. Processing of the envelope glycoproteins of pestiviruses[J]. J Virol, 1993, 67(6): 3288-3294.
[URI]
|
| [4] |
Stark R, Meyers G, Rümenapf T, Thiel HJ. Processing of pestivirus polyprotein: cleavage site between autoprotease and nucleocapsid protein of classical swine fever virus[J]. J Virol, 1993, 67(12): 7088-7095.
[URI]
|
| [5] |
Riedel C, Lamp B, Heimann M, Rümenapf T. Characterization of essential domains and plasticity of the classical Swine Fever virus Core protein[J]. J Virol, 2010, 84(21): 11523-11531.
[DOI]
|
| [6] |
Riedel C, Lamp B, Heimann M, König M, Blome S, Moennig V, Schüttler C, Thiel HJ, Rümenapf T. The core protein of classical Swine Fever virus is dispensable for virus propagation in vitro[J]. PLoS Pathog, 2012, 8(3): e1002598.
[DOI]
|
| [7] |
Gladue DP, Holinka LG, Fernandez-Sainz IJ, Prarat MV, O'Donnell V, Vepkhvadze NG, Lu Z, Risatti GR, Borca MV. Interaction between Core protein of classical swine fever virus with cellular IQGAP1 protein appears essential for virulence in swine[J]. Virology, 2011, 412(1): 68-74.
[DOI]
|
| [8] |
Gladue DP, Holinka LG, Fernandez-Sainz IJ, Prarat MV, O'Donell V, Vepkhvadze N, Lu Z, Rogers K, Risatti GR, Borca MV. Effects of the interactions of classical swine fever virus Core protein with proteins of the SUMOylation pathway on virulence in swine[J]. Virology, 2010, 407(1): 129-136.
[DOI]
|
| [9] |
张鑫, 刘宁, 时洪艳, 徐佳. 猪瘟病毒衣壳蛋白核仁定位信号鉴定[J]. 中国预防兽医学报, 2015, 37(2): 93-96. [DOI]
|
| [10] |
Mori Y, Okabayashi T, Yamashita T, Zhao Z, Wakita T, Yasui K, Hasebe F, Tadano M, Konishi E, Moriishi K, Matsuura Y. Nuclear localization of Japanese encephalitis virus core protein enhances viral replication[J]. J Virol, 2005, 79(6): 3448-3458.
[DOI]
|
| [11] |
Sangiambut S, Keelapang P, Aaskov J, Puttikhunt C, Kasinrerk W, Malasit P, Sittisombut N. Multiple regions in dengue virus capsid protein contribute to nuclear localization during virus infection[J]. J Gen Virol, 2008, 89(Pt 5): 1254-1264.
[URI]
|
| [12] |
Levin A, Neufeldt CJ, Pang D, Wilson K, Loewen-Dobler D, Joyce MA, Wozniak RW, Tyrrell DL. Functional characterization of nuclear localization and export signals in hepatitis C virus proteins and their role in the membranous web[J]. PLoS One, 2014, 9(12): e114629.
[DOI]
|
| [13] |
Cerutti A, Maillard P, Minisini R, Vidalain PO, Roohvand F, Pecheur EI, Pirisi M, Budkowska A. Identification of a functional, CRM-1-dependent nuclear export signal in hepatitis C virus core protein[J]. PLoS One, 2011, 6(10): e25854.
[DOI]
|
| [14] |
Markoff L, Falgout B, Chang A. A conserved internal hydrophobic domain mediates the stable membrane integration of the dengue virus capsid protein[J]. Virology, 1997, 233(1): 105-117.
[DOI]
|
| [15] |
Samsa MM, Mondotte JA, Iglesias NG, Assunção-Miranda I, Barbosa-Lima G, Da Poian AT, Bozza PT, Gamarnik AV. Dengue virus capsid protein usurps lipid droplets for viral particle formation[J]. PLoS Pathog, 2009, 5(10): e1000632.
[DOI]
|
 2019, Vol. 14
2019, Vol. 14


