2. 中国疾病预防控制中心病毒病预防控制所传染病预防控制国家重点实验室, 北京 102206
2. State Key Laboratory of Infectious Disease Prevention and Control, Collaborative Innovation Center for Diagnosis and Treatment of Infectious Diseases, National Institute for Viral Disease Control and Prevention, Chinese Center for Disease Control and Prevention, Beijing 102206, China
柯萨奇病毒(Coxsackie virus, CV)是微小RNA病毒科肠道病毒属的单股正链RNA病毒,可分为A(Coxsackie virus groups A, CVA)和B(Coxsackie virus groups B, CVB)两组。CVA分为23个型别,CVA1~23;CVB分为6个型别,CVB1~6。其中,柯萨奇病毒B3型(Coxsackie virus B3, CVB3)是引起人类病毒性心肌炎的重要病原体之一[1]。
目前,已有确凿证据表明,病毒来源的小RNA (viral small RNA,vsRNA)存在于哺乳动物。在微小RNA病毒科病毒感染的宿主细胞中发现了2种vsRNA,1种是病毒来源的小干扰RNA (virus-derived small interfering RNA,vsiRNA),它是在RNA复制过程中由病毒双链RNA中间体加工而成;另1种来自高度碱基配对的单链基因组区域的小RNA,例如微小RNA病毒的内部核糖体进入位点序列(internal ribosome entry site, IRES)。微小RNA病毒科病毒,如脑心肌炎病毒、肠道病毒A71 (EV-A71)和脊髓灰质炎病毒(poliovirus)等,均已被证实可诱导哺乳动物细胞产生病毒衍生小干扰RNA(small interfering RNA,siRNA),即vsiRNA[2-5]。
病毒基因在转录过程中产生一些双链RNA(double-stranded RNA, dsRNA)[6]。而宿主细胞质中的RNA内切酶Dicer可迅速针对dsRNA发生反应,将其切割成多个特定小片段RNA(21~23 bp),此为外源性siRNA(exogenous siRNA,exo-siRNA)[7-8]。siRNA也来源于细胞基因组产生的信使RNA(messenger RNA,mRNA)转录物、假基因衍生的反义转录物的双链体和发夹RNA(hairpin RNA,hpRNA)[7, 9]。
siRNA与Argonaute(AGO)蛋白家族相互作用,通过RNA干扰(RNA interference,RNAi)过程控制病毒RNA的复制。siRNA在RNA解旋酶的作用下解链成正义链和反义链,继之反义链与解旋酶、内外切酶等结合,形成RNA诱导的沉默复合物(RNA-induced silencing complex,RISC)[6]。RISC与外源基因表达的mRNA同源区进行特异性结合。由于RISC具有核酸酶的功能,可在与siRNA反义链互补结合的两端切割mRNA,使其降解。此外,siRNA可作为引物与靶RNA结合并在RNA聚合酶(RNA-dependent RNA polymerase, RdRP)作用下合成更多新的dsRNA,新合成的dsRNA再由Dicer切割产生大量的次级siRNA,从而使RNAi的作用进一步放大,最终将靶mRNA完全降解[6, 9]。siRNA在基因转录后修饰、抗病毒感染等方面发挥了重要作用[7, 10-12]。本研究通过小RNA高通量测序,了解CVB3感染是否诱导内源性siRNA的产生,期望为进一步研究CVB3感染对siRNA表达的影响及其作用机制以及是否通过siRNA影响病毒复制等奠定基础。
1 材料和方法 1.1 材料HeLa细胞和CVB3由本实验室保存,细胞培养液和胎牛血清购自GIBCO公司。
1.2 方法 1.2.1 CVB3感染HeLa细胞HeLa细胞以6×106/孔传至6孔板,过夜培养至细胞密度为90%。CVB3(MOI=5)接种HeLa细胞,细胞在培养箱(5% CO2、37 ℃)孵育1 h。随后加入含2%血清的细胞维持液,继续培养3 h和6 h后收集细胞。
1.2.2 高通量测序(二代测序)使用试剂盒(QIAamp RNA kit,德国QIAGEN公司)提取细胞RNA,反转录(Superscript Ⅲ First-Strand Synthesis System,美国Invitrogen公司)合成cDNA。取通过质控检测的样本进行下游的文库构建。cDNA经纯化后生成Illumina’s Cluster Station上的Cluster,即可加入测序仪(HiSeq 2000,Illumina公司)进行测序。测序过程中使用Illumina’s sequencing control studio software version 2.8软件,实时分析测序图片,提取原始数据用于基本数据分析(Illumina’s real-time analysis version 1.8.70)。
1.2.3 测序结果分析过滤数据,去除插入片段过长的序列、低质量序列、poly A序列和小片段序列,得到clean tags。将clean tags和已知的小RNA数据库包括miRBase[13]、Rfam[14]、siRNA、piRNA、snoRNA等进行比对。除Rfam使用cmsearch外[15],其余都用Bowtie2软件[16]对clean tag和小RNA数据库进行比对。通过与已知的小RNA数据库比对,鉴定出已知的siRNA。
统计siRNA的种类及数量,并对其做长度分布统计。使用每千个碱基的转录每百万映射读取的转录本数(transcripts per kilobase of exonmodel per million mapped reads,TPM)标准化小RNA的表达方式,参照Audic等[17]的差异基因检测法,筛选两样本的差异表达基因。对差异检验的P值进行多重假设检验校正,通过控制错误发现率(false discovery rate,FDR)决定P值的域值[18]。
1.2.4 反转录-聚合酶链反应使用茎-环结构设计novel_sir3502和novel_sir2806的反转录-聚合酶链反应(reverse transcription-polymerase chain reaction,RT-PCR),引物为5 ′ -CTCAACTGGT-GTCGTGGAGTCGGCAATTCAGTTGAG-3 ′,使用反向引物进行反转录。内参:U6 F CTCGCTT-CGGCAGCACA; U6 R AACGCTTCACGAAT-TTGCGT。体系为5×SYBR Green qPCR Mix 4 μL、20×miRNA Primer F 1 μL、20×miRNA Primer R 1 μL、cDNA模板1 μL,ddH2O加至20 μL。采用两步法扩增:95 ℃ 15 min;95 ℃ 5 s、60 ℃ 30 s, 35个循环。通过2- ΔΔ CT值计算病毒感染细胞与正常细胞之间siRNA表达量的变化。
2 结果 2.1 CVB3感染HeLa细胞后siRNA的总体出现差异为了明确CVB3感染HeLa细胞3 h和6 h后,细胞中siRNA种类和表达水平的总体差异,我们分析比较了样本的公共序列和特有序列。结果显示,与正常培养3 h和6 h的对照细胞相比,CVB3感染HeLa细胞3 h和6 h后,siRNA增加数目分别为1 846、1 154,降低数目分别为831和1 009,表明siRNA在CVB3感染HeLa细胞中发挥了重要作用。病毒感染HeLa细胞6 h相对感染3 h的siRNA增加数目为978,降低数目为1 662。表明CVB3感染后,细胞中各种siRNA表达量随时间发生改变。
2.2 CVB3感染引起HeLa细胞中siRNA的改变采用Bowtie比对软件识别出已知的siRNA,滤除其中与已知高度同源的序列,以UNAFold二级结构折叠预测全新的siRNA序列。同时对siRNA的表达量进行归一化处理,去掉在两样本归一化后表达量log2 ratio < 1的siRNA。筛选与CVB3感染相关的siRNA,对测序样本中全新siRNA的表达进行差异分析。相对正常细胞,病毒感染3 h、6 h后,HeLa细胞中发生变化的siRNA分别为14种和5种。
与正常细胞相比,病毒感染3 h的HeLa细胞中siRNA表达有变化的为novel_sir4680、novel_sir1648、novel_sir3502、novel_sir2806、novel_sir1897、novel_sir509、novel_sir1383、novel_sir1962、novel_sir2353、novel_sir4290、novel_sir2764、novel_sir3623、novel_sir1973和novel_sir2914,其中novel_sir2914表达下调,其余均升高(表 1)。
| siRNA id | Log2 ratio | Regulation |
| novel_sir4680 | 5.08746284 | Up |
| novel_sir1648 | 4.4429435 | Up |
| novel_sir3502 | 4.04439412 | Up |
| novel_sir2806 | 3.9248125 | Up |
| novel_sir1897 | 3.87651695 | Up |
| novel_sir509 | 3.25738784 | Up |
| novel_sir1383 | 3.24792751 | Up |
| novel_sir1962 | 2.67807191 | Up |
| novel_sir2353 | 2.34146828 | Up |
| novel_sir4290 | 1.66337539 | Up |
| novel_sir2764 | 1.62803122 | Up |
| novel_sir3623 | 1.43888424 | Up |
| novel_sir1973 | 1.16528461 | Up |
| novel_sir2914 | -1.9558002 | Down |
| Log2 ratio:CVB3-HeLa_3h_RNA/HeLa RNA. | ||
与正常细胞相比,病毒感染6 h后HeLa细胞中siRNA表达发生变化的有novel_sir1399、novel_sir3502、novel_sir2806、novel_sir2627、novel_sir3115,其中novel_sir3115表达下调,其余均升高(表 2)。
| siRNA id | Log2 ratio | Regulation |
| novel_sir1399 | 3.594946589 | Up |
| novel_sir3502 | 3.448460501 | Up |
| novel_sir2806 | 3.426264755 | Up |
| novel_sir2627 | 2.906890596 | Up |
| novel_sir3125 | -1.825426495 | Down |
| Log2 ratio:CVB3-HeLa_6h_RNA/HeLa RNA. | ||
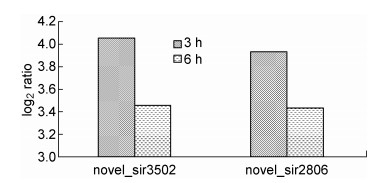
相对正常细胞,高通量测序结果显示,在病毒感染3 h和6 h的HeLa细胞中novel_sir3502和novel_sir2806均发生了改变(图 1)。
|
| 图 1 CVB3感染3 h和6 h后novel_sir3502、novel_sir2806的表达水平 Fig. 1 The expression of novel_sir3502, novel_sir2806 after 3 h, 6 h of CVB3 infection |
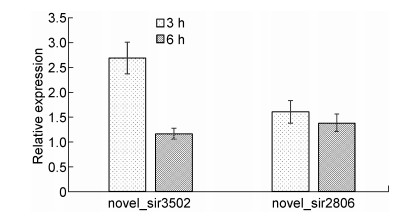
为了进一步验证高通量测序结果,我们对novel_sir3502和novel_sir2806两种siRNA通过茎-环结构设计引物进行验证,荧光定量RT-PCR结果发现CVB3感染HeLa细胞的确引起了两种siRNA的升高,相对于正常HeLa细胞,CVB3感染HeLa细胞3 h和6 h后,novel_sir3502和novel_sir2806表达量的增长倍数分别为1.6,1.52;1.77和1.65,均高于1.5倍(图 2),虽然升高量稍微低于测序结果。结果表明CVB3感染HeLa细胞诱导两种新siRNA的升高。
|
| n=3. 图 2 CVB3感染后3 h和6 h细胞中novel_sir3502、novel_sir2806相对表达量 Fig. 2 The relative expression of novel_sir3502, novel_sir2806 after 3 h, 6 h of CVB3 infection |
为了解novel_sir3502和novel_sir2806调控细胞作用,将novel_sir3502和novel_sir2806通过美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)进行序列比对,发现这两种siRNA与核糖体RNA序列均有同源序列,均识别45S核糖体前体RNA N1、N3、N4、N5和28S核糖体前体RNA N1、N2、N3、N4、N5。(图 3、图 4)。因此,CVB3感染诱导的两种新siRNA可能在结合核糖体RNA后干扰核糖体成熟。

|
| 图 3 novel_sir3502通过NCBI进行的序列比对 Fig. 3 The sequence alignment of novel_sir3502 using BLAST |

|
| 图 4 novel_sir2806通过NCBI进行的序列比对 Fig. 4 The sequence alignment of novel_sir2806 using BLAST |
内源性siRNA在基因调控通路中发挥关键作用,它们来源于双链固有转录物,引导AGO效应蛋白互补性靶向核酸[19-20]。Dicer属于核糖核酸酶Ⅲ(RNase Ⅲ)家族,能够裂解病毒的dsRNA分子,产生20~24 nt长的vsiRNA分子。随后,vsiRNA被RISC中AGO家族蛋白识别。在RISC中,只有1条被称为导向链的保留下来,以针对病毒基因组中的互补序列。目标序列与siRNA完美互补,导致病毒基因组由AGO切割。在秀丽隐杆线虫中,内源性siRNA参与基因组保护和基因调控[21]。果蝇黑腹内siRNA在特定的Dicer和Argonaute蛋白家族成员帮助下产生,在转座子控制和病毒感染保护中起重要作用[22]。脊椎动物内siRNA的生物学功能包括抑制转座因子、阻止染色质组成及在转录和转录后水平调控基因[6, 19, 23]。
与其他小RNA相比,对内源性siRNA的研究还不够深入。内源性siRNA来源于由Dicer切割处理的完全互补的内源性RNA前体[24]。由此产生的siRNA与一种特定的前体蛋白结合,最终确定了复合物的生物学影响。内源性siRNA的合成机制以及生物学作用显示出物种特异性差异。内源性siRNA可抑制转录后的基因表达,影响特定基因位点的转录和染色体结构。因此,内源性siRNA通过在转录和转录后水平调控靶基因表达,在许多生物学过程中发挥重要作用,如保护外来病原体、抑制转座因子和染色质组成[19, 25]。
目前,对于siRNA调控基因表达的研究还在不断摸索中,其调控模型也在不断完善。同时,小分子RNA的研究也存在很多问题,虽然小分子RNA的种类在增加,但其功能并不是十分清楚。RNA干扰技术已被迅速而广泛地应用到基因功能、基因表达调控等热门研究领域,但在应用方面仍存在着许多疑难问题[26-27]。
本研究发现CVB3感染细胞后,siRNA的表达出现变化。将持续表达的novel_sir3502、novel_sir2806与核糖体前体RNA进行同源序列比对分析,结果显示存在互补位点,可以识别45S核糖体前体RNA N1、N3、N4、N5和28S核糖体前体RNA N1、N2、N3、N4、N5。真核细胞核糖体的大小亚基是在核中形成的,在核仁部位rDNA经RNA聚合酶Ⅰ转录出45S rRNA(纤维部的纤维状物质),是rRNA的前体分子,与胞质运来的蛋白质结合,形成RNP复合体。45S rRNA甲基化后,经RNA酶裂解为2个分子:18S rRNA和32S rRNA;后者再裂解为28S rRNA和5.8S rRNA。成熟的rRNA仅为45S rRNA的一半,因此,这两种内源性siRNA可能对于HeLa细胞的核糖体成熟具有一定的作用,提示CVB3感染可能干扰核糖体成熟。本文对siRNA的研究,时间周期较短,未对CVB3感染后siRNA发生变化的机制及对细胞或病毒复制的影响做进一步的探讨研究。在后续实验中我们将采用MOI=1的病毒感染细胞,观察感染6 h、12 h等多个时间点的siRNA变化,探讨siRNA随着感染时间不同而产生的变化,并进一步研究CVB3感染对siRNA表达的影响及其作用机制和是否通过siRNA影响病毒复制等。
| [1] |
Garmaroudi F S, Marchant D, Hendry R, Luo H, Yang D, Ye X, Shi J, McManus B M. Coxsackievirus B3 replication and pathogenesis[J]. Future Microbiol, 2015, 10(4): 629-53.
[DOI]
|
| [2] |
Qiu Y, Xu Y, Zhang Y, Zhou H, Deng YQ, Li XF, Miao M, Zhang Q, Zhong B, Hu Y, Zhang FC, Wu L, Qin CF, Zhou X. Human virus-derived small RNAs can confer antiviral immunity in mammals[J]. Immunity, 2018, 49(4): 780-781.
[DOI]
|
| [3] |
Weng KF, Hung CT, Hsieh PT, Li ML, Chen GW, Kung YA, Huang PN, Kuo RL, Chen LL, Lin JY, Wang RY, Chen SJ, Tang P, Horng JT, Huang HI, Wang JR, Ojcius DM, Brewer G, Shih SR. A cytoplasmic RNA virus generates functional viral small RNAs and regulates viral IRES activity in mammalian cells[J]. Nucleic Acids Res, 2014, 42(20): 12789-12805.
[DOI]
|
| [4] |
Parameswaran P, Sklan E, Wilkins C, Burgon T, Samuel MA, Lu R, Ansel KM, Heissmeyer V, Einav S, Jackson W, Doukas T, Paranjape S, Polacek C, dos Santos FB, Jalili R, Babrzadeh F, Gharizadeh B, Grimm D, Kay M, Koike S, Sarnow P, Ronaghi M, Ding SW, Harris E, Chow M, Diamond MS, Kirkegaard K, Glenn JS, Fire AZ. Six RNA viruses and forty-one hosts:viral small RNAs and modulation of small RNA repertoires in vertebrate and invertebrate systems[J]. PLoS Pathog, 2010, 6(2): e1000764.
[DOI]
|
| [5] |
Maillard PV, van der Veen AG, Poirier EZ, Reis E Sousa C. Slicing and dicing viruses:antiviral RNA interference in mammals[J]. EMBO J, 2019, 38(8): pii:e100941.
[URI]
|
| [6] |
Carthew RW, Sontheimer E. Origins and mechanisms of miRNAs and siRNAs[J]. Cell, 2009, 136(47): 642-655.
[URI]
|
| [7] |
Ghildiyal M, Seitz H, Horwich MD, Li C, Du T, Lee S, Xu J, Kittler EL, Zapp ML, Weng Z, Zamore PD. Endogenous siRNAs derived from transposons and mRNAs in drosophila somatic cells[J]. Science, 2008, 320: 1077-1081.
[DOI]
|
| [8] |
Lee KM, Gong YN, Shih SR. Methods for detection and study of virus-derived small RNAs produced from the intramolecular base-pairing region of the picornavirus genome[J/OL]. Methods, 2019. http://doi.org/10.1016/j.yneth.2019.08.11. [URI]
|
| [9] |
Golden DE, Gerbasi VR, Sontheimer EJ. An Inside Job for siRNAs[J]. Mol Cell, 2008, 31(3): 309-312.
[DOI]
|
| [10] |
Guo Z, Li Y, Ding SW. Small RNA-based antimicrobial immunity[J]. Nat Rev, Immunol, 2019, 19(1): 31-44.
[DOI]
|
| [11] |
Tassetto M, Kunitomi M, Andino R. Circulating immune cells mediate a systemic RNAi-based adaptive antiviral response in Drosophila[J]. Cell, 2017, 169(2): 314-325.
[DOI]
|
| [12] |
Poirier EZ, Goic B, Tomé-Poderti L, Frangeul L, Boussier J, Gausson V, Blanc H, Vallet T, Loyd H, Levi L, Lanciano S, Baron C, Merkling SH, Lambrechts L, Mirouze M, Carpenter S, Vignuzzi M, Saleh MC. Dicer-2-dependent generation of viral DNA from defective genomes of RNA viruses modulates antiviral immunity in insects[J]. Cell Host Microbe, 2018, 23(3): 353-365.
[DOI]
|
| [13] |
Kozomara A, Griffiths-Jones S. miRBase:annotating high confidence microRNAs using deep sequencing data[J]. Nucleic Acids Res, 2014, 42(Database issue): D68-D73.
[URI]
|
| [14] |
Nawrocki EP, Burge SW, Bateman A, Daub J, Eberhardt RY, Eddy SR, Floden EW, Gardner PP, Jones TA, Tate J, Finn RD. Rfam 12.0:updates to the RNA families database[J]. Nucleic Acids Res, 2015, 43(Database issue): D130-D137.
[URI]
|
| [15] |
Nawrocki EP, Eddy SR. Infernal 1.1:100-fold faster RNA homology searches[J]. Bioinformatics, 2013, 29(22): 2933-2935.
[DOI]
|
| [16] |
Külahoglu C, Bräutigam A. Quantitative transcriptome analysis using RNA-seq[J]. Methods Mol Biol, 2014, 1158: 71-91.
[URI]
|
| [17] |
Audic S, Claverie JM. The Significance of digital gene expression profiles[J]. Genome Res, 1997, 7(10): 986-995.
[DOI]
|
| [18] |
Peña EA, Habiger JD, Wu W. Power-enhanced multiple decision functions controlling family-wise error and false discovery rate[J]. Ann Stat, 2011, 39(1): 556-583.
[URI]
|
| [19] |
Piatek MJ, Werner A. Endogenous siRNAs:regulators of internal affairs[J]. Biochem Soc Trans, 2014, 42(4): 1174-1179.
[URI]
|
| [20] |
Werner A, Cockell S, Falconer J, Carlile M, Alnumeir S, Robinson J. Contribution of natural antisense transcription to an endogenous siRNA signature in human cells[J]. BMC Genomics, 2014, 15: 19.
[DOI]
|
| [21] |
Hoogstrate SW, Volkers RJ, Sterken MG, Kammenga JE, Snoek LB. Nematode endogenous small RNA pathways[J]. Worm, 2014, 3: e28234.
[DOI]
|
| [22] |
Elmer K, Helfer S, Mirkovic-Hösle M, Förstemann K. Analysis of endo-siRNAs in Drosophila[J]. Methods Mol Biol, 2014, 1173: 33-49.
[URI]
|
| [23] |
Claycomb JM. Ancient endo-siRNA pathways reveal new tricks[J]. Curr Biol, 2014, 24(15): R703-715.
[DOI]
|
| [24] |
Song MS, Rossi JJ. Molecular mechanisms of Dicer:endonuclease and enzymatic activity[J]. Biochem J, 2017, 474(10): 1603-1618.
[DOI]
|
| [25] |
Giri BR, Ye J, Chen Y, Wei C, Cheng G. In silico analysis of endogenous siRNAs associated transposable elements and NATs in Schistosoma japonicum reveals their putative roles during reproductive development[J]. Parasitol Res, 2018, 117(5): 1549-1558.
[DOI]
|
| [26] |
Ipsaro JJ, Joshua-Tor L. From guide to target:molecular insights into eukaryotic RNA-interference machinery[J]. Nat Struct Mol Biol, 2015, 22(1): 20-28.
[DOI]
|
| [27] |
Sarett SM, Werfel TA, Lee L, Jackson MA, Kilchrist KV, Brantley-Sieders D, Duvall CL. Lipophilic siRNA targets albumin in situ and promotes bioavailability, tumor penetration, and carrier-free gene silencing[J]. Proc Natl Acad Sci U S A, 2017, 114(32): E6490-E6497.
[DOI]
|
 2020, Vol. 15
2020, Vol. 15


