2. 绍兴市人民医院临床检验中心, 绍兴 312000
2. Clinical Laboratory Center, Shaoxing People's Hospital, Shaoxing 312000, Zhejiang Province, China
Hfq(host factor for RNA phage Q β replicase)蛋白首先在大肠埃希菌中被发现,普遍存在于细菌中,是一个伴侣蛋白,属于Sm/Lsm家族成员[1]。Hfq蛋白为同源六聚体结构,通过促进sRNA与mRNA结合而发挥作用[2]。当其存在时,sRNA不仅可与mRNA的5 ′ UTR及编码区结合,还可与3 ′ UTR结合。一条sRNA通过不同位点可与不同的mRNA结合,广泛参与转录后调控[3]。
Hfq蛋白参与的sRNA调节途径被认为是细菌耐药的重要机制之一,它也可以调节菌株毒力,可能是潜在的药物治疗靶点[4-5]。在铜绿假单胞菌中,缺乏Hfq会导致多黏菌素耐药[6]。Hfq蛋白还与气单胞菌持留菌有关[7]。另有研究发现,致病性大肠埃希菌的MgrR、RyhB以依赖Hfq蛋白的方式调节grlRA,从而调节其毒力[8]。
肺炎克雷伯菌(Klebsiella pneumoniae,KP)是临床常见感染的病原体,中国细菌耐药监测网(China Antimicrobial Surveillance Network, CHINET)监测提示其检出率及耐药率均逐年升高,尤其是碳青霉烯类耐药株检出率明显上升,给临床治疗带来极大困难[9],甚至出现了耐多黏菌素KP菌株的局部暴发[10]。Hfq蛋白在KP中的功能相关研究较为少见,对KP耐药、毒力的影响及其机制尚未明了。
本研究将我院在2017年8月―2018年8月收集的KP与其他常见菌株的Hfq蛋白进行比较分析,并敲除1株KP菌的hfq基因用于后续研究,期望对KP感染的临床诊治提供参考。
1 材料与方法 1.1 材料 1.1.1 菌株59株KP自2017年8月―2018年8月在我院就诊的临床病例中分离。11株临床常见菌株hfq基因从NCBI基因库中下载(https://www.ncbi.nlm.nih.gov),分别是铜绿假单胞菌PAO1(Pseudomonas aeruginosa,AAG08329.1)、沙门菌LT2(Salmonella enterica,NP_463222.1)、霍乱弧菌N16961(Vibrio cholerae,NP_230001.1)、副溶血性弧菌RIMD 2210633(Vibrio parahaemolyticus,NP_799196.1)、KP HS11286(KP,NC_016845.1)、洋葱伯克霍尔德菌ATCC 25416(Burkholderia cepacia,NZ_CP012981.1)、痢疾志贺菌Sd197(Shigella dysenteriae, NC_007606.1)、大肠埃希菌MG1655(Escherichia coli,NC_000913.3)、阴沟肠杆菌ATCC13047(Enterobacter cloacae,NC_014121.1)、产单核细胞李斯特菌NCTC7974(Listeria monocytogenes,VEH62636.1)、金黄色葡萄球菌NCTC 8325(Staphylococcus aureus,NC_007795.1)。
1.1.2 基因分析软件LINUX系统安装ncbi-blast-2.9.0+、Samtools软件,WINDOWS下安装MAFFT(v7.037b)软件,同时使用下列在线工具:EMBOSS Transeq(https://www.ebi.ac.uk/Tools/st/emboss_transeq)、ESPript3.0(http://espript.ibcp.fr/ESPript/ESPript)、EMBL-EBI(https://www.ebi.ac.uk/Tools/st/)、BoxShade(https://embnet.vital-it.ch/software/BOX_form.html)、ProtParam(https://web.expasy.org/protparam/)、NCBI-conserved domains(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi?)和Phyre2(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)。
1.1.3 实验材料2X HiFi Master Mix、聚合酶链反应(PCR)产物回收磁珠购自Biorise Life Science公司;质粒pTargetF、pBBR1MCS-2、pCas9购自Addgene公司,其中pTargetF (Cm)质粒为本实验室保存,在pTargetF的664~1 323位点间加入了氯霉素抗性基因;质粒pSMART-LCKan购自Lucigen公司;Kpn I-HF、Hind Ⅲ-HF、Turbo感受态、Gibson组装克隆试剂盒购自NEW ENGLAND Biolabs公司;LB培养基:蛋白胨(tryptone)、酵母粉(yeast extract)为Oxoid公司产品,氯化钠(NaCl)、琼脂(agar)为Sigma公司产品;甘油、阿拉伯糖购自Sigma公司;卡那霉素(Kan)、氯霉素(Cm)购自索莱宝公司;亚胺培南/西司他丁(泰能)为默沙东公司产品;DNA提取试剂盒、质粒提取试剂盒购自天根公司;所用引物由擎科公司合成,KP 2017009菌株hfq基因敲除所用引物见表 1。
| Primer | Primer sequence 5′-3′ |
| Hfq sgRNA F | TGTTGCTGTGGTGAGACACCGTTTTAGAGCTAGAAATAGCAAG |
| Hfq sgRNA R | GGTGTCTCACCACAGCAACAACTAGTATTATACCTAGGACTGA |
| pTargetF Seq | CCGCCTTTGAGTGAGCTGAT |
| HfqΔ Up F | atcaagcttgaattcgttATGAATGATGTAACCGAGGC |
| HfqΔ Up R | agcactggccTCAGTTCTGGCTCGCACCAAC |
| HfqΔ Down F | ccagaactgaGGCCAGTGCTGTTTTTCCAC |
| HfqΔ Down R | atatctagagaattcgtcCCACATAGTCAACAAGCGTTG |
| SL1 | CAGTCCAGTTACGCTGGAGTC |
| SR2 | GGTCAGGTATGATTTAAATGGTCAGT |
| HfqΔ Seq1 | GCTCCATCAACGCATAGAAC |
| HfqΔ Seq2 | CGGCAGTGATCTGGTTAAAG |
| Hfq F | GAGTGAAACGATGGCTAAGGGGCAATCTTTACAAG |
| Hfq R | agggaacaaaagctgggtacttaGAGATCTTCTTCGCTAATCAGTTTCTGTTCGGCGTCGTCGCTGTCTT |
| HfqΔ F | ATGAATGATGTAACCGAGGCGA |
| HfqΔ R | CCACATAGTCAACAAGCGTTGG |
| araP F | GCTGCAGGAATTCGATATCATTATGACAACTTGACGGCTAC |
| araP R | CCTTAGCCATCGTTTCACTCCATCCAAAAAAAC |
提取细菌基因组,送浙江天科公司进行基因组测序。在LINUX下采用cat函数合并所有59例KP基因组,根据Blast比对结果,Samtools faidx提取菌株hfq基因。采用MAFFT软件将本院收集菌株的基因序列及蛋白质序列与下载的基因序列及蛋白序列进行多重序列比对,比对结果采用BoxShade标记。
| Drug* | Drug sensitivity | ||
| Sensitive | Intermediary | Resistance | |
| Ampicillin | 0 | 0 | 59 |
| Amoxicillin/Clavulanate | 19 | 7 | 33 |
| Piperacillin/tazobactam | 31 | 1 | 27 |
| Cefazolin | 18 | 0 | 41 |
| Cefuroxime | 16 | 0 | 43 |
| Ceftriaxone | 22 | 0 | 37 |
| Ceftazidime | 22 | 2 | 34 |
| Cefepime | 31 | 4 | 24 |
| Cefoperazone/sulbactam | 24 | 3 | 26 |
| Cefoxitin | 23 | 3 | 33 |
| Ertapenem | 31 | 0 | 28 |
| Imipenem | 34 | 0 | 25 |
| Meropenem | 32 | 0 | 24 |
| Ciprofloxacin | 28 | 6 | 25 |
| Levofloxacin | 36 | 1 | 22 |
| Gentamicin | 39 | 0 | 20 |
| Amikacin | 50 | 0 | 9 |
| Fosfomycin | 11 | 3 | 4 |
| Tigecycline | 35 | 1 | 23 |
| Aztreonam | 23 | 0 | 35 |
| Tobramycin | 34 | 8 | 17 |
| Nitrofurantoin | 7 | 22 | 30 |
| Sulfamethoxazole and trimethoprim | 32 | 0 | 26 |
| Ceftazidime, Aztreonam, Sulfamethoxazole and trimethoprim were detected in 58 cases, Meropenem was detected in 56 cases, Cefoperazone/sulbactam was detected in 53 cases, Fosfomycin was detected in 18 cases, and the others were detected in 59 cases. | |||
使用EMBOSS Transeq将59例KP的hfq基因翻译成蛋白质氨基酸序列,以编号20170009菌株的Hfq氨基酸序列为代表,联合11株临床常见菌株Hfq蛋白进行分析。以MAFFT软件进行氨基酸序列的多序列比对,用在线网站ESPript3.0分析蛋白二级结构。氨基酸序列理化性质用在线网站ProtParam计算。以NCBI数据库中保守结构域预测Hfq蛋白结构域。采用Phyre2预测Hfq蛋白三级结构。
1.2.3 KP hfq基因的敲除采用CRISPR/Cas9同源重组敲除20170009菌株hfq基因,步骤如下:引物Hfq sgRNA F/Hfq sgRNA R扩增pTargetF(Cm)质粒,添加Hfq sgRNA序列。PCR条件:预变性95 ℃ 3 min,变性98 ℃ 20 s,退火59 ℃ 15 s,延伸72 ℃ 1 min,10个循环;变性98 ℃ 20 s,退火+延伸72 ℃ 75 s,20个循环;最终延伸72 ℃ 120 s。PCR产物用Gibson组装克隆试剂盒参照说明书步骤连接线性化pTargetF(Cm)质粒,构建pTargetF(Cm)-Hfq质粒。电转化Turbo感受态细胞,Cm抗性选择LB培养板,37 ℃培养过夜。提取阳性菌质粒,用pTargetF Seq测序验证。分别使用引物Hfq Δ Up F/Hfq Δ Up R、Hfq Δ Down F/Hfq Δ Down R扩增KP 20170009 gDNA,PCR条件1:预变性95 ℃ 3 min,变性98 ℃ 20 s,退火63 ℃ 15 s,延伸72 ℃ 30 s,10个循环;变性98 ℃ 20 s,退火+延伸72 ℃ 45 s,20个循环;最终延伸72 ℃ 1 min。PCR条件2:预变性95 ℃ 3 min,变性98 ℃ 20 s,退火65 ℃ 15 s,延伸72 ℃ 45 s,10个循环;变性98 ℃ 20 s,退火+延伸72 ℃ 1 min,20个循环;最终延伸72 ℃ 90 s。上述所得产物采用Gibson组装克隆试剂盒参照说明书步骤连接线性化pSMART-LCKan质粒,构建Hfq Δ (009)/pSMART-LCKan质粒。电转化Turbo感受态细胞,Kan抗性选择LB培养板,37 ℃培养过夜。提取阳性菌质粒,用SL1/SR2、Hfq Δ Seq1/Hfq Δ Seq2测序验证。电转pCas9质粒到上述KP 20170009感受态,30 ℃复苏,铺LB+Kan平板,30 ℃培养过夜;次日取上述菌30 ℃培养,培养液加入阿拉伯糖10 mmol/L。参照1.2.3方法,制备KP 20170009 (pCas9)电转感受态细胞。电转100 ng pTargetF(Cm)-Hfq和400 ng Hfq Δ (009) KP 20170009 (pCas9)感受态细胞,30 ℃复苏。铺Kan+Cm抗性LB平板,30 ℃培养。使用引物Hfq Δ Seq1/Hfq Δ Seq2扩增阳性敲除菌株gDNA,PCR条件:预变性95 ℃3 min,变性98 ℃ 20 s,退火64 ℃ 10 s,延伸72 ℃ 45 s,30个循环;最终延伸72 ℃ 90 s。对产物进行琼脂糖凝胶电泳并测序验证。
1.2.4 菌株耐药性测定将20170009及20170009 Δ hfq菌株过夜振荡培养,1:100稀释于4 ml MH肉汤培养基中,分别加入0.1 μ g/mL、0.2 μ g/mL、0.3 μ g/mL泰能共培养,设未加泰能组为对照,分别取1 h、2 h、3 h、4 h菌液计数,绘制生长曲线。
1.2.5 统计学方法采用SPSS 22.0统计软件分析数据,以独立样本t检验比较各时间点菌落数,P < 0.05为差异有统计学意义。
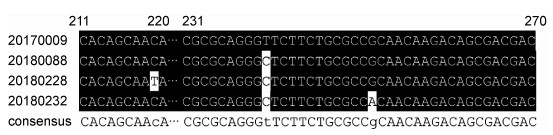
2 结果 2.1 临床分离KP hfq基因密码子相对保守共收集本院59例KP,其中分离自男性患者33例,女性患者26例。来源于胆汁及腹腔引流液36例,痰液15例,尿液6例,血液及咽拭子各1例。超广谱β -内酰胺酶(extended-spectrum β -lactamase,ESBL)阴性19例,ESBL阳性11例,碳青霉烯类耐药28例。分析所收集的菌株hfq基因发现,3株hfq基因存在个别密码子位点差异,如20180228的219位点C→T,20180088、20180228、20180232的240位点T→C,20180232的252位点G→A,见图 1。
|
| The nucleotide sequence alignment results by MAFFT are marked by the BoxShade, the black background color are labeled as a consensus sites, the unlabeled are mutation sites, the consensus is all sequence identity information, and the mutation site position is marked by a lowercase letter. 图 1 部分KP hfq基因的核酸序列比对结果 Fig. 1 Part of Klebsiella pneumoniae hfq gene nucleic acid sequence alignment results |
ProtParam预测的氨基酸pI、消光系数、稳定性、亲水性等理化指数详见表 3。所有菌株Hfq蛋白均为亲水蛋白。金黄色葡萄球菌、产单核细胞李斯特菌Hfq蛋白稳定性高,其他菌株Hfq蛋白均不稳定。肠杆菌Hfq蛋白含有相同的正负电荷氨基酸数,与弧菌、铜绿假单胞菌、洋葱伯克霍尔德菌存在差别,其中仅金黄色葡萄球菌含有较多的负电荷氨基酸。大肠埃希菌Hfq蛋白序列与KP存在2个位点差别,分别为大肠埃希菌79位点插入苏氨酸(T),94位点缺失脯氨酸(P)。在2017009序列的上述位点插入T及删除P,然后分别计算理化值(表 3),20170009-79Tins和20170009-94Pdel主要表现为指数不稳定及亲水性改变,对消光系数无影响。
| Strain | Aminoacid | Molecular weight |
pI | Negative AA1 |
Positive AA |
Extinction coefficient |
Unstable index |
Aliphatic index |
GRAVY |
| 20170009 | 102 | 11 003.21 | 6.96 | 8 | 8 | 4 470 | 66.59 | 78.33 | -0.456 |
| E.coli | 102 | 11 166.39 | 6.97 | 8 | 8 | 4 470 | 66.91 | 77.35 | -0.505 |
| 20170009-79Tins | 103 | 111 04.32 | 6.96 | 8 | 8 | 4 470 | 65.21 | 77.57 | -0.458 |
| 20170009-94Pdel | 101 | 10 906.10 | 6.96 | 8 | 8 | 4 470 | 63.33 | 79.11 | -0.445 |
| S.enterica | 102 | 11 133.36 | 6.97 | 8 | 8 | 4 470 | 63.96 | 78.33 | -0.503 |
| V.parahaemolyticus | 87 | 9 839.23 | 9.22 | 9 | 11 | 2 980 | 62.29 | 89.54 | -0.508 |
| V.cholerae | 87 | 9 769.18 | 9.22 | 9 | 11 | 2 980 | 61.61 | 91.84 | -0.391 |
| S.dysenteriae | 102 | 11 192.42 | 6.97 | 8 | 8 | 4 470 | 73.10 | 76.37 | -0.538 |
| E.cloacae | 103 | 11 118.34 | 6.97 | 8 | 8 | 4 470 | 61.32 | 77.57 | -0.457 |
| B.cepacia | 79 | 8 849.19 | 8.12 | 6 | 7 | 4 470 | 53.73 | 102.28 | -0.084 |
| P.aeruginosa | 82 | 9 103.52 | 9.52 | 6 | 9 | 4 470 | 58.03 | 97.32 | -0.244 |
| S.aureus | 77 | 8 778.73 | 4.69 | 12 | 6 | 5 960 | 39.96* | 74.68 | -0.544 |
| L.monocytogenes | 77 | 8 714.99 | 9.16 | 7 | 9 | 2 980 | 33.93* | 92.34 | -0.275 |
| The negative amino acids are Asp and Glu, and positive amino acids are Arg and Lys. GRAVY: grand average of hydropathicity. | |||||||||
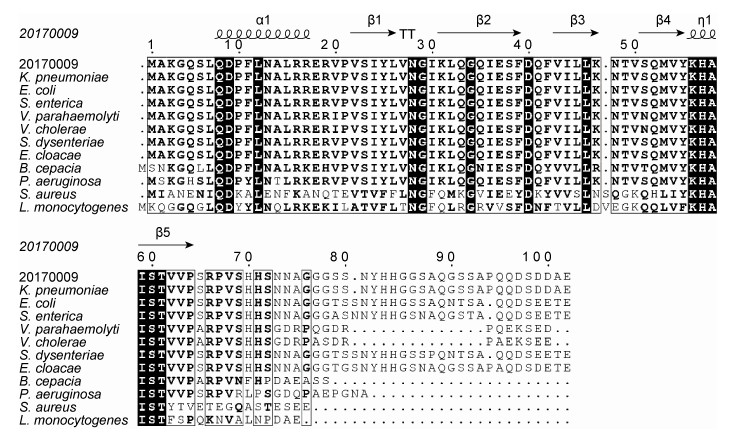
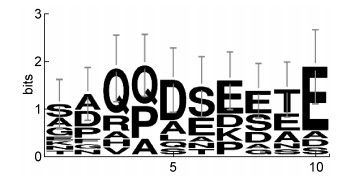
将上述hfq核苷酸序列翻译成蛋白质氨基酸序列,59株KP Hfq氨基酸序列完全一致,且与NCBI数据库中的KP(HS11286)序列一致,见图 2。因此,选用20170009菌株Hfq氨基酸序列与数据库下载的11株临床常见菌株Hfq氨基酸序列进行后续分析。氨基酸位点一致性如图 2中黑色阴影标注所示,所有菌株序列在N末端高度保守,均具有1个α螺旋及后续连着5个β折叠(β 1~ β 5)的二级结构,金黄色葡萄球菌、产单核细胞李斯特菌Hfq氨基酸的N末端与其他菌株也有明显差别。KP的Hfq氨基酸序列与大肠埃希菌、阴沟肠杆菌、痢疾志贺菌相似性较高,弧菌、铜绿假单胞菌、洋葱伯克霍尔德菌Hfq氨基酸与肠杆菌的主要差别在C末端,表现为明显截短,如图 2。C末端序列以酸性氨基酸为主,末端10个氨基酸pI在3.37~4.41,包括谷氨酰胺(Q)、脯氨酸(P)、天冬氨酸(D)、丝氨酸(S)、谷氨酸(E)、苏氨酸(T)等,见图 3。
|
| The amino acid sequence alignment results are labeled by ESPript3.0, the sequence identity marked black background marker, and which secondary structure are predicted, such as the α 1 helix and β 1-β 5 fold upper in the figure, and the amino acid position is indicated by the sequence position of 20170009 strain. 图 2 Hfq氨基酸序列比对结果及二级结构预测 Fig. 2 Hfq amino acid sequence alignment results and secondary structure |
|
| The 10 amino acids of the C-terminal of Hfq protein were shown by weblogo3. 图 3 Hfq蛋白C末端氨基酸保守位点 Fig. 3 Conserved sites of Hfq protein C-terminal amino acid sequence |
用NCBI保守结构域进行检索,结果提示20170009 Hfq氨基酸序列保守结构位于1~76位点,与图 2结果一致。其中,22-VSIYLVN*GIKLQGQIESRDQFVILLK-47为Sm1基序,51-SQMVYKHAISTV-62为Sm2基序。含有11个RNA结合位点,分别为25Y、29G、30I、31K、32L、33Q、46L、52Q、60S、61T和63V。
2.5 β4-β5区是Hfq蛋白的重要功能区域用NCBI保守结构域检索Hfq氨基酸序列,提示六聚体结合位点为6-SLQ-8、11F、26-LVNGI-30、38-SFD-40、42-FV-43、50-VSQMVYKHAISTVVP-64。采用Phyre2预测三级结构,PSI-Blast多序列比对同源蛋白,评估E值< 0.001的有50条氨基酸序列;利用c3qhsD(E-coli)模板建模,68(66.67%)个氨基酸比对上模板序列,置信度为100%。单体的模型质量、保守区域、突变敏感区、接触界面及口袋预测结果详见图 4。分析发现,其中较为保守且具备重要功能的位点主要位于β 4~ β 5连接区的54-VYKHAI-59,序列谱提示各位点为I、Y、K、H、A、I可能性最大,56、57、58位点最为保守,其他存在54位点V、55位点F、59位点V的可能,55位点存在F和Y两个野生型位点,详见图 5。3DLigandSite配体结合位点预测出25Y和31K。

|
| The results were predicted by Phyre2. A: the ProQ2 quality assessment. B: The prediction results of conservative area. C: The mutational sensitivity of Hfq. D: The ProtinDB interface. E: the pocket detection. 图 4 Hfq单体三级结构特点预测结果 Fig. 4 Prediction results of three-dimensional structural characteristics of Hfq monomer |

|
| The four conservative residues sequence profiles were by scanning against the database using PSI-Blast and the mutations were made using the SuSPect method in Phyre2. A: Residue 54V. B: Residue 55Y. C: Residue 56K. D: Residue 57H. E: Residue 58A. F: Residue 59I. 图 5 Hfq氨基酸残基序列谱及突变倾向 Fig. 5 Hfq amino acid residue sequence profile and mutation tendency |
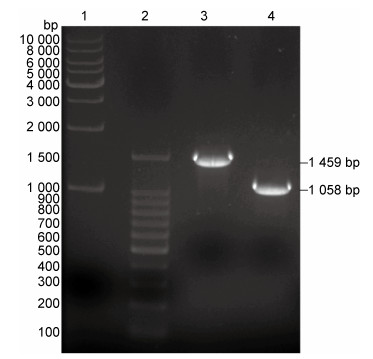
采用CRISPR/Cas9同源重组敲除20170009菌株hfq基因,挑选基因敲除后的菌株及原始菌株,煮沸法提取基因组,采用Hfq Δ Seq1/Hfq Δ Seq2引物进行PCR扩增及1%琼脂糖凝胶电泳,结果见图 6。PCR产物测序结果提示hfq基因成功敲除。
|
| Line 1: Marker 1(1kb DNA Ladder, 1000-10000bp);Line 2: Marker 2(100 bp DNA Ladder, 100-1500 bp);Line 3: 20170009 PCR results(1 459 bp);Line 4: 20170009 Δ hfq PCR results (1 058 bp). 图 6 20170009及Δhfq菌株基因组PCR鉴定结果 Fig. 6 Genomic PCR results of 20170009 and Δhfq strain |
将野生株及基因敲除株在不同泰能浓度下培养,绘制生长曲线,结果见图 7。两者在无抗生素存在下,菌落数无明显统计学差异。在各浓度的泰能组,009 Δ hfq菌落数均较009野生株菌落数减少,其中0.1 μ g/mL浓度组在第4小时P=0.039,差异有统计学意义;0.2 μ g/mL浓度组在第1、3、4小时P值分别为0.008、0.005、0.08,差异有统计学意义;0.3 μ g/mL浓度组在第1、3、4小时P值分别为0.007、0.001、0.001, 差异有统计学意义。结果表明,hfq基因敲除菌株的耐药能力下降。

|
| IMP: Imipenem. 图 7 20170009及Δhfq菌株在抗生素不同溶度刺激下的生长曲线 Fig. 7 Growth curve of 20170009 and Δhfq strain stimulated by different concentration of antibiotics |
Hfq蛋白存在于约50%的细菌中,是一个全局性调节因子,在细菌生长、趋化、毒力、耐药、应对外界选择压力方面起重要调节作用[11]。本研究结果发现KP的Hfq蛋白氨基酸序列非常保守,仅3株菌株的个别密码子存在差异,氨基酸序列则完全一致,且与NCBI数据库中的KP株(HS11286)保持一致。与同为肠杆菌科的大肠埃希菌比对,发现仅2个位点存在差别,分别为在79位点缺失苏氨酸(T)及在94位点插入脯氨酸(P),且均发生在保守区外的C末端,理化性质上主要影响Hfq蛋白的稳定性和亲水性。
二级结构分析提示N末端更加保守,预测KP的Hfq氨基酸N末端含有Sm1和Sm2两个结构,且发现β 4~ β 5连接区较为保守,尤其是56 K位点,提示是重要功能位点。同区域中,铜绿假单胞菌的57H[12]、恶臭假单胞菌56 K[13]均与空间结构的稳定相关。此外,与本例产单核细胞李斯特菌Hfq 58位点的“A”不同。Kovach等研究结果显示,58位点为“K”,也是RNA的结合位点[14]。KP Hfq蛋白的该区域是否与空间位置形成相关还需后续研究证实。
Hfq蛋白的C末端由于在不同菌株存在较大差异,目前对其结构及功能的了解有限。有研究发现,大肠埃希菌C末端可形成淀粉样蛋白的纤维状结构,有利于Hfq蛋白在细胞内定位[15],且Hfq蛋白的C末端所含氨基酸偏酸性,有利于RNA退火,对sRNA与mRNA和DNA的结合以及RNA与Sm区域的结合也有促进作用[16]。本研究发现包括KP在内的各菌株C末端氨基酸pI都在4.0左右,可能具有类似的功能。以大肠埃希菌的Hfq序列为模板,用Phyre2预测KP的Hfq蛋白三维模型,仅N末端68(66.67%)个氨基酸比对上模板序列,单体结构见图 4和图 5。Hfq功能蛋白为由6个单体组成的圆盘状六聚体结构,研究表明近侧面结合RNA的poly U结构[17],远侧面可结合AAN重复序列而发挥调节作用。此外它也可参与自身调节[18]。近年来还发现部分RNA与Hfq蛋白的边缘结构结合而发挥作用,侧面边缘和近端在结构上更靠近C末端区域,观察到之间存在瞬时联系,可能有利于C末端释放双链RNA和sRNA[19]。此外,Hfq六聚体侧面边缘结构与RNA的结合在空间上依赖于各单体的精氨酸在侧面边缘结构的聚集[20]。大肠埃希菌由于缺乏精氨酸,其侧面边缘结构对sRNA、mRNA的作用有限[2]。KP的Hfq蛋白与大肠埃希菌Hfq蛋白类似,也缺乏精氨酸,边缘结构的作用可能同样有限。
既往研究提示,Hfq蛋白与KP的适应能力及毒力相关。如Chiang等研究发现,有17.3%和19.5%的基因依赖Hfq蛋白而作用于RpoS、RpoE等因子,调节KP毒力基因表达[21]。也有研究提示,Hfq蛋白介导sRNA广泛参与代谢酶、外排泵、转运蛋白等的调节,从而参与耐药机制[4-5]。敲除hfq基因的铜绿假单胞菌对抗生素的敏感性提高,该过程与viz泵、能量代谢、细胞壁和脂多糖(lipopolysaccharides, LPS)组分的改变有关[22]。在维氏气单胞菌中,hfq基因缺失株可对替加环素、头孢噻肟、环丙沙星等多种抗生素的耐药性下降,且可能与RelE、CspD、ClpB、RpoS、OxyR基因下调和OppB基因上调有关[23]。大肠埃希菌在敲除hfq基因后对多种抗生素的敏感性恢复,如同时敲除外排泵acrB基因,可抵消这种作用,提示这可能与hfq调节外排泵有关[24]。另有研究发现,多种sRNA通过Hfq蛋白参与大肠埃希菌和沙门菌的耐药[25]。本研究采用CRISPR/Cas同源重组法获得1株敲除hfq基因的KP菌,与野生株相比,Δ hfq菌株耐碳青霉烯类能力下降,但是菌株生长无明显影响,提示hfq基因参与了KP的耐药调节。此外,该Δ hfq菌株可用于进一步研究其在KP病理生理中的作用,揭示Hfq蛋白不同氨基酸位点的功能。
总之,本研究结果提示KP的Hfq蛋白较为保守,VYKHAI可能是重要的功能结构域,hfq基因缺失株耐药性明显下降,明确Hfq蛋白功能具有重要的临床意义。今后将进一步研究KP耐药及其毒力调节的分子机制,以期为抗感染药物的研发奠定基础。
| [1] |
Gottesman S. Trouble is coming:Signaling pathways that regulate general stress responses in bacteria[J]. J Biol Chem, 2019, 294(31): 11685-11700.
[DOI]
|
| [2] |
Santiago-Frangos A, Woodson SA. Hfq chaperone brings speed dating to bacterial sRNA[J]. Wiley Interdiscip Rev RNA, 2018, 9(4): e1475.
[DOI]
|
| [3] |
Dos Santos RF, Arraiano CM, Andrade JM. New molecular interactions broaden the functions of the RNA chaperone Hfq[J]. Curr Genet, 2019, 65(6): 1313-1319.
[DOI]
|
| [4] |
Dersch P, Khan MA, Mühlen S, Görke B. Roles of regulatory RNAs for antibiotic resistance in bacteria and their potential value as novel drug targets[J]. Front Microbiol, 2017, 8: 803.
[DOI]
|
| [5] |
Pei XY, Dendooven T, Sonnleitner E, Chen S, Bläsi U, Luisi BF. Architectural principles for Hfq/Crc-mediated regulation of gene expression[J]. Elife, 2019, 8: e43158.
[DOI]
|
| [6] |
Zhang YF, Han K, Chandler CE, Tjaden B, Ernst RK, Lory S. Probing the sRNA regulatory landscape of P. aeruginosa:post-transcriptional control of determinants of pathogenicity and antibiotic susceptibility[J]. Mol Microbiol, 2017, 106(6): 919-937.
[DOI]
|
| [7] |
Zhang L, Yu W, Tang Y, Li H, Ma X, Liu Z. RNA chaperone hfq mediates persistence to multiple antibiotics in Aeromonas veronii[J]. Microb Pathog, 2019, 132: 124-128.
[DOI]
|
| [8] |
Bhatt S, Egan M, Ramirez J, Xander C, Jenkins V, Muche S, El-Fenej J, Palmer J, Mason E, Storm E, Buerkert T. Hfq and three Hfq-dependent small regulatory RNAs-MgrR, RyhB and McaS-coregulate the locus of enterocyte effacement in enteropathogenic Escherichia coli[J]. Pathog Dis, 2017, 75(1): ftw113.
[DOI]
|
| [9] |
胡付品, 郭燕, 朱德妹, 汪复, 蒋晓飞, 徐英春, 张小江, 张朝霞, 季萍, 谢轶, 康梅, 王传清, 王爱敏, 徐元宏, 沈继录, 孙自镛, 陈中举, 倪语星, 孙景勇, 褚云卓, 田素飞, 胡志东, 李金, 俞云松, 林洁, 单斌, 杜艳, 郭素芳, 魏莲花, 邹凤梅, 张泓, 王春, 胡云建, 艾效曼, 卓超, 苏丹虹, 汪瑞忠, 房华, 俞碧霞, 赵勇, 龚萍, 郭大文, 赵金英, 刘文恩, 李艳明, 金炎, 邵春红, 温开镇, 张贻荣, 徐雪松, 鄢超, 喻华, 黄湘宁, 王山梅, 楚亚菲, 张利侠, 马娟, 周树平, 周艳, 朱镭, 孟晋华, 董芳, 郑红艳, 沈瀚, 周万青, 贾伟, 李刚, 吴劲松, 卢月梅. 2017年CHINET中国细菌耐药性监测[J]. 中国感染与化疗杂志, 2018, 18(3): 241-251. [URI]
|
| [10] |
Liu YY, Wang Y, Walsh TR, Yi LX, Zhang R, Spencer J, Doi Y, Tian G, Dong B, Huang X, Yu LF, Gu D, Ren H, Chen X, Lv L, He D, Zhou H, Liang Z, Liu JH, Shen J. Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China:a microbiological and molecular biological study[J]. Lancet Infect Dis, 2016, 16(2): 161-168.
[DOI]
|
| [11] |
Kavita K, de Mets F, Gottesman S. New aspects of RNA-based regulation by Hfq and its partner sRNAs[J]. Curr Opin Microbiol, 2018, 42: 53-61.
[DOI]
|
| [12] |
Moskaleva O, Melnik B, Gabdulkhakov A, Garber M, Nikonov S, Stolboushkina E, Nikulin A. The structures of mutant forms of Hfq from Pseudomonas aeruginosa reveal the importance of the conserved His57 for the protein hexamer organization[J]. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2010, 66(Pt 7): 760-764.
[URI]
|
| [13] |
Wirebrand L, Madhushani AWK, Irie Y, Shingler V. Multiple Hfq-Crc target sites are required to impose catabolite repression on (methyl)phenol metabolism in Pseudomonas putida CF600[J]. Environ Microbiol, 2018, 20(1): 186-199.
[DOI]
|
| [14] |
Kovach AR, Hoff KE, Canty JT, Orans J, Brennan RG. Recognition of U-rich RNA by Hfq from the Gram-positive pathogen Listeria monocytogenes[J]. RNA, 2014, 20(10): 1548-1559.
[DOI]
|
| [15] |
Fortas E, Piccirilli F, Malabirade A, Militello V, Trépout S, Marco S, Taghbalout A, Arluison V. New insight into the structure and function of Hfq C-terminus[J]. Biosci Rep, 2015, 35(2): e00190.
[DOI]
|
| [16] |
Santiago-Frangos A, Jeliazkov JR, Gray JJ, Woodson SA. Acidic C-terminal domains autoregulate the RNA chaperone Hfq[J]. Elife, 2017, 6: e27049.
[DOI]
|
| [17] |
Chen J, Morita T, Gottesman S. Regulation of transcription termination of small RNAs and by small RNAs:molecular mechanisms and biological functions[J]. Front Cell Infect Microbiol, 2019, 9: 201.
[DOI]
|
| [18] |
Morita T, Aiba H. Mechanism and physiological significance of autoregulation of the Escherichia coli hfq gene[J]. RNA, 2019, 25(2): 264-276.
[DOI]
|
| [19] |
Wen B, Wang W, Zhang J, Gong Q, Shi Y, Wu J, Zhang Z. Structural and dynamic properties of the C-terminal region of the Escherichia coli RNA chaperone Hfq:integrative experimental and computational studies[J]. Phys Chem Chem Phys, 2017, 19(31): 21152-21164.
[DOI]
|
| [20] |
Panja S, Schu DJ, Woodson SA. Conserved arginines on the rim of Hfq catalyze base pair formation and exchange[J]. Nucleic Acids Res, 2013, 41(15): 7536-7546.
[DOI]
|
| [21] |
Chiang MK, Lu MC, Liu LC, Lin CT, Lai YC. Impact of Hfq on global gene expression and virulence in Klebsiella pneumoniae[J]. PLoS One, 2011, 6(7): e22248.
[DOI]
|
| [22] |
Pusic P, Sonnleitner E, Krennmayr B, Heitzinger DA, Wolfinger MT, Resch A, Bläsi U. Harnessing metabolic regulation to increase Hfq-dependent antibiotic susceptibility in Pseudomonas aeruginosa[J]. Front Microbiol, 2018, 9: 2709.
[DOI]
|
| [23] |
Kim W, Choi JS, Kim D, Shin D, Suk S, Lee Y. Mechanisms for Hfq-independent activation of rpoS by DsrA, a small RNA, in Escherichia coli[J]. Mol Cells, 2019, 42(5): 426-439.
[URI]
|
| [24] |
Yamada J, Yamasaki S, Hirakawa H, Hayashi-Nishino M, Yamaguchi A, Nishino K. Impact of the RNA chaperone Hfq on multidrug resistance in Escherichia coli[J]. J Antimicrob Chemother, 2010, 65(5): 853-858.
[DOI]
|
| [25] |
Kim T, Bak G, Lee J, Kim KS. Systematic analysis of the role of bacterial Hfq-interacting sRNAs in the response to antibiotics[J]. J Antimicrob Chemother, 2015, 70(6): 1659-1668.
[URI]
|
 2020, Vol. 15
2020, Vol. 15


