2. 复旦大学附属华山医院感染科,上海 200040
2. Department of Infectious Diseases, Huashan Hospital, Fudan University, Shanghai 200040, China
乙型肝炎病毒(hepatitis B virus, HBV)感染作为一个主要的健康问题仍然影响着全世界2.4亿人口[1]。虽然,积极使用核苷类似物(nucleotide analogue,NA)和干扰素(interferon, IFN)治疗可以抑制病毒复制,延缓病理学改变和降低肝癌发生风险[2],但并不能完全清除肝内共价闭合环状DNA(covalently closed circular DNA, cccDNA)的复制,所以慢性乙型肝炎(chronic hepatitis B,CHB)患者在用药后仍然难以达到临床治愈[3]。
最近有研究报道,血清HBV RNA的浓度能反映肝脏中cccDNA的转录活性[4-8]。进一步研究发现,血清HBV RNA在NA和IFN治疗队列中都可以用于预测CHB患者的治疗疗效[6-7, 9-11]、指导停药时间[4]和监测抗病毒治疗期间病毒的突变[12-13]。因此,监测血清HBV RNA水平有助于在临床实践中指导CHB患者的起始治疗、治疗方案的改变和停止治疗的时间确定[4-5, 14-15]。由于CHB患者开始实施NA或IFN治疗之后,患者的血清HBV DNA可在短时间内低于检测下限(<100 IU/mL),但仍然可被检测到[16]。所以为了进一步研究HBV感染者的病毒学变化,对HBV DNA<100 IU/mL的CHB患者RNA监测显得尤为重要。
目前,国内外主要采用实时荧光核酸恒温扩增试验(simultaneous amplification testing, SAT)[17-18]和定量反转录-聚合酶链反应(quantitative reverse transcription-polymerase chain reaction, qRT-PCR)[16, 19]检测血清HBV RNA。但关于血清HBV RNA在HBV相关疾病的研究,不同文献的结果存在差异[17, 20-22]。除了研究对象的基线特征、样本量等方面的偏倚外,还可能由于各研究的血清HBV RNA检测方法不同所致。目前对上述2种方法的对比和一致性评价尚无明确报道。由于国内外血清HBV RNA测定流程缺乏标准化,同时无可用的公认标准品,因此无法确切评价该2种方法的灵敏度和特异度,所以现阶段只能通过不同方法间检测结果的一致性评价判断方法的差异性对各研究结果的影响。本研究旨在以SAT和qRT-PCR的常用RNA检测方法进行一致性评价,观察2种方法对HBV DNA≥100 IU/mL、HBV DNA<100 IU/mL的CHB患者以及非HBV感染患者血清中RNA检测结果的差异,以期为科研和临床应用提供依据。
1 材料和方法 1.1 材料 1.1.1 研究对象收集2019年4—9月在复旦大学附属公共卫生临床中心就诊的患者212例。其中CHB感染患者157例,无合并甲型肝炎病毒(hepatitis A virus, HAV)、丙型肝炎病毒(hepatitis C virus, HCV)、戊型肝炎病毒(hepatitis E virus, HEV)和人类免疫缺陷病毒(human immunodeficiency virus,HIV)感染,其诊断符合2019年修订的《慢性乙型肝炎防治指南》[23]。慢性HCV样本37例,无合并HAV、HBV、HEV和HIV感染,其诊断符合2019年修订的《慢性丙型肝炎防治指南》[24]。急性HEV感染患者18例,无合并HAV、HBV、HCV和HIV感染,其诊断符合《2018年欧洲肝病学会HEV感染临床实践指南》[25]。入组患者均签署知情同意书,并获得伦理委员会批准同意。
1.1.2 样本收集采集入组患者静脉血各5 mL,加至含分离胶的黄色促凝管中,室温静置30 min,室温下3 000 g离心15 min,将血清转移至1.5 mL离心管,-80 ℃保存。
1.1.3 试剂及仪器RNA SAT采用AutoSAT全自动核酸检测分析系统和RNA SAT检测试剂盒(仁度生物技术有限公司,上海)。qRT-PCR检测使用StepOne plus实时荧光定量PCR仪(Applied Biosystems公司,美国)。国产核酸提取或纯化试剂、国产HBV RNA定量检测试剂盒(PCR-荧光探针法)均为湖南圣湘公司产品。重组脱氧核糖核酸酶Ⅰ(recombinant DNase Ⅰ)和重组核糖核酸酶抑制剂(recombinant RNase inhibitor, RRI)均为日本TaKaRa Bio Group产品。HBV DNA定量检测试剂盒购自德国QIAGEN公司,采用罗氏公司的PCR仪进行检测,最低检测下限为100 IU/mL。
1.2 方法 1.2.1 SAT检测CHB患者和非HBV感染患者检测均采用AutoSAT全自动核酸检测分析系统。将400 μL血清放入仪器内,按照仪器和试剂使用说明书运行对应的程序,提取、扩增、检测及定量结果计算全部由仪器和软件自动完成,软件结果界面自动显示HBV RNA定量检测结果。仪器内部操作过程中,由HBV特异性RNA寡核苷酸磁珠微粒提取RNA,MMLV酶反转录目标RNA,T7 RNA聚合酶转录目标RNA,然后用荧光和淬灭剂标记的RNA信标探针进行检测。使用不同于HBV和人类基因组的核酸作为内对照(internal control, IC)。在核酸提取步骤中将相同剂量的IC加入每个样品中,IC扩增结果在理论上保持恒定。通过IC扩增结果校准,避免样本处理、扩增和检测中受HBV RNA扩增异常变化的影响。HBV阴性人血清稀释HBV RNA阳性标准品形成一系列浓度梯度检测,建立检测线性范围和标准曲线,再通过标准曲线得到血清HBV RNA浓度。线性浓度范围为2~8 log copies/mL。线性方程的R2≥0.95。最低检测下限为50 copies/mL。
1.2.2 qRT-PCR检测CHB患者和非HBV感染患者均按照试剂说明采用磁珠法从200 μL血清中提取HBV核酸。提取后的核酸用DNA酶消化和RNA酶抑制剂处理,以避免PCR假阳性。然后反转录HBV核酸中的pgRNA,用StepOne plus实时荧光定量PCR仪自动扩增和检测核酸。qRT-RCR反应条件:在反转录阶段,95 ℃ 1 min、60 ℃ 30 min;在PCR阶段,95 ℃ 1 min、95 ℃ 15 s、60 ℃ 30 s(45个循环),25 ℃ 10 s。该试剂盒设有阳性内对照(内标),以监测提取的核酸中是否具有PCR抑制物,从而评价核酸提取的质量,避免PCR假阴性。该试剂还设有内参比荧光ROX,用于校正加样误差和管间差异,以提高定量的准确性。试剂盒包含4个HBV RNA标准品,通过标准品建立标准曲线,再根据标准曲线计算样品浓度,所做标准曲线相关性R2≥0.98。试剂盒检测范围为2~9 log copies/mL,最低检测下限为100 copies/mL。
1.2.3 对2种检测方法的评价选用HBV DNA≥100 IU/mL的81例CHB患者血清样本,分别用SAT和qRT-PCR检测HBV RNA,分析和评价两者的检出率、相关性与一致性。选用HBV DNA<100 IU/mL的76例CHB患者血清样本,分别用SAT和qRT-PCR检测HBV RNA,分析和评价两者的检出率、相关性与一致性。以非HBV感染的37例HCV和18例HEV患者作为阴性对照样本进行检测,统计假阳性率,评价其特异度。
1.3 统计学分析数据采用SPSS19.0软件分析,图形用GrapPad Prism 8和MedCalc软件绘制。将HBV RNA和DNA载量换算成对数形式进行统计学分析,计量资料以均数±标准差(x±s)表示。连续变量比较采用配对t检验,分类变量比较采用χ2检验。对2种方法检测范围内数据计算决定性系数(R2)。采用Deming回归法进行相关性分析,用一致性相关系数(concordance correlation coefficient,CCC)及其95%置信区间(confidence interval, CI)比较方法间的一致性。绘制Bland-Altman图比较方法间的绝对与相对偏倚。P < 0.05为差异有统计学意义。
2 结果 2.1 入组患者的一般情况入组患者212例,其中HBV感染者157例、非HBV感染者55例(作为对照),所有入组患者的基线资料见表 1。
| Variables | Chronic HBV infection (n=157) | HCV infection (n=37) |
HEV infection (n=18) |
|
| HBV DNA≥ 100 IU/mL (n=81) |
HBV DNA< 10 IU/mL (n=76) |
|||
| Male [n (%)] | 45(55.56%) | 52(68.42%) | 23(62.16%) | 14(77.78%) |
| Age (year) | 38(32-42) | 40(33-47) | 53(43-57) | 61(55-68) |
| HBsAg (log IU/mL) | 3.54(3.15-4.05) | 3.04(2.69-3.47) | 0 | 0 |
| HBeAg positive [n (%)] | 52(64.20%) | 23(30.26%) | 0 | 0 |
| HBV DNA (log IU/mL) | 4.37(2.23-6.81) | <100 | <100 | <100 |
| ALT (IU/L) | 85(26-123) | 29(19-28) | 69(34-81) | 985(144-1263) |
| AST (IU/L) | 48(21-55) | 24(20-25) | 54(37-67) | 468(53-718) |
| HBV: hepatitis B virus; HCV: hepatitis C virus; HEV: hepatitis E virus.HBsAg: hepatitis B surface antigen; HBeAg: hepatitis B e antigen. ALT: alanine aminotransferase; AST: aspartate aminotransferase. | ||||
采用SAT和qRT-PCR分别检测HBV DNA≥100 IU/mL的81例CHB患者血清样本中的HBV RNA。SAT中,除4例HBV DNA低于检测下限外,77例检测结果阳性,检出率为95.06%(77/81);qRT-PCR中,除4例HBV DNA低于检测下限外,也有77例检测结果阳性,检出率为95.06%(77/81)。比较2种方法的检出率,差异无统计学意义(P>0.05)。
在81例样本中,血清HBV DNA 2例低于SAT检测下限,2例低于qRT-PCR检测下限,2例同时低于2种方法的检测下限。最终以75例有效数据进行方法学对比。DNA≥100 IU/mL的样本,2种方法间对比Deming回归法斜率的95% CI区间包括1,截距的95% CI区间不包括0,说明2种方法间不存在比例性偏差,仅存在非比例性偏差。2种方法间R2值为0.803,CCC为0.882 (见表 2)。2种方法间Deming回归图见图 1A。2种方法间的绝对偏倚为0.1 log copies/mL,数据显示4.00%(3/75)的样本在95%一致性界线外(图 1B);相对偏倚为0.97%,数据显示4.00%(3/75)的样本在95%一致性界线外(图 1C)。经配对t检验分析,2种方法的检测结果差异无统计学意义(t=1.617,P=0.110)。
| Viral load | SAT vs qRT-PCR | ||||||||
| Deming regression | Bland-altman | Consistency test | |||||||
| Slope (95%CI) |
Intercept (95%CI) |
R2 | Absolute bias (log copies/mL) |
Relative bias(%) |
CCC | 95%CI | |||
| HBV DNA≥ 100 IU/mL |
0.858 (0.714~1.002) |
0.973 (0.012~1.935) |
0.803 | -0.100 | 0.970 | 0.882 | 0.824~0.922 | ||
| HBV DNA< 100 IU/mL |
0.545 (0.337~0.753) |
1.985 (1.322~2.647) |
0.326 | -0.500 | 0.860 | 0.438 | 0.282~0.572 | ||
| SAT: simultaneous amplification testing; qRT-PCR: quantitative reverse transcription-polymerase chain reaction. HBV: hepatitis B virus; CHB: chronic hepatitis B; CCC: concordance correlation coefficient; CI: confidence interval. | |||||||||
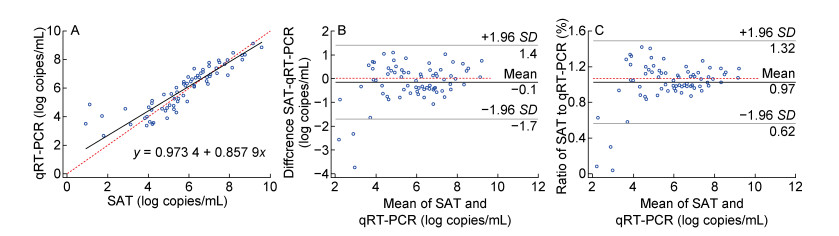
|
| A.Deming Regression lines indicated by the black Line. X=Y is indicated by the red Line. B. Bland Altman plot show absolute bias between SAT and qRT-PCR methods. The mean difference between SAT and qRT-PCR is given (Y-axis) for the mean results (average of SAT and qRT-PCR). C. Bland Altman plot show relative bias between SAT and qRT-PCR methods. The radio between SAT and qRT-PCR is given (Y-axis) for the mean results (average of SAT and qRT-PCR). Limits of agreement (LoA) are defined by 95% confidence intervals (gray lines). Bias is indicated by the red Line. SAT: simultaneous amplification testing; qRT-PCR: quantitative reverse transcription-polymerase chain reaction. 图 1 SAT和qRT-PCR方法在HBV DNA≥100 IU/mL样本中的Deming回归、绝对偏倚和相对偏倚 Fig. 1 Deming Regression, absolute bias and relative bias between SAT and qRT-PCR methods for samples with HBV RNA≥100 IU/mL |
采用SAT和qRT-PCR分别检测HBV DNA<100 IU/mL的76例CHB患者血清样本中的HBV RNA,结果见表 3。SAT中,除1例HBV DNA低于检测下限外,75例检测结果HBV RNA阳性,检出率为98.68%(75/76);qRT-PCR中,除9例HBV DNA低于检测下限外,67例检测结果阳性,检出率为88.16%(67/76)。SAT方法的检出率>qRT-PCR。2种方法的检测结果,差异有统计学意义(P < 0.001)。
| SAT | qRT-PCR | P value | ||
| Positive | Undetected | Total | ||
| Positive | 67 | 8 | 75 | <0.001 |
| Undetected | 0 | 1 | 1 | |
| Total | 67 | 9 | 76 | |
| SAT: simultaneous amplification testing; qRT-PCR: quantitative reverse transcription-polymerase chain reaction. | ||||
在76例CHB患者血清样本中,8例HBV DNA低于qRT-PCR检测下限,1例HBV DNA同时低于2种方法的检测下限。最终以67例有效数据进行方法学对比。结果显示,2种方法对比的Deming回归法斜率的95% CI区间不包括1,截距的95% CI区间不包括0,说明它们之间存在比例性和非比例性偏差。2种方法间R2值为0.326,CCC为0.438(见表 2)。2种方法间Deming回归图见图 2A。2种方法的绝对偏倚为0.5 log copies/mL,数据显示无样本在95%一致性界线外(图 2B);相对偏倚为0.86%,数据显示4.48%(3/67)的样本在95%一致性界线外(图 2C)。经配对t检验分析,2种方法的检测结果差异有统计学意义(t=3.654,P < 0.001)。qRT-PCR的检测结果均值>SAT(图 3,P=0.010)。

|
| A. Deming Regression line is indicated by the black Line. X=Y is indicated by the red Line. B. Bland Altman plot show absolute bias between SAT and qRT-PCR methods. The mean difference between SAT and qRT-PCR is given (Y-axis) for the mean results (average of SAT and qRT-PCR). C. Bland Altman plot show relative bias between SAT and qRT-PCR methods. The radio between SAT and qRT-PCR is given (Y-axis) for the mean results (average of SAT and qRT-PCR). Limits of agreement (LoA) are defined by 95% confidence intervals (gray lines). Bias is indicated by the red Line. SAT: simultaneous amplification testing; qRT-PCR: quantitative reverse transcription-polymerase chain reaction. 图 2 SAT和qRT-PCR方法在HBV DNA<100 IU/mL样本中的Deming回归、绝对偏倚和相对偏倚 Fig. 2 Deming Regression, absolute bias and relative bias between SAT and qRT-PCR methods for samples with HBV RNA < 100 IU/mL |

|
| 图 3 SAT和qRT-PCR方法在HBV DNA<100 IU/mL样本中的比较 Fig. 3 Comparison of SAT and qRT-PCR methods for samples with HBVDNA < 100 IU/mL |
采用SAT和qRT-PCR检测55例对照样本(均为非HBV感染患者,其中37例为HCV感染患者、18例为HEV感染患者)。最终纳入分析的55例对照样本中,SAT和qRT-PCR均未检出HBV RNA阳性。可以认为,2种方法对非HBV感染样本的检测结果,差异均无统计学意义。
3 讨论在分析CHB疾病进展、抗病毒治疗效果、肝内病毒复制状态以及耐药检测中,血清HBV RNA的定量检测具有重要意义[5, 13, 26-27]。目前国内外血清HBV RNA检测暂时缺乏标准化,主要采用SAT和qRT-PCR进行检测。有关血清HBV RNA的临床研究,各自结果存在差异,而不同的RNA检测方法对研究结果差异的影响仍未知。因此,本研究对SAT和qRT-PCR分别在HBV DNA<100 IU/mL和HBV DNA≥100 IU/mL的CHB患者样本以及非HBV感染患者样本中的检测结果进行了对比。
通过实验发现,HBV DNA≥100 IU/ mL的样本中,2种方法表现出较好的相关性与一致性,说明2种方法的检测结果可比性好,可较准确地反映血清HBV RNA水平。对于HBV DNA≥100 IU/ mL的CHB患者,在血清HBV RNA与乙型肝炎自然史和肝脏病理变化的相关性、RNA评估抗病毒治疗效果和预测停药后复发的研究中,2种方法的检测结果可信度高,结论可互相参考。在HBV DNA<100 IU/mL的CHB患者样本中,2种方法的HBV RNA检出率均较高,但qRT-PCR方法的检出率小于SAT(88.166% vs 98.68%, P=0.009)。这一结果表明,当报告样本HBV DNA浓度低于检测下限时大部分患者仍可被检测到RNA。这与Fan等[16]研究结果一致,提示血清HBV RNA检测在HBV DNA<100 IU/mL的CHB患者中有一定价值,然而2种方法间的相关性与一致性较差,检测的结果可信度低,不能互相参考。所以对于HBV DNA<100 IU/mL的CHB患者,单一的血清HBV RNA检测对于监测病毒载量的变化、指导治疗和判断停药终点无较好效果。因此在将血清HBV RNA的检测用于监测患者抗病毒疗效时应综合考虑免疫学和生化学指标,发现可疑结果时应使用不同方法对RNA进行复检。此外,2种方法在非HBV感染样本的HBV RNA检测中均表现出较好的特异性,没有出现假阳性结果。
出现上述结果的可能原因是因为2种方法的检测步骤存在显著差异,主要表现在RNA抽提、引物和探针设计、内对照、标准品制备和检测方式。首先,qRT-PCR检测的HBV pgRNA序列经反转录后长度为HBV基因组大小的1.1倍,完全覆盖了与HBV基因组长度相同的整个HBV松弛环状DNA(relaxed circular DNA,rcDNA)序列[28]。所以在进行qRT-PCR前,需预先消化和提取DNA和RNA混合物中的HBV DNA[4, 29]。但在实际操作中很少能通过DNase完全消除DNA,残留的DNA会造成污染。且DNA消化步骤不可避免地导致RNA丢失,降低检测的准确性。而SAT通过磁珠微粒直接特异性捕获HBV pgRNA,避免了DNA消化步骤。第二,2种方法在引物和探针的设计上不同。qRT-PCR是利用针对HBV pgRNA序列设计的一对接头引物和一条特异性荧光探针(TaqMan探针),实现HBV pgRNA的定量检测。而SAT在特异性引物基础上使用的是分子信标技术。第三,qRT-PCR将内对照加在扩增体系中,虽然可监测样品扩增过程,但无法监测提取过程中样品的损耗。而SAT在核酸提取步骤就加入了IC,可以同时控制样本制备和扩增过程。第四,2种方法在定量步骤中均使用各自内部合成的标准品,由于方法本身引物和探针的特异性,标准品之间无法进行有效对比。第五,qRT-PCR需要人工操作且步骤较为烦琐,而SAT技术为全自动检测,在检测过程中造成误差的可能性更低。这些原因使得qRT-PCR的结果与SAT相比可能会出现一定的差异。尤其是HBV DNA<100 IU/mL的样本中,在病毒复制较不活跃,这一差异更为显著。在关于HBV DNA检测方法的研究中[30],同样出现不同方法对高病毒载量样本检测差异无统计学意义,但对低病毒载量样本差异有统计学意义这一现象。虽然2种方法之间存在诸多不同,但设计的引物和探针均对血清HBV RNA表现出较好的特异性。
本研究仅在所纳入2种主要的血清HBV RNA检测系统进行对比,研究样本量有限,故具有一定的局限性。需要强调的是,目前国际上尚无统一的HBV RNA检测方法与标准品,不同方法的一致性好不等同于正确度佳,只有以公认参考方法为参比方法时,不同方法间一致性好才能被认为具有良好的正确度。在下一步研究中,应纳入更多的检测方法,在更大规模的样本中全面评估现有方法检测的准确性,加快国际公认的检测方法和标准品的研究。
综上所述,目前常用的SAT和qRT-PCR测定血清HBV RNA的结果在DNA≥100 IU/mL中具有较好的相关性与一致性。2种方法基本能反映血清HBV RNA水平且结果具有一定的可比性,在相关研究的对比中有参考价值。但在HBV DNA<100 IU/mL中相关性和一致性存在一定差异,可比性不足。在对相关研究结果进行对比时应考虑检测方法不同造成的差异。另外,亟待建立国际血清HBV RNA检测标准方法,规范定量标准品的制备和改进试剂检测的灵敏度与特异度,推动临床血清HBV RNA标准化检测的建立。
| [1] |
Schweitzer A, Horn J, Mikolajczyk RT, Krause G, Ott JJ. Estimations of worldwide prevalence of chronic hepatitis B virus infection: a systematic review of data published between 1965 and 2013[J]. Lancet, 2015, 386(10003): 1546-1555.
[DOI]
|
| [2] |
Trépo C, Chan HL, Lok A. Hepatitis B virus infection[J]. Lancet, 2014, 384(9959): 2053-2063.
[DOI]
|
| [3] |
Ding S, Robek MD. Cytidine deamination and cccDNA degradation: A new approach for curing HBV[J]. Hepatology, 2014, 60(6): 2118-2121.
[DOI]
|
| [4] |
Wang J, Shen T, Huang X, Kumar GR, Chen X, Zeng Z, Zhang R, Chen R, Li T, Zhang T, Yuan Q, Li PC, Huang Q, Colonno R, Jia J, Hou J, McCrae MA, Gao Z, Ren H, Xia N, Zhuang H, Lu F. Serum hepatitis B virus RNA is encapsidated pregenome RNA that may be associated with persistence of viral infection and rebound[J]. J Hepatol, 2016, 65(4): 700-710.
[DOI]
|
| [5] |
Giersch K, Allweiss L, Volz T, Dandri M, Lütgehetmann M. Serum HBV pgRNA as a clinical marker for cccDNA activity[J]. J Hepatol, 2017, 66(2): 460-462.
[DOI]
|
| [6] |
van BÖmmel F1, Bartens A, Mysickova A, Hofmann J, Krüger DH, Berg T, Edelmann A. Serum hepatitis B virus RNA levels as an early predictor of hepatitis B envelope antigen seroconversion during treatment with polymerase inhibitors[J]. Hepatology, 2015, 61(1): 66-76.
[DOI]
|
| [7] |
Jansen L, Kootstra NA, van Dort KA, Takkenberg RB, Reesink HW, Zaaijer HL. Hepatitis B virus pregenomic RNA is present in virions in plasma and is associated with a response to pegylated interferon Alfa-2a and nucleos(t)ide analogues[J]. J Infect Dis, 2016, 213(2): 224-232.
[DOI]
|
| [8] |
Nassal M. HBV cccDNA: viral persistence reservoir and key obstacle for a cure of chronic hepatitis B[J]. Gut, 2015, 64(12): 1972-1984.
[DOI]
|
| [9] |
Huang YW, Takahashi S, Tsuge M, Chen CL, Wang TC, Abe H, Hu JT, Chen DS, Yang SS, Chayama K, Kao JH. On-treatment low serum HBV RNA level predicts initial virological response in chronic hepatitis B patients receiving nucleoside analogue therapy[J]. Antivir Ther, 2015, 20(4): 369-375.
[URI]
|
| [10] |
Rokuhara A, Matsumoto A, Tanaka E, Umemura T, Yoshizawa K, Kimura T, Maki N, Kiyosawa K. Hepatitis B virus RNA is measurable in serum and can be a new marker for monitoring lamivudine therapy[J]. J Gastroenterol, 2006, 41(8): 785-790.
[DOI]
|
| [11] |
Huang YW, Chayama K, Tsuge M, Takahashi S, Hatakeyama T, Abe H, Hu JT, Liu CJ, Lai MY, Chen DS, Yang SS, Kao JH. Differential effects of interferon and lamivudine on serum HBV RNA inhibition in patients with chronic hepatitis B[J]. Antivir Ther, 2010, 15(2): 177-184.
[DOI]
|
| [12] |
Hatakeyama T, Noguchi C, Hiraga N, Mori N, Tsuge M, Imamura M, Takahashi S, Kawakami Y, Fujimoto Y, Ochi H, Abe H, Maekawa T, Kawakami H, Yatsuji H, Aisaka Y, Kohno H, Aimitsu S, Chayama K. Serum HBV RNA is a predictor of early emergence of the YMDD mutant in patients treated with lamivudine[J]. Hepatology, 2007, 45(5): 1179-1186.
[DOI]
|
| [13] |
Bottecchia M, Barcaiu HS, Lewis-Ximenez LL, da Silva E Mouta Junior S, de Moraes MT. Monitoring the emergence of HBV resistance mutations by HBV-RNA pyrosequencing[J]. Braz J Infect Dis, 2016, 20(2): 216-217.
[DOI]
|
| [14] |
Yu Y, Wang J, Li G, Shen C, Chen S, Huang J, Wang X, Zhao C, Huang Y, Wang B, Wang X, Zhang J, Qiu C, Zhang W. Nucleos(t)ide analogue interruption: alternative approach to intrahepatic set point for spontaneous control of HBV replication?[J]. J Hepatol, 2018, 68(3): 609-610.
[DOI]
|
| [15] |
鲁凤民, 王杰, 陈香梅, 江建宁, 张文宏, 赵景民, 任红, 侯金林, 夏宁邵. 乙型肝炎病毒RNA病毒颗粒的发现及其对抗病毒治疗临床实践的潜在影响[J]. 中华肝脏病杂志, 2017, 25(2): 105-110. [DOI]
|
| [16] |
Fan R, Zhou B, Xu M, Tan D, Niu J, Wang H, Ren H, Chen X, Wang M, Ning Q, Shi G, Sheng J, Tang H, Bai X, Liu S, Lu F, Peng J, Sun J, Xie Q, Hou J; Chronic Hepatitis B Study Consortium. Association between negative results from tests for HBV DNA and RNA and durability of response after discontinuation of nucleos(t)ide analogue therapy[J]. Clin Gastroenterol Hepatol, 2020, 18(3): 719-727.
[DOI]
|
| [17] |
Liu Y, Jiang M, Xue J, Yan H, Liang X. Serum HBV RNA quantification: useful for monitoring natural history of chronic hepatitis B infection[J]. BMC Gastroenterol, 2019, 19(1): 53.
[DOI]
|
| [18] |
Chen EQ, Wang ML, Tao YC, Wu DB, Liao J, He M, Tang H. Serum HBcrAg is better than HBV RNA and HBsAg in reflecting intrahepatic covalently closed circular DNA[J]. J Viral Hepat, 2019, 26(5): 586-595.
[DOI]
|
| [19] |
Gao M, Feng C, Ying R, Nie Y, Deng X, Zhu Y, Tang X, Guan Y, Hu F, Li F. A novel one-step quantitative reverse transcription PCR assay for selective amplification of hepatitis B virus pregenomic RNA from a mixture of HBV DNA and RNA in serum[J]. Arch Virol, 2019, 164(11): 2683-2690.
[DOI]
|
| [20] |
Wang J, Yu Y, Li G, Shen C, Li J, Chen S, Zhang X, Zhu M, Zheng J, Song Z, Wu J, Shao L, Meng Z, Wang X, Huang Y, Zhang J, Qiu C, Zhang W. Natural history of serum HBV-RNA in chronic HBV infection[J]. J Viral Hepat, 2018, 25(9): 1038-1047.
[DOI]
|
| [21] |
Liao H, Liu Y, Li X, Wang J, Chen X, Zou J, Li Q, Liu L, Wang J, Huang B, Lu F, Xu D. Monitoring of serum HBV RNA, HBcrAg, HBsAg and anti-HBc levels in patients during long-term nucleoside/nucleotide analogue therapy[J]. Antivir Ther, 2019, 24(2): 105-115.
[PubMed]
|
| [22] |
Wang J, Yu Y, Li G, Shen C, Meng Z, Zheng J, Jia Y, Chen S, Zhang X, Zhu M, Zheng J, Song Z, Wu J, Shao L, Qian P, Mao X, Wang X, Huang Y, Zhao C, Zhang J, Qiu C, Zhang W. Relationship between serum HBV-RNA levels and intrahepatic viral as well as histologic activity markers in entecavir-treated patients[J]. J Hepatol, 2018, 68(1): 16-24.
[DOI]
|
| [23] |
中华医学会感染病学分会, 中华医学会肝病学分会. 慢性乙型肝炎防治指南(2019年版)[J]. 中华肝脏病杂志, 2019, 27(12): 938-961. [DOI]
|
| [24] |
中华医学会肝病学分会, 中华医学会感染病学分会. 丙型肝炎防治指南(2019年版)[J]. 中华肝脏病杂志, 2019, 27(12): 962-979. [DOI]
|
| [25] |
European Association for the Study of the Liver. EASL Clinical Practice Guidelines on hepatitis E virus infection[J]. J Hepatol, 2018, 68(6): 1256-1271.
[DOI]
|
| [26] |
Limothai U, Chuaypen N, Poovorawan K, Chotiyaputta W, Tanwandee T, Poovorawan Y, Tangkijvanich P. Baseline and kinetics of serum hepatitis B virus RNA predict response to pegylated interferon-based therapy in patients with hepatitis B e antigen-negative chronic hepatitis B[J]. J Viral Hepat, 2019, 26(12): 1481-1488.
[DOI]
|
| [27] |
Carey I, Gersch J, Wang B, Moigboi C, Kuhns M, Cloherty G, Dusheiko G, Agarwal K. Pre-genomic HBV RNA and HBcrAg predict outcomes in HBeAg negative chronic hepatitis B patients suppressed on nucleos(t)ide analogue therapy [J/OL]. Hepatology, 2019. https://aasldpubs.onlinelibrary.wiley.com/doi/abs/10.1002/hep.31026.
|
| [28] |
Block TM, Gish R, Guo H, Mehta A, Cuconati A, Thomas London W, Guo JT. Chronic hepatitis B: what should be the goal for new therapies?[J]. Antiviral Res, 2013, 98(1): 27-34.
[DOI]
|
| [29] |
Butler EK, Gersch J, Mcnamara A, Luk KC, Holzmayer V, de Medina M, Schiff E, Kuhns M, Cloherty GA. Hepatitis B virus serum DNA and RNA levels in nucleos(t)ide analog-treated or untreated patients during chronic and acute infection[J]. Hepatology, 2018, 68(6): 2106-2117.
[DOI]
|
| [30] |
张海莹, 季颖, 朱凌, 饶慧瑛, 王江华, 张恒辉, 谢兴旺, 魏来. 两种HBV DNA检测试剂的比较[J]. 中华检验医学杂志, 2011, 34(5): 459-464. [DOI]
|
 2020, Vol. 15
2020, Vol. 15


