柯萨奇病毒B组(Coxsackievirus B,CVB)是小RNA病毒科(Picornaviridae)肠道病毒属成员,基因组为长7.4 kb的单股正链RNA(+ssRNA)[1],编码一个大分子前体蛋白,被自身编码的蛋白酶2APro、3CPro逐级切割,最终形成4个结构蛋白(VP1~VP4)和7个功能蛋白(2A、2B、2C、3A、3B、3C和3D)[2-4],其中,3D是病毒的RNA依赖性RNA聚合酶(RNA-dependent RNA polymerase,RdRp),为病毒复制的关键酶[5-6]。
CVB在复制传代过程中存在基因组不稳定现象。研究显示,在感染小鼠心脏组织中发现了5′端7~49个碱基缺失的CVB3基因组RNA,并证明缺失RNA与全长基因组的混合物可在宿主体内复制并持续存在[7]。本课题组前期的研究也显示,插入外源增强型绿色荧光蛋白(enhanced green fluorescent protein,eGFP)基因的CVB3-eGFP重组毒株在感染细胞后,病毒基因组RNA出现剪切和拼接[8]。由此推测CVB感染可能会产生不完整的基因组剪切片段,但目前尚无实验证据证明,因此CVB基因组剪切的序列特点和机制均不清楚。
cDNA末端快速扩增技术是基于聚合酶链反应(PCR)原理的cDNA末端片段扩增方法[9],可用于cDNA的3′和5′端扩增,其中5′ cDNA末端快速扩增(rapid amplification of cDNA ends,RACE, 5′ RACE)以cDNA为模板,利用3′端已知序列为引物,扩增获得第1条链后,在其5′端添加人工接头,以人工接头序列为5′引物,完成PCR扩增。本研究利用5′ RACE,从CVB感染细胞中扩增和克隆病毒基因组3′端片段,分析CVB基因组剪切序列的特点,为进一步研究CVB基因组的剪切和生物学意义提供实验依据。
1 材料和方法 1.1 材料HeLa细胞、CVB3 Woodruff毒株、3D抗体来自哈尔滨医科大学微生物学教研室。内参GAPDH抗体购于美国Proteintech公司,辣根过氧化物酶标记山羊抗兔IgG购于万类生物科技有限公司,CVB全基因cDNA克隆质粒pMKS1由J. Linsay Whitton教授(美国Scripps Research Institute)惠赠。PrimeScript RT reagent Kit with gDNA Eraser反转录试剂盒、SYBR Premix Ex Taq聚合酶、SMARTer RACE 5′/3′试剂盒、凝胶回收试剂盒与In-Fusion HD Cloning Kit购于日本TaKaRa公司,TRIzol和质粒提取试剂盒购自美国赛默飞公司,10% PAGE凝胶试剂盒购于美国EpiZyme公司,CDP-Star显色液购自哈尔滨欣科瑞有限公司,探针及引物合成由苏州金唯智生物科技有限公司完成,测序由北京睿博兴科生物技术有限公司完成。
1.2 方法 1.2.1 引物设计5′ RACE扩增的上游引物为通用引物,序列为5′-CTAATACGACTCACTATA-GGGCAAGCAGTGGTATCAACGCAGAGT-3′。为获得编码区完整的剪切片段,依据CVB3基因组3′非翻译区(untranslated regions,UTR)序列,经Gene Runner软件设计下游特异性引物。为便于后续In-fusion克隆,下游引物5′端引入特定碱基序列GATTACGCCAAGCTT。下游引物序列:5′-GATTACGCCAAGCTTTGCACCGTTGTCTAG-T T C G GTTAGTACAGTAGGGTTAAGCCAAT-3′。
1.2.2 感染细胞总RNA的提取将HeLa细胞接种于6孔板内(1×106/孔),37 ℃、5% CO2常规培养24 h; 当细胞生长融合度为80%~90%时,更换新鲜完全培养液; 实验孔加2 μL CVB3液(TCID50 = 10-4.75/0.1 mL); 孵育1 h后,换新鲜细胞培养液继续培养; 24 h后收取细胞沉淀。参照TRIzol说明书操作提取感染细胞总RNA。收集的感染细胞沉淀物中加入TRIzol试剂并混匀,加入氯仿进行相分离,取水相加入异丙醇沉淀RNA,经75%乙醇洗涤沉淀物,用焦碳酸二乙酯(diethypyrocarbonate,DEPC)水溶解获得总RNA。
1.2.3 通过5′ RACE及In-fusion克隆获取剪切片段序列RNA与通用引物混合,72 ℃孵育3 min,42 ℃冷却2 min,14 000×g离心10 s。加入DTT、dNTPs、SMARTer Ⅱ A寡核苷酸及反转录酶混匀,42 ℃孵育90 min,70 ℃加热10 min。加入10 mL Tricine-EDTA缓冲液稀释cDNA产物。以cDNA为模板,进行5′ RACE扩增。反应条件:94 ℃变性30 s,68 ℃退火30 s,72 ℃延伸3 min,共25个循环。凝胶电泳回收扩增产物,回收产物经In-fusion连接酶与线性化pUC19载体50 ℃孵育15 min; 连接产物转化大肠埃希菌Stellar感受态细胞后涂布于Amp+ LB固体培养基平板,37 ℃倒置过夜培养; 随机挑取单克隆菌落,接种于Amp+ LB液体培养基中,37 ℃过夜培养; 提取质粒,以pUC19质粒为对照筛选的阳性克隆,送北京睿博兴科公司测序,测序引物为通用引物M13F及M13R。
1.2.4 Northern印迹(Northern blot)杂交实验HeLa细胞感染病毒后分别于0、12、24 h收获细胞,提取RNA,用1.5%甲醛变性琼脂糖凝胶分离,变性RNA转印至阳性尼龙膜,固定后预杂交,再加入地高辛标记的探针杂交过夜,探针为DIG-UTP标记的病毒3D编码区一段200个核苷酸的片段。用含有0.1%十二烷基硫酸钠的0.1×枸橼酸钠盐水(saline-sodium citrate,SSC)洗膜,封闭液常温封闭1 h,加入碱性磷酸酶标记的地高辛抗体0.5 μL,常温孵育1 h,洗膜,加CDP-Star显色液显色。
1.2.5 实时定量PCR和蛋白质免疫印迹法收取CVB3感染24 h的HeLa细胞并提取RNA和蛋白,使用实时定量PCR(RT-qPCR)和蛋白质免疫印迹(Western blot)法对CVB3的表达进行检测。根据PrimeScript RT reagent Kit with gDNA Eraser和SYBR Premix Ex Taq试剂盒说明书,依据CVB3基因组5′ UTR设计并合成特异性引物,以GAPDH基因作为内参进行RT-qPCR检测,用2-ΔΔCt方法分析结果。Western印迹法检测蛋白:准备10% PAGE凝胶,以每孔25 μL蛋白量上样,预染蛋白相对分子质量标准上样量为10 μL。实验条件:120 V恒压电泳2 h; 300 mA恒流转膜60 min; 室温置封闭液中2 h; 将膜置于1:3 000稀释的3D抗体及GAPDH抗体中,室温孵育2 h; 用含吐温20的Tris-HCl缓冲盐溶液(Tris buffered saline with Tween 20,TBST)洗3次; 室温孵育辣根过氧化物酶标记的兔二抗2 h; TBST洗5次; 显色并记录结果。
1.2.6 基因序列分析及二级结构预测基因测序结果经BLAST、ApE及DNAMAN软件比对分析并绘制模式图。二级结构预测由RNAfold(http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi)在线完成。
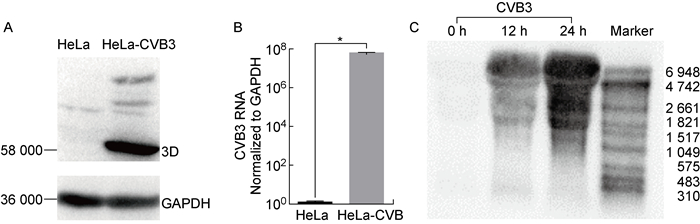
2 结果 2.1 CVB3基因组存在基因剪切片段HeLa细胞接种于6孔板,细胞密度为80%~90%时用CVB3液感染,24 h后收取细胞沉淀物,分别提取感染细胞总蛋白和总RNA。Western印迹检测到病毒3D蛋白条带,相对分子质量约为58 000(图 1A); RT-qPCR显示,CVB3 RNA高表达(P < 0.01),差异有统计学意义(图 1B); Northern印迹检测显示,在病毒感染后12、24 h,CVB3的基因组中出现剪切片段并且有固定的条带分布,非随机剪切的产物(图 1C)。
|
| A: Western blot for 3D protein in the HeLa cells infected with CVB3 for 24 h. B: RT-qPCR for CVB3 RNA in the HeLa cells infected with CVB3 at 24 h. *: P < 0.01, unpaired t test. C: Northern blot for CVB3 genomic RNA in the HeLa cells infected with CVB3 for 0, 12, 24 h. 图 1 CVB3基因组剪切片段的鉴定 Fig. 1 Identification of CVB3 genomic splicing fragments |
感染细胞总RNA通过去RNA酶反应和连接5′ RACE接头序列后,反转录得到cDNA模板,PCR扩增后进行凝胶电泳。结果显示,2 500~7 500 bp区域可见大小不同的多条扩增产物条带(图 2A)。依据课题组前期预测,回收2.0与7.5 kb之间所有的目的基因片段。

|
| A: The gel electrophoresis of the 5′ RACE product, recover fragments ranging from 2.0~7.5 kb and cloned into pUC19. M1: DL15000 marker; 1: CVB genomic fragments; M2: wide-range 10 000 molecular weight marker. B: The position of the spliced fragments correspond the CVB3 full-length genome, and the number of start and stop bases. 图 2 5′ RACE法扩增剪切片段及测序 Fig. 2 Amplification results of spliced fragments by 5′ RACE and sequencing |
凝胶电泳回收目的基因片段,连接于pUC19线性载体,随机挑取单克隆鉴定,对筛选出的20个阳性克隆进行测序。这些RNA片段长度在2 067~5 547 bp之间,与预期相符。BLAST和DNAMAN分析显示,所获得的片段为病毒基因组的3′片段。与完整病毒基因组相比,缺少长约793~5 144 bp的5′端序列。其中有5个片段的断端位于CVB3基因组的2Apro编码区,6个片段的断端位于CVB3基因组的2C编码区(图 2B)。除5′端基因缺失外,部分片段还出现序列丢失和拼接现象。如图 2B所示,20条片段基因序列中,有11条片段是CVB3基因组某一段的连续性基因片段,另9条为CVB3基因组非连续的拼接片段。
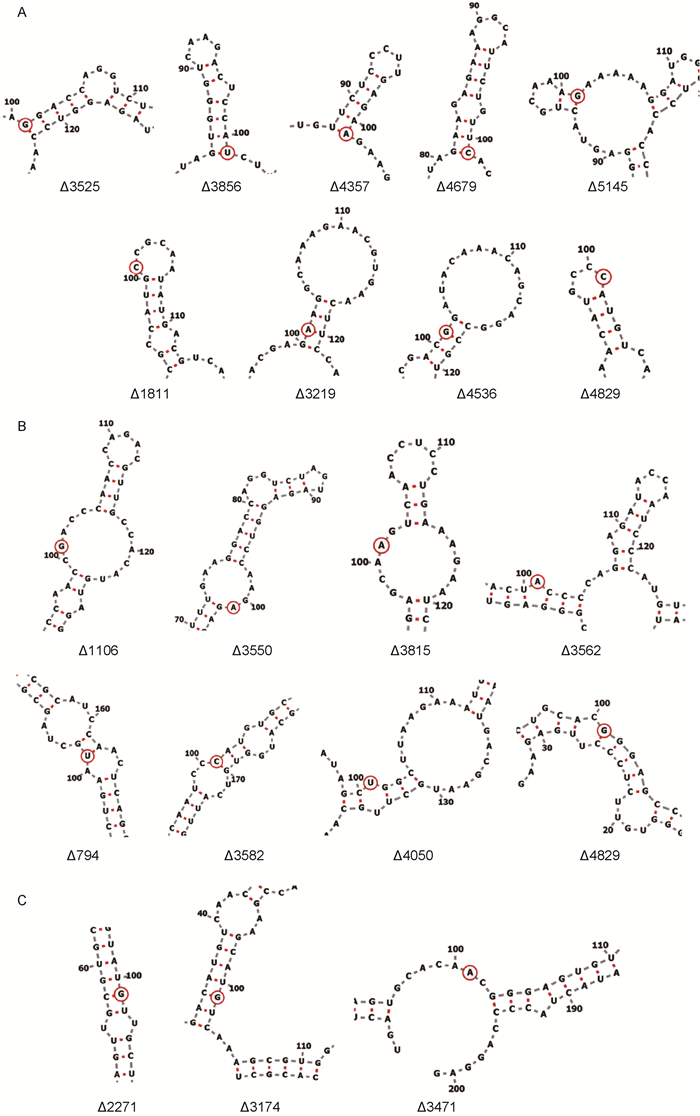
2.3 CVB3基因组剪切片段断端基因序列二级结构的预测以CVB3基因组为参照,分别选取20条片段的断端位点上下游各100 bp碱基,应用RNAfold预测其二级结构。结果显示,有9条剪切片段的断端序列位于发卡结构上(图 3A),有8条位于发卡结构附近或内环结构上(图 3B),还有3条剪切片段断端序列未形成特殊的二级结构(图 3C)。
|
| The secondary structure was predicted by selecting 100 bp around the starting position of the CVB genomic fragments (the position of the site was circled in red). A: The breakpoint sequence is located on the hairpin loop. B: The breakpoint sequence is near the hairpin structure or located on interior loop. C: No special structure. 图 3 剪切片段断端的二级结构预测 Fig. 3 Secondary structure prediction of cut ends |
CVB是人类病毒性心肌炎的主要病原体,可造成严重的心肌损伤及纤维化,为不可逆的扩张型心肌病的致病原因之一。CVB基因组的5′ UTR含有特殊的二级结构,介导病毒RNA与宿主核糖体结合,启动病毒蛋白翻译[10-11]。研究发现[12-13],在扩张型心肌病患者心脏组织中,CVB3基因组部分5′ UTR有多个碱基缺失,可能与其持续感染相关。本课题组的前期研究推测CVB基因组在感染细胞过程中存在不稳定现象[8]。
本研究通过Northern印迹实验结果发现,CVB3在感染细胞过程中可产生大量不完整的病毒RNA片段,5′ RACE扩增实验进一步证实了病毒基因组断裂片段的序列特点,支持此前关于CVB在细胞中存在病毒基因组不稳定现象的推测。
从获取的20条CVB3基因组片段分析可见,断裂片段的5′端起点不是随机分布,而是主要集中在2Apro和2C编码区。断裂片段中有11条为连续性序列,9条为非连续性拼接片段,提示断裂片段在细胞中经过了RNA编辑。因此,病毒基因组断裂并非是提取核酸时机械性破坏造成的结果。
RNA剪接是真核细胞mRNA成熟过程中重要的一环,这个剪切和拼接机制也能用于病毒基因组RNA[14]。有报道称,甲型流感病毒复制过程中可使用剪接的mRNA增加病毒复制所需相关蛋白的数量[15-16]; 人类免疫缺陷病毒基因组的转录本通过最佳剪接过程可产生40多种mRNA,用于产生前体蛋白或其他调节基因的单剪接mRNA[17]。研究表明,宿主细胞中的Dicer、AGO2等RNA酶可识别病毒基因组特定序列或特定的茎-环结构,产生小片段RNA并形成沉默复合物,靶向作用病毒RNA并降解[18-19]。本研究检测到的是CVB3基因组来源的长片段,对这些片段5′断点附近序列进行二级结构预测,结果提示多数断端位点可形成茎-环结构,局部形成双链RNA,可能成为Dicer、AGO2等细胞RNA酶的靶序列。断裂片段的产生机制有待后续实验阐明。
病毒基因组还可被剪切产生有功能的小片段[20-21],如Dicer可切割肠道病毒71型5′端序列,产生病毒源性小RNA(virus-derived small RNA,vsRNA),并可反向作用于病毒基因组,干扰病毒复制。本研究发现的CVB3基因组RNA断裂片段也可能被进一步剪切,形成有生物学活性的小片段。本课题组另一项研究显示,在CVB3感染的细胞中存在大量vsRNA,而且部分vsRNA可发挥miRNA样作用,影响病毒复制(结果另文发表)。
本研究是一项初步探索性工作,仅以HeLa细胞为例,并且在简化感染条件下观察CVB基因组的不稳定性,结果证实了我们的推测。CVB在感染细胞(尤其是心肌细胞)的过程中,病毒基因组的稳定性如何变化需要进一步实验观察。本研究的发现为进一步研究CVB基因组的剪切和生物学意义提供了实验依据。
| [1] |
Garmaroudi FS, Marchant D, Hendry R, Luo H, Yang D, Ye X, Shi J, McManus BM. Coxsackievirus B3 replication and pathogenesis[J]. Future Microbiol, 2015, 10(4): 629-653.
[DOI]
|
| [2] |
van der Linden L, Wolthers KC, van Kuppeveld FJ. Replication and inhibitors of enteroviruses and parechoviruses[J]. Viruses, 2015, 7(8): 4529-4562.
[DOI]
|
| [3] |
Castelló A, Alvarez E, Carrasco L. The multifaceted poliovirus 2A protease: regulation of gene expression by picornavirus proteases[J]. J Biomed Biotechnol, 2011, 2011: 369648.
[DOI]
|
| [4] |
Chase AJ, Semler BL. Viral subversion of host functions for picornavirus translation and RNA replication[J]. Future Virol, 2012, 7(2): 179-191.
[DOI]
|
| [5] |
Karr JP, Peersen OB. ATP is an allosteric inhibitor of coxsackievirus B3 polymerase[J]. Biochemistry, 2016, 55(28): 3995-4002.
[URI]
|
| [6] |
Ogram SA, Boone CD, McKenna R, Flanegan JB. Amiloride inhibits the initiation of coxsackievirus and poliovirus RNA replication by inhibiting VPg uridylylation[J]. Virology, 2014, 464-465: 87-97.
[DOI]
|
| [7] |
Kim KS, Tracy S, Tapprich W, Bailey J, Lee CK, Kim K, Barry WH, Chapman NM. 5'-terminal deletions occur in coxsackievirus B3 during replication in murine hearts and cardiac myocyte cultures and correlate with encapsidation of negative-strand viral RNA[J]. J Virol, 2005, 79(11): 7024-7041.
[DOI]
|
| [8] |
Tong L, Lin L, Zhao W, Wang B, Wu S, Liu H, Zhong X, Cui Y, Gu H, Zhang F, Zhong Z. Destabilization of coxsackievirus B3 genome integrated with enhanced green fluorescent protein gene[J]. Intervirology, 2011, 54(5): 268-275.
[DOI]
|
| [9] |
Ohara O, Dorit RL, Gilbert W. One-sided polymerase chain reaction: the amplification of cDNA[J]. Proc Natl Acad Sci USA, 1989, 86(15): 5673-5677.
[DOI]
|
| [10] |
Mahmud B, Horn CM, Tapprich WE. Structure of the 5' untranslated region of enteroviral genomic RNA[J]. J Virol, 2019, 93(23): e01288-19.
[DOI]
|
| [11] |
Dutkiewicz M, Stachowiak A, Swiatkowska A, Ciesiołka J. Structure and function of RNA elements present in enteroviral genomes[J]. Acta Biochim Pol, 2016, 63(4): 623-630.
[URI]
|
| [12] |
Lévêque N, Garcia M, Bouin A, Nguyen JHC, Tran GP, Andreoletti L, Semler BL. Functional consequences of RNA 5' terminal deletions on coxsackievirus B3 RNA replication and ribonucleoprotein complex formation[J]. J Virol, 2017, 91(16): e00423-17.
[DOI]
|
| [13] |
Bouin A, Gretteau PA, Wehbe M, Renois F, N'Guyen Y, Lévêque N, Vu MN, Tracy S, Chapman NM, Bruneval P, Fornes P, Semler BL, Andreoletti L. Enterovirus persistence in cardiac cells of patients with idiopathic dilated cardiomyopathy is linked to 5' terminal genomic RNA-deleted viral populations with viral-encoded proteinase activities[J]. Circulation, 2019, 139(20): 2326-2338.
[DOI]
|
| [14] |
Padgett RA, Hardy SF, Sharp PA. Splicing of adenovirus RNA in a cell-free transcription system[J]. Proc Natl Acad Sci USA, 1983, 80(17): 5230-5234.
[URI]
|
| [15] |
Wise HM, Hutchinson EC, Jagger BW, Stuart AD, Kang ZH, Robb N, Schwartzman LM, Kash JC, Fodor E, Firth AE, Gog JR, Taubenberger JK, Digard P. Identification of a novel splice variant form of the influenza A virus M2 ion channel with an antigenically distinct ectodomain[J]. PLoS Pathog, 2012, 8(11): e1002998.
[DOI]
|
| [16] |
Duriez M, Mandouri Y, Lekbaby B, Wang H, Schnuriger A, Redelsperger F, Guerrera CI, Lefevre M, Fauveau V, Ahodantin J, Quetier I, Chhuon C, Gourari S, Boissonnas A, Gill U, Kennedy P, Debzi N, Sitterlin D, Maini MK, Kremsdorf D, Soussan P. Alternative splicing of hepatitis B virus: A novel virus/host interaction altering liver immunity[J]. J Hepatol, 2017, 67(4): 687-699.
|
| [17] |
Stoltzfus CM. Chapter 1. Regulation of HIV-1 alternative RNA splicing and its role in virus replication[J]. Adv Virus Res, 2009, 74: 1-40.
[DOI]
|
| [18] |
穆宇松, 陈思佳, 张林月, 钟照华. AGO2蛋白的抗病毒应用[J]. 国际病毒学杂志, 2017, 24(3): 212-216. [URI]
|
| [19] |
Liu YP, Schopman NC, Berkhout B. Dicer-independent processing of short hairpin RNAs[J]. Nucleic Acids Res, 2013, 41(6): 3723-3733.
[DOI]
|
| [20] |
Lin JY, Brewer G, Li ML. HuR and Ago2 bind the internal ribosome entry site of enterovirus 71 and promote virus translation and replication[J]. PLoS One, 2015, 10(10): e0140291.
[DOI]
|
| [21] |
Weng KF, Hung CT, Hsieh PT, Li ML, Chen GW, Kung YA, Huang PN, Kuo RL, Chen LL, Lin JY, Wang RY, Chen SJ, Tang P, Horng JT, Huang HI, Wang JR, Ojcius DM, Brewer G, Shih SR. A cytoplasmic RNA virus generates functional viral small RNAs and regulates viral IRES activity in mammalian cells[J]. Nucleic Acids Res, 2014, 42(20): 12789-12805.
|
 2020, Vol. 15
2020, Vol. 15


