单纯疱疹病毒1型(herpes simplex virus type 1, HSV-1)属于疱疹病毒科α -疱疹病毒亚科,是一种具有囊膜的双链线性DNA病毒。HSV-1是一种嗜神经型病毒,通过上皮黏膜细胞感染后,一部分病毒通过裂解感染(lytic infection),引起角膜炎和口唇疱疹; 另一部分病毒则沿着感觉神经逆行到达腰背神经节,并潜伏在三叉神经节中,直到宿主免疫力下降时再重新开始复制,启动新一轮感染[1]。
DNA聚合酶在病毒感染过程中发挥着重要作用,也是抗病毒药物作用的关键靶位。HSV-1 DNA聚合酶由和UL42共同组成:UL30为催化亚基(catalytic subunit),具有聚合酶和3’-5’外切酶活性(校对活性),负责DNA的复制[2]; UL42为辅助亚基(accessory subunit),负责提高UL30的持续合成能力,以及UL30与DNA的连接[2-4]。这2个亚基在胞质内翻译成蛋白质并结合后,依赖核定位信号(nuclear location signal, NLS)共同转运至宿主细胞核中,并与其他病毒蛋白一同参与DNA的复制[5]。本文主要介绍辅助亚基UL42的结构特点、主要功能、作用机制及其在抗病毒药物开发中的研究进展,旨在为病毒致病机制的后续研究和抗病毒药物的研发提供参考。
1 UL42蛋白概述 1.1 UL42蛋白的结构特点HSV-1 UL42蛋白由488个氨基酸组成,是DNA聚合酶中的辅助亚基,也称为持续合成因子(processivity factor),在不同疱疹病毒中高度保守。UL42 N端1~340位氨基酸是主要功能域,这段肽段负责结合UL30以形成聚合酶全酶、结合DNA以辅助病毒基因组复制,促进UL30的持续合成[6]。图 1标注了不同功能域的位置,可见C端148个氨基酸的折叠很松散,不负责UL42的基本功能[6]。

|
| UL42 main activity domains include UL30-binding domain, UL30-activating domain and DNA-binding domain. It can also inhibit TNF-α activated NF-κB signal pathway, phosphorylation of IRF-3 and activity of DNA polymerase. In addition, UL42 contains a connector loop and one NLS. 图 1 UL42的主要功能结构域 Fig. 1 The main functional domains of UL42 protein |
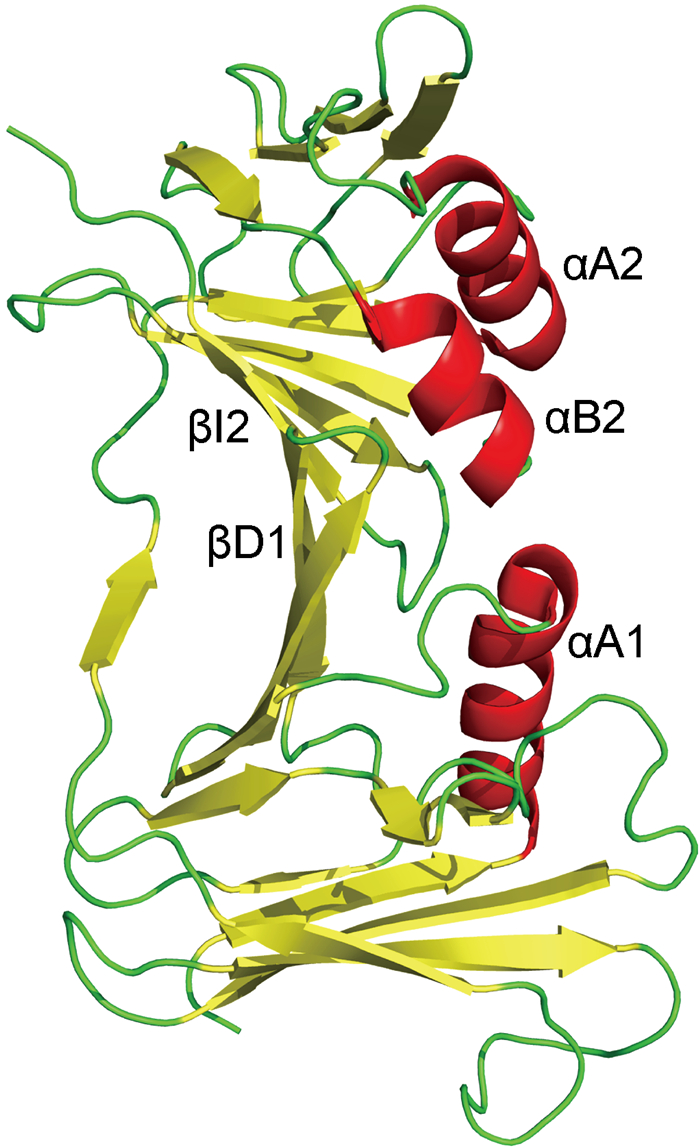
UL42在体内外均以单体形式存在[7],其结构与PCNA(增殖细胞核抗原)三聚体的单体结构高度相似。UL42由2个拓扑域构成,分别为29~160、175~319位氨基酸,2个拓扑域之间的旋转角度为40°,小于PCNA的60°旋转角度,因此UL42的结构更平面。每个拓扑域都含有2个反向平行的β折叠,各有一条支链βD1、βI2与对方非共价结合。两个β折叠之间的肽段(160~175位氨基酸,详见图 1)构成1个连接环。连接环背侧有3个α螺旋,αA1位于N端,αA2、αB1位于C端,具体如图 2所示。该表面富含的正电残基在α-疱疹病毒中高度保守,并与DNA的磷酸骨架结合,使得UL42在DNA链上滑动[8]。
|
| UL42 is composed by two antiparallel beta sheets, these two domains interact noncovalently through βD1 and βI2. The connector loop covalently connects the two domains. The opposite of the connector loop is three α helices: αA1、αA2、αB2. 图 2 UL42的三维结构图 Fig. 2 Three-dimensional structure of UL42 |
DNA病毒在宿主细胞质合成DNA聚合酶,转运至细胞核内启动病毒复制。研究显示,多种疱疹病毒聚合酶辅助蛋白(polymerase accessory protein,PAP)的C端都有功能性核定位信号NLSs[9-10]。
Loregian等[11]发现,UL30能够通过C端pol肽中的RRMLHR序列介导入核,该NLS是UL30唯一的NLS,属于非经典NLS。Alvisi等[5]发现,UL42 C端含有1个双分型NLS(NLSbip:PTTKRGRSGGEDARADALKKPK413),位于391~413位氨基酸,介导入核转运需要Ras相关核蛋白(Ran)、三磷酸腺苷(ATP)和内膜蛋白(IMP),是一种经典NLS。单独敲除UL30或UL42的NLS并不影响HSV-1 DNA聚合酶入核,但同时敲除二者则使聚合酶失去入核能力,说明DNA聚合酶先在细胞质内形成复合体,再借助2个亚基的NLS转运进入细胞核,而UL42很可能是UL30的细胞核协同转运蛋白[5]。图 1中标注了UL42 NLS在氨基酸链的位置。
1.3 UL42蛋白对病毒复制的影响目前,HSV-1 DNA的复制机制尚不完全明确,有证据表明,有7种病毒蛋白负责HSV-1 DNA的复制,包括由UL30和UL42组成的DNA聚合酶,单链DNA结合蛋白细胞感染多肽8(cell infection polypeptides 8,ICP8),由UL5、UL8、UL52组成的解旋酶-引物酶复合体,以及序列特异性的复制起始位点结合蛋白UL9[12]。其中最重要的是DNA聚合酶。
ICP8既是经典的单链DNA结合蛋白,也是单链退火蛋白,对病毒感染时的DNA复制至关重要[13]。由UL5、UL8、UL52组成的解旋酶-引物酶复合体在复制叉上将dsDNA解链,并启动滞后链的合成,但效率很低。UL30-UL42复合物与解旋酶结合能提高后者的解旋活性[14]。UL9具有部分ssDNA的3’-5’DNA解旋酶活性[15],并能够特异性地与任意复制起始位点结合,启动DNA的复制。UL30的酶活性可以催化DNA的合成,而UL42能够将UL30与DNA模板链结合,提高全酶的持续合成能力[2]。二者构成HSV-1 DNA聚合酶全酶,在HSV-1 DNA合成与病毒复制过程中发挥了至关重要的作用。
2 UL42蛋白辅助病毒复制的分子机制 2.1 UL42与UL30的结合机制UL30 1200~1235位氨基酸形成的α螺旋是主要的UL42结合位点,而UL42 N端三分之二的区域能够在体内外与UL30结合并发挥作用[6]。UL42与UL30单纯的物理结合并不能提高其催化能力[16],说明两者的相互作用更为复杂。Hamatake等[17]用胰蛋白酶消化UL42时,发现两种富含精氨酸与赖氨酸的片段,在dsDNA存在时不被胰蛋白酶消化,其相对分子质量分别为2.8×104和8×103。限制性酶切发现,2.8×104片段为1~245位或1~254位氨基酸,8×103片段起始于255位氨基酸,可能终止于319、340或370位氨基酸。2.8×104和8×103混合物能够与UL30结合并激活其酶活性,且两者无法通过纯化分离,说明其共存时相互连接,并与UL30结合。这2个片段可能包含了UL42的UL30结合域的最小范围。
Zuccola等[8]研究发现,UL42与UL30之间有多种作用形式,两个蛋白的主链上,UL30 A1208的羰基氧和UL42 V296的氮连接,UL30主链的A1235、L1234之间的氢键与UL42 K289侧链的氨基发生作用。两蛋白的侧链之间也形成多个氢键连接,包括UL30 R1224、D1232、H1228与UL42 D63、R64之间的3个氢键,以及R1229、Q171、E1211三者之间形成的氢键网络。UL42连接环的165~169位氨基酸与UL30的1211~1216位氨基酸形成反向平行的氢键。UL30的H1228、R1229与UL42的R64、Q171是2个蛋白相互作用的关键结合位点。此外,破坏UL42 E161和R134之间的盐桥可能会影响UL42与UL30的结合。图 1中标注了部分与UL30结合相关的位点或肽段。
2.2 UL42促进UL30持续合成能力的机制UL42作为DNA聚合酶中的持续合成因子,与UL30结合后可以激活其持续合成病毒基因组的能力。研究表明,UL30 C端227个氨基酸负责与UL42功能性相互作用[18]。Monahan等[19]为了确定UL42中同时与UL30物理及功能性相互作用的区域,构建了一系列突变体。他们发现,d129~163和d202~337两个突变体不能激活UL30,说明这2个区域是UL42激活UL30的功能域。其中129~163区域为螺旋-卷曲-螺旋结构,不影响UL42与UL30的物理结合,其中的157位氨基酸对UL42的功能非常重要。202~337区域可能不止一个功能域,然而由于实验灵敏度不足,尚未确定该区域是否也与UL30-UL42的物理结合相关。图 1中标注了以上两个区域在氨基酸链上所处的位置。
研究表明,UL42对dsDNA具有高度亲和力,而UL30对ssDNA的亲和力更高[20-21]。Weisshart等[22]发现,UL42通过提高UL30对dsDNA的特异性结合,以减少全酶从模板链上解离。Gottlieb等[23]利用Tsurimoto和Stillman[24]制备的引物模板模型,发现当UL42与UL30结合后,全酶对引物模板链3’端的亲和力增加了10倍。他们认为,UL42在全酶内作为滑动夹子,通过增加UL30与引物模板链3’端的亲和力来提高其持续合成能力。Chaudhuri和Parris[25]发现,UL42对引物模板的亲和力与稳定性随着离子强度的增加而降低,而UL30-UL42复合物对离子强度变化不敏感,说明UL42并非仅仅通过与UL30结合而提高其持续合成能力。他们猜测,UL42可能和T7 DNA聚合酶[26]一样,UL30 C端与UL42结合后,其无序的构象会发生变化,可能会在DNA周围形成更紧密的构象,导致解离率降低。
基于Gottlieb和Challberg[23]的猜想,Chow和Coen[27]提出了如下假设:不同于其他持续合成因子,UL42与DNA的结合能促进UL30的活性。为了证明此猜想,他们试图找到只影响DNA结合活性、不影响UL30结合活性的UL42突变体。结果表明,插入突变体I-203和I-206不能与dsDNA结合、无法刺激UL30,却能与UL30形成免疫共沉淀,保留了DNA聚合酶的基本功能,证明了UL42在体内外增强UL30的持续合成能力需要DNA结合活性。
Zuccola等[8]建立了UL42/Pol/DNA的模型,发现在UL42与DNA结合的正电表面有5个保守的残基可能与DNA结合有关,分别为R113、R182、R279、R280、Q282。Randell等[28]用Ala替代4个保守的Arg,构建出4个单突变UL42,发现UL42与DNA的结合受到了影响。虽然UL42与UL30的结合并不受影响,但长链DNA的合成却随之减少,说明UL42结合DNA与结合UL30的能力是相互独立的,但UL42结合DNA的能力的确能够影响UL30合成长链DNA的能力。图 1中标注了与DNA结合有关的所有位点。
2.3 UL42在DNA链上的移动机制在DNA复制过程中,前导链需要持续合成因子,以保证每次催化后,DNA聚合酶不会从模板链上解离。持续合成因子通过与催化亚基和DNA链相互作用,增加全酶的作用时间,例如滑动夹子模型。许多持续合成因子都通过形成滑动夹子环绕在DNA上,在不影响合成速度的同时促进DNA合成,包括PCNA、E.coli Pol Ⅲ的β亚基、T4噬菌体等。滑动夹子并没有DNA亲和力,与DNA的结合依赖于夹子载体和ATP[29]。
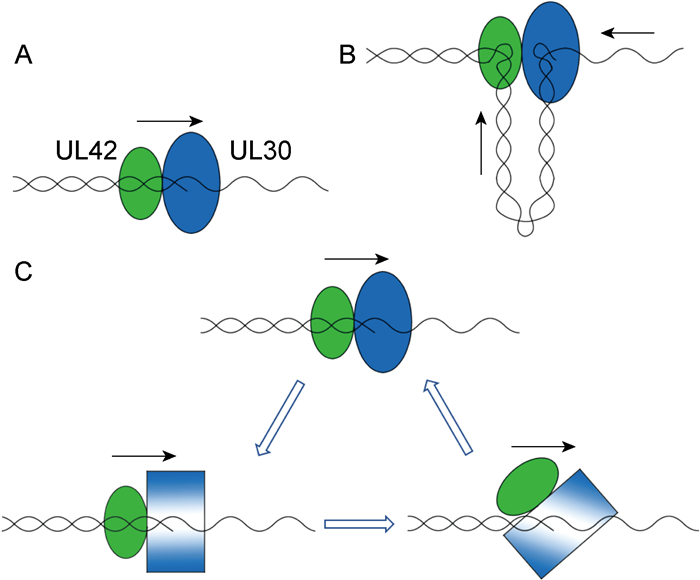
Weisshart等[22]认为,UL30-UL42复合物与DNA稳定结合的同时,并不会降低DNA的合成速度,因此其移动机制必定与滑动夹子不同。他讨论了ratcheting模型和sitting模型(由Thomas J. Kelly提出)两种模型,用于解释UL30-UL42复合物在模板链上的移动机制。在ratcheting模型中,催化导致酶发生了构象变化,与模板链特异性强的构象占主导地位,同时UL42的DNA结合活性下降,便于聚合酶在模板链上移动。T4 DNA聚合酶是此模型的典型代表。而在sitting模型中,UL42始终结合在模板链的初始结合位点,DNA链穿过UL30进入UL42,并被挤出。这种模型与T7RNA聚合酶的scrunching模型非常相似[30]。但Randell等[29]发现,UL42能在保持高亲和性的同时沿着DNA滑动,这与sitting模型相反,反而能够解释ratcheting模型。图 3为3种移动模型的示意图。
|
| A. Sliding clamp model: lacking in DNA affinity and depending on clamp loader and ATP. B. Sitting model: UL42 remains at the initial binding site. After being squeezed out of UL30, DNA enters UL42. C. Ratcheting model: the catalytic activity causes a conformational change on UL30, and binds to DNA with a template-specific conformation. UL42 DNA-binding activity decreases, and thus the polymerase moves alone the template. 图 3 UL42在DNA链上的3种滑动机制 Fig. 3 Three sliding mechanisms of UL42 along DNA chain |
Zuccola等[8]综合了以上观点,提出了如下猜想:UL42通过静电作用与DNA结合并滑动。UL42连接环的背侧表面为正电,可与DNA的负电磷酸骨架发生静电相互作用,使得UL42沿着其骨架滑动。不与UL30结合时,UL42只能进行单向、随机的移动; 与UL30结合后,两者的复合物获得了动力和方向。
3 UL42抑制宿主天然免疫通路的分子机制NF-κB是一类转录因子,在炎症、免疫、细胞增殖、分化和生长等细胞活动中发挥重要作用,会影响多种基因的表达[31],是重要的免疫因子。TNF-α是固有免疫中重要的多功能细胞因子,能够诱导免疫应答中多种基因的表达,可经一系列反应激活NF-κB[32-33]。Zhang等[34]发现,UL42通过其N端(1~390位氨基酸)结合p65和p50,阻碍后者的核转移,抑制TNF-α激活的NF-κB信号通路。R113、R279和R280是UL42与DNA结合的关键残基[28],并位于UL42 N端,可能与NF-κB信号通路的抑制有关。突变UL42的3个DNA结合位点(R113、R279和R280)使UL42失去阻碍p65和p50核转移的能力,抑制NF-κB信号通路激活的作用也有所下降,表明这3个保守的Arg在UL42抑制NF-κB信号通路激活中具有重要作用。此外,UL42还可以抑制NF-κB诱导激酶(NF-κB inducing kinase, NIK)和NF-κB激酶抑制剂α (inhibitor of nuclear factor kappa-B kinase, IKK α)诱导的NF-κB启动子活性,提示UL42还可能抑制非经典NF-κB信号通路[34]。图 1中标注了抑制TNF-α激活的NF-κB信号通路有关的肽段。
Chapon等[35]建立了HSV-1基因组的转座子插入突变体库,发现UL42能够调控干扰素(interferon,IFN)调控因子3(IRF-3)的功能。UL42特异性抑制IRF-3磷酸化,进而下调IFN-β的转录。R279-280A突变体既不能影响IFN-β的激活,也不能与IRF-3结合,表明这2个Arg在抑制IFN-Ⅰ产生中也具有关键作用。图 1中标注了这2个位点在氨基酸链上的位置。
4 UL42在抗病毒药物开发中的研究进展Owsianka等[36]根据UL42的氨基酸序列,合成了96个十五肽,以检测UL42是否含有可以抑制DNA聚合酶活性的肽段,并得到了5个十五肽,分别为23~38、64~78、89~102、229~243、279~293位氨基酸。后4个肽段可能通过与DNA结合发挥其抑制作用,而第1个肽段的抑制机制有待进一步研究。但遗憾的是,这5个肽段特异性有限,并不适合作为抗病毒药物研发的先导化合物。图 1中标注了这5个肽段的位置。
RNA沉默是脊椎动物常见的抗病毒机制,可由RNA干扰(RNA interference, RNAi)诱导产生,导致靶mRNA特异性降解[37]。Mollaei等[37]设计了2种靶向HSV-1 UL42 mRNA的siRNA,转染siRNA的HeLa细胞形成的HSV-1病毒空斑显著减少,同时未发现细胞病变。Real-time PCR结果显示,siRNA能够阻断UL42基因表达,显著抑制HSV-1 DNA的复制,提示RNAi可以作为抑制HSV-1的有效手段,并且UL42特异性siRNA不会对宿主细胞造成伤害。
阿昔洛韦是首个用于治疗HSV的药物。此外,还有其前体药物万乃洛韦、喷昔洛韦及其前体药物泛昔洛韦,这4种药物的磷酸化形式均可作为病毒DNA聚合酶竞争性抑制剂,以抑制DNA合成[38-39]。然而,由于这类药物生物活性较弱、半衰期短,加之HSV能够长期潜伏在神经节内,并不能阻止HSV复发。鉴于以上情况,研究HSV DNA复制相关因子的功能与结构,对于研发长期有效的抗病毒药物具有启发作用。
目前,有效阻断或干扰蛋白之间相互作用的抑制剂成为研究热点。HSV-1的DNA聚合酶2个亚基的相互作用为抗病毒研究提供了一个有力靶位。Liuzzi等[40]设计了一种HSV RNR抑制剂,命名为BILD 1263,通过抑制RNR亚基之间的结合来抑制HSV-1、HSV-2及耐阿昔洛韦HSV的体内复制。Loregian等[11]发现,利用UL30 C端27个氨基酸可以破坏HSV-1 UL30与UL42之间的相互作用,病毒复制也无法进行。以上证据为研发抗HSV-1药物提供了新的研究方向。
5 HSV-1 UL42同源蛋白的研究进展作为疱疹病毒复制过程中重要的辅助亚基,HSV-1 UL42拥有多种同源蛋白,包括PRV(pseudorabies virus)UL42、HCMV(human cytomegalovirus)UL44、KSHV(Kaposi’s sarcoma-associated herpesvirus)PF-8、HHV-7(human herpes virus-7)UL27、EBV(Epstein-Barr virus) BMRF1等。这些蛋白无论是在结构还是功能上,都具有一定的相似性。对UL42的结构功能研究,能够启发对其他同源蛋白的探索。
伪狂犬病毒(Pseudorabies virus, PRV)的DNA聚合酶同样由UL30和UL42蛋白组成,其UL42含有384个氨基酸。与HSV-1相同,PRV UL42能够单独与UL30相互作用,并提高其复制能力[41]; 可通过单体形式与dsDNA结合[42]。PRV UL42含有2个单分型NLS和1个双分型NLS,只有双分型NLS(354~370位氨基酸)真正具有核定位作用,因此PRV UL30只有在UL42存在时才能转运入核[43]。使用siRNA敲低UL42能够显著抑制PRV复制,但尚不清楚UL42对病毒复制的作用与其核定位功能之间是否具有相关性[44]。
人巨细胞病毒(human cytomegalovirus,HCMV)的DNA聚合酶由催化亚基UL54和持续合成因子UL44组成。HCMV的UL44含有433个氨基酸,156~290位氨基酸为DNA结合域。UL44能以二聚体或单体形式与dsDNA高亲和性结合,其结合不依赖于滑动夹子和ATP[45]。UL44背侧面上有一凸出的可动环(flexible loop),是结合宿主细胞核与病毒DNA的必需片段,但不涉及与UL54的结合[46]。UL44的N端折叠形成头对头的同源二聚体,称为C形夹子[45]。借助背侧面和C形夹子形成的间隙环,UL44可以环绕在DNA上[47]。未二聚化的UL44无法支持DNA的持续合成,但并不影响与UL54的相互作用及入核[48]。HCMV UL44仅有一个NLS,位于425~431位氨基酸[10]。
卡波氏肉瘤病毒(Kaposi’s sarcoma-associated herpesvirus,KSHV)的持续合成因子为PF-8,与dsDNA具有亲和力,与催化亚基Pol-8结合后促进其复制能力[49]。PF-8与HCMV UL44同样能够形成头对头的同源二聚体,但前者缺少间隙环,无法环绕DNA,提示两者的DNA结合机制可能不同[50]。在此过程中,10~27位氨基酸负责与Pol-8相互作用,N端和279~301位氨基酸负责结合dsDNA,共同提高Pol的合成能力[51]。而与HSV-1 UL42一样,PF-8的C端与主要功能无关。1~21位、277~304位氨基酸是实现二聚化的重要片段[52]。PF-8通过C端的NLS(369~377位氨基酸),并通过N端或C端某一肽段与Pol-8结合,来介导Pol-8入核[9]。
6 小结HSV-1的潜伏感染和反复发作,对公共卫生造成了严重影响。UL42作为HSV-1重要的DNA聚合酶组成亚基,具有调控病毒DNA复制和拮抗宿主天然免疫反应的双重作用,但仍须进一步的机制研究。图 1总结了UL42的主要功能域及其分布情况。考虑到结构和功能的相似性,对HSV-1 UL42同源蛋白的研究可为其机制的探索提供基础。未来的研究应集中于对UL42参与生理和病理过程分子机制的阐述,以及筛选能够与UL42 DNA特异性结合域互补的抗病毒药物。对UL42蛋白功能和作用机制的进一步阐释,将为理解疱疹病毒在细胞和宿主体内的生命周期、病原和宿主细胞的相互作用以及特异性抗病毒药物的开发提供重要的参考数据。
| [1] |
Shen Y, Nemunaitis J. Herpes simplex virus 1 (HSV-1) for cancer treatment[J]. Cancer Gene Ther, 2006, 13(11): 975-992.
[DOI]
|
| [2] |
Berthomme H, Monahan SJ, Parris DS, Jacquemont B, Epstein AL. Cloning, sequencing, and functional characterization of the two subunits of the pseudorabies virus DNA polymerase holoenzyme: evidence for specificity of interaction[J]. J Virol, 1995, 69(5): 2811-2818.
[DOI]
|
| [3] |
Gallo ML, Dorsky DI, Crumpacker CS, Parris DS. The essential 65-kilodalton DNA-binding protein of herpes simplex virus stimulates the virus-encoded DNA polymerase[J]. J Virol, 1989, 63(12): 5023-5029.
[DOI]
|
| [4] |
Gottlieb J, Marcy AI, Coen DM, Challberg MD. The herpes simplex virus type 1 UL42 gene product: a subunit of DNA polymerase that functions to increase processivity[J]. J Virol, 1990, 64(12): 5976-5987.
[DOI]
|
| [5] |
Alvisi G, Avanzi S, Musiani D, Camozzi D, Leoni V, Ly-Huynh JD, Ripalti A. Nuclear import of HSV-1 DNA polymerase processivity factor UL42 is mediated by a C-terminally located bipartite nuclear localization signal[J]. Biochemistry, 2008, 47(52): 13764-13777.
[DOI]
|
| [6] |
Digard P, Chow CS, Pirrit L, Coen DM. Functional analysis of the herpes simplex virus UL42 protein[J]. J Virol, 1993, 67(3): 1159-1168.
[DOI]
|
| [7] |
Randell JCW, Coen DM. The herpes simplex virus processivity factor, UL42, binds DNA as a monomer[J]. J Mol Biol, 2004, 335(2): 409-413.
[DOI]
|
| [8] |
Zuccola HJ, Filman DJ, Coen DM, Hogle JM. The crystal structure of an unusual processivity factor, herpes simplex virus UL42, Bound to the C terminus of its cognate polymerase[J]. Mol Cell, 2000, 5(2): 267-278.
[DOI]
|
| [9] |
Chen Y, Ciustea M, Ricciardi RP. Processivity factor of KSHV contains a nuclear localization signal and binding domains for transporting viral DNA polymerase into the nucleus[J]. Virology, 2005, 340(2): 183-191.
[DOI]
|
| [10] |
Alvisi G, Jans DA, Guo J, Pinna LA, Ripalti A. A protein kinase CK2 site flanking the nuclear targeting signal enhances nuclear transport of human cytomegalovirus ppUL44[J]. Traffic, 2005, 6(11): 1002-1013.
[DOI]
|
| [11] |
Loregian A, Piaia E, Cancellotti E, Papini E, Marsden HS, Palù G. The catalytic subunit of herpes simplex virus type 1 DNA polymerase contains a nuclear localization signal in the UL42-binding region[J]. Virology, 2000, 273(1): 139-148.
[DOI]
|
| [12] |
Tenney DJ, Hurlburt WW, Bifano M, Stevens JT, Micheletti PA, Hamatake RK, Cordingley MG. Deletions of the carboxy terminus of herpes simplex virus type 1 UL42 define a conserved amino-terminal functional domain[J]. J Virol, 1993, 67(4): 1959-1966.
[DOI]
|
| [13] |
Weerasooriya S, Discipio KA, Darwish AS, Bai P, Weller SK. Herpes simplex virus 1 ICP8 mutant lacking annealing activity is deficient for viral DNA replication[J]. Proc Natl Acad Sci U S A, 2019, 116(3): 1033-1042.
[DOI]
|
| [14] |
Dickerson SM, Kuchta RD. Protein displacement by herpes helicase-primase and the key role of UL42 during helicase-coupled DNA synthesis by the herpes polymerase[J]. Biochemistry, 2017, 56(21): 2651-2662.
[DOI]
|
| [15] |
Marintcheva B, Weller SK. A tale of two HSV-1 helicases: roles of phage and animal virus helicases in DNA replication and recombination[J]. Prog Nucleic Acid Res Mol Biol, 2001, 70: 77-118.
[PubMed]
|
| [16] |
Digard P, Bebrin WR, Weisshart K, Coen DM. The extreme C terminus of herpes simplex virus DNA polymerase is crucial for functional interaction with processivity factor UL42 and for viral replication[J]. J Virol, 1993, 67(1): 398-406.
[DOI]
|
| [17] |
Hamatake R, Bifano M, Tenney D, Hurlburt WW, Cordingley MG. The herpes simplex virus type 1 DNA polymerase accessory protein, UL42, contains a functional protease-resistant domain[J]. J Gen Virol, 1993, 74(Pt 10): 2181-2189.
[PubMed]
|
| [18] |
Digard P, Coen DM. A novel functional domain of an α-like DNA polymerase: the binding site on the herpes simplex virus polymerase for the viral UL42 protein[J]. J Biol Chem, 1990, 265: 17393-17396.
[DOI]
|
| [19] |
Monahan SJ, Barlam TF, Crumpacker CS, Parris DS. Two regions of the herpes simplex virus type 1 UL42 protein are required for its functional interaction with the viral DNA polymerase[J]. J Virol, 1993, 67(10): 5922-5931.
[DOI]
|
| [20] |
Gallo ML, Jackwood DH, Murphy M, Marsden HS, Parris D. Purification of the herpes simplex virus type 1 65-kilodalton DNA-binding protein: properties of the protein and evidence of its association with the virus-encoded DNA polymerase[J]. J Virol, 1988, 62(8): 2874-2883.
[DOI]
|
| [21] |
Vaughan PJ, Purifoy DJ, Powell KL. DNA-binding protein associated with herpes simplex virus DNA polymerase[J]. J Virol, 1985, 53(2): 501-508.
[DOI]
|
| [22] |
Weisshart K, Chow CS, Coen DM. Herpes simplex virus processivity factor UL42 imparts increased DNA-binding specificity to the viral DNA polymerase and decreased dissociation from primer-template without reducing the elongation rate[J]. J Virol, 1999, 73(1): 55-66.
[DOI]
|
| [23] |
Gottlieb J, Challberg MD. Interaction of herpes simplex virus type 1 DNA polymerase and the UL42 accessory protein with a model primer template[J]. J Virol, 1994, 68(8): 4937-4945.
[DOI]
|
| [24] |
Tsurimoto T, Stillman B. Replication factors required for SV40 DNA replication in vitro: Ⅱ. Switching of DNA polymerase α and δ during initiation of leading and lagging strand synthesis[J]. J Biol Chem, 1991, 266: 1961-1968.
[DOI]
|
| [25] |
Chaudhuri M, Parris DS. Evidence against a simple tethering model for enhancement of herpes simplex virus DNA polymerase processivity by accessory protein UL42[J]. J Virol, 2002, 76(20): 10270-10281.
[DOI]
|
| [26] |
Doublié S, Tabor S, Long AM, Richardson CC, Ellenberger T. Crystal structure of a bacteriophage T7 DNA replication complex at 2.2Å resolution[J]. Nature, 1998, 391(6664): 251-258.
[DOI]
|
| [27] |
Chow CS, Coen DM. Mutations that specifically impair the DNA binding activity of the herpes simplex virus protein UL42[J]. J Virol, 1995, 69(11): 6965-6971.
[DOI]
|
| [28] |
Randell JCW, Komazin G, Jiang C, Hwang CB, Coen DM. Effects of substitutions of arginine residues on the basic surface of herpes simplex virus UL42 support a role for DNA binding in processive DNA synthesis[J]. J Virol, 2005, 79(18): 12025-12034.
[DOI]
|
| [29] |
Randell JC, Coen DM. Linear diffusion on DNA despite high-affinity binding by a DNA polymerase processivity factor[J]. Mol Cell, 2001, 8(4): 911-920.
[DOI]
|
| [30] |
Cheetham GM, Steitz TA. Structure of a transcribing T7 RNA polymerase initiation complex[J]. Science, 1999, 286(5448): 2305-2309.
[DOI]
|
| [31] |
Oeckinghaus A, Ghosh S. The NF-kappaB family of transcription factors and its regulation[J]. Cold Spring Harb Perspect Biol, 2009, 1(4): a000034.
[DOI]
|
| [32] |
Nennig SE, Schank JR. The role of NFkB in drug addiction: beyond inflammation[J]. Alcohol Alcohol, 2017, 52(2): 172-179.
[DOI]
|
| [33] |
Li J, Li J, Yue Y, Hu Y, Cheng W, Liu R, Pan X, Zhang P. Genistein suppresses tumor necrosis factor alpha-induced inflammation via modulating reactive oxygen species/Akt/nuclear factor kappaB and adenosine monophosphate-activated protein kinase signal pathways in human synoviocyte MH7A cells[J]. Drug Des Devel Ther, 2014, 8: 315-323.
[PubMed]
|
| [34] |
Zhang J, Wang S, Wang K, Zheng C. Herpes simplex virus 1 DNA polymerase processivity factor UL42 inhibits TNF-α-induced NF-κB activation by interacting with p65/RelA and p50/NF-κB1[J]. Med Microbiol Immunol, 2013, 202(4): 313-325.
[DOI]
|
| [35] |
Chapon M, Parvatiyar K, Aliyari SR, Zhao JS, Cheng G. Comprehensive mutagenesis of herpes simplex virus 1 genome identifies UL42 as an inhibitor of type I interferon induction[J]. J Virol, 2019, 93(23): e01446-19.
[DOI]
|
| [36] |
Owsianka AM, Hart G, Murphy M, Gottlieb J, Boehme R, Challberg M, Marsden HS. Inhibition of herpes simplex virus type 1 DNA polymerase activity by peptides from the UL42 accessory protein is largely nonspecific[J]. J Virol, 1993, 67(1): 258-264.
[DOI]
|
| [37] |
Mollaei HR, Monavari SH, Arabzadeh S, Shahrabadi MS, Fazlalipour M. Antiviral activity of sirna UL42 against herpes simplex virus type 1 in HeLa cell culture[J]. J Antivir Antiretrovir, 2014, 6(3): 114-119.
|
| [38] |
Reardon JE, Spector T. Herpes simplex virus type 1 DNA polymerase. Mechanism of inhibition by acyclovir triphosphate[J]. J Biol Chem, 1989, 264(13): 7405-7411.
[DOI]
|
| [39] |
Schmid-Wendtner MH, Korting HC. Penciclovir cream-improved topical treatment for herpes simplex infections[J]. Skin Pharmacol Physiol, 2004, 17(5): 214-218.
[DOI]
|
| [40] |
Liuzzi M, Déziel R, Moss N, Beaulieu P, Bonneau AM, Bousquet C, Chafouleas JG, Garneau M, Jaramillo J, Krogsrud RL. A potent peptidomimetic inhibitor of HSV ribonucleotide reductase with antiviral activity in vivo[J]. Nature, 1994, 372(6507): 695-698.
[DOI]
|
| [41] |
Wang YP, Du WJ, Huang LP, Wei YW, Wu HL, Feng L, Liu CM. The pseudorabies virus dna polymerase accessory subunit UL42 directs nuclear transport of the holoenzyme[J]. Front Microbiol, 2016, 7: 124.
[DOI]
|
| [42] |
Wang Y-P, Huang L-P, Du W-J, Wei YW, Xia DL, Wu HL, Feng L, Liu CM. The pseudorabies virus DNA polymerase processivity factor UL42 exists as a monomer in vitro and in vivo[J]. Arch Virol, 2016, 161(4): 1027-1031.
[DOI]
|
| [43] |
王一平. 伪狂犬病病毒DNA聚合酶入核转运的分子机制[D]. 北京: 中国农业科学院, 2016.
|
| [44] |
Wang YP, Huang LP, Du WJ, Wei YW, Wu HL, Feng L, Liu CM. Targeting the pseudorabies virus DNA polymerase processivity factor UL42 by RNA interference efficiently inhibits viral replication[J]. Antiviral Res, 2016, 132: 219-224.
[DOI]
|
| [45] |
Appleton BA, Loregian A, Filman DJ, Coen DM, Hogle JM. The cytomegalovirus DNA polymerase subunit UL44 forms a C clamp-shaped dimer[J]. Mol Cell, 2004, 15(2): 233-244.
[DOI]
|
| [46] |
Alvisi G, Roth DM, Camozzi D, Pari GS, Loregian A, Ripalti A, Jans DA. The flexible loop of the human cytomegalovirus DNA polymerase processivity factor ppUL44 is required for efficient DNA binding and replication in cells[J]. J Virol, 2009, 83(18): 9567-9576.
[DOI]
|
| [47] |
Komazin-Meredith G, Petrella RJ, Santos WL, Filman DJ, Hogle JM, Verdine GL, Karplus M, Coen DM. The human cytomegalovirus UL44 C clamp wraps around DNA[J]. Structure, 2008, 16(8): 1214-1225.
[DOI]
|
| [48] |
Sinigalia E, Alvisi G, Mercorelli B, Coen DM, Pari GS, Jans DA, Ripalti A, Palù G, Loregian A. Role of homodimerization of human cytomegalovirus DNA polymerase accessory protein UL44 in origin-dependent DNA replication in cells[J]. J Virol, 2008, 82(24): 12574-12579.
[DOI]
|
| [49] |
Lin K, Dai CY, Ricciardi RP. Cloning and functional analysis of Kaposi's sarcoma-associated herpesvirus DNA polymerase and its processivity factor[J]. J Virol, 1998, 72(7): 6228-6232.
[DOI]
|
| [50] |
Baltz JL, Filman DJ, Ciustea M, Silverman JE, Lautenschlager CL, Coen DM, Ricciardi RP, Hogle JM. The crystal structure of PF-8, the DNA polymerase accessory subunit from Kaposi's sarcoma-associated herpesvirus[J]. J Virol, 2009, 83(23): 12215-12228.
[DOI]
|
| [51] |
Chan SR, Chandran B. Characterization of human herpesvirus 8 ORF59 protein (PF-8) and mapping of the processivity and viral DNA polymerase-interacting domains[J]. J Virol, 2000, 74(23): 10920-10929.
[DOI]
|
| [52] |
Zhou X, Liao Q, Ricciardi RP, Peng C, Chen X. Kaposi's sarcoma-associated herpesvirus processivity factor-8 dimerizes in cytoplasm before being translocated to nucleus[J]. Biochem Biophys Res Commun, 2010, 397(3): 520-525.
[DOI]
|
 2021, Vol. 16
2021, Vol. 16


