乙型肝炎病毒(hepatitis B virus,HBV)慢性感染所致慢性乙型肝炎及其相关肝硬化、肝癌等肝脏疾病严重威胁人类的健康[1]。尽管预防性乙型肝炎疫苗的使用已大大降低HBV在人群中的感染率,但目前仍缺乏能有效治愈慢性乙型肝炎的药物和治疗手段。HBV基因组以松弛环状DNA(relaxed circular DNA,rcDNA)形式包裹在病毒核衣壳内,在病毒感染宿主细胞后,其会被修复成环进而以共价闭合环状DNA(covalently closed circular DNA,cccDNA)的形式存在于细胞核中。HBV cccDNA兼具病毒转录复制模板和基因储存库的功能,可转录出各种HBV RNA,进而翻译出包括乙型肝炎表面抗原(hepatitis B surface antigen,HBsAg)在内的各类病毒蛋白[2]。HBV cccDNA在肝内的持续存在、难以清除是乙型肝炎慢性化和难治愈的关键,也是HBV研究领域的重点[3]。
在HBV cccDNA研究中,HBV cccDNA的准确检测至关重要,而从细胞中抽提获取可供DNA印迹(Southern blot)和定量聚合酶链式反应(quantitative polymerase chain reaction, qPCR)检测的HBV cccDNA是前提。传统Hirt法是由Hirt等[4]报道的用来抽提病毒感染细胞后存在的非整合病毒DNA的方法。该法利用染色体DNA很长且与大量蛋白质相结合的特性,以高盐溶液使染色体DNA沉淀,而游离于染色体外的微小DNA大多不被沉淀而留在上清中,因此被分离[4]。此法结合酚/氯抽提,很早便应用于HBV cccDNA的抽提[5],但存在操作复杂和耗时长等弊端。为简化操作和缩短实验时间,早前有报道通过改良Hirt法来快速抽提染色体外DNA,该法裂解细胞和沉淀染色体DNA的原理类似于传统Hirt法,不同之处在于通过改变盐溶液的溶质成分(详见材料与方法)使蛋白质的沉淀更加完全也更迅速[6],所获上清不经传统Hirt法中的酚/氯仿抽提步骤,而利用硅胶膜离心柱使染色体外DNA吸附于硅胶膜,随后通过洗脱完成分离[6-8],整个过程仅需2~3 h。与传统Hirt-酚/氯仿抽提法相比,改良Hirt-过柱法操作简便、耗时显著缩短,但尚未被广泛用于HBV cccDNA抽提,且其抽提效果与传统Hirt-酚/氯仿法的差异尚不清楚。
基于此,本研究利用多种HBV cccDNA细胞研究模型,以Southern blot和qPCR为评价手段,比较了传统Hirt-酚/氯仿法与改良Hirt-过柱法这2种方法对细胞来源HBV cccDNA的抽提效率。结果表明,在对以上来源HBV cccDNA的抽提中,两种方法具有相当的抽提效率和抽提特异性,而改良Hirt-过柱法耗时更短。
1 材料与方法 1.1 材料 1.1.1 细胞系Huh 7细胞和HepG2细胞由本实验室保存。HepDES19细胞系由含1.1拷贝HBV基因组的质粒稳定转染HepG2细胞建立而成,不表达HBsAg,且病毒的转录和复制受四环霉素(doxycycline,Dox)调控(Tet-off系统)[9],由Jutao Guo教授惠赠。HepG2-NTCP细胞系是整合人NTCP基因、能稳定表达人NTCP的HepG2细胞[10],由Stephan Urban教授惠赠。人原代肝细胞(primary human hepatocyte,PHH)购自上海瑞德肝脏有限公司。
1.1.2 质粒prcccDNA[11-12]和pCDH-CMV-Cre(构建在慢病毒载体上的Cre重组酶表达质粒)由邓强研究员惠赠。
1.1.3 仪器和试剂主要仪器包括酸度(pH)计(上海天达仪器有限公司)、二氧化碳细胞培养箱(Thermo Fisher Scientific,美国)、荧光定量PCR仪(Eppendorf,德国)、电泳槽(北京百晶生物技术有限公司)、化学发光成像系统(BIO-RAD,美国)、杂交炉(UVP,美国)。主要试剂包括胎牛血清(fotal bovine serum, FBS)(Biological Industries,以色列)、DMEM(CORNING,美国)、Williams’ Medium E(Gibco,美国)、Trypsin(CORNING,美国)、质粒中量抽提试剂盒Nucleo Bond Xtra Midi Plus(Macherey-Nagel,美国)、转染试剂Turbofect(Thermo Fisher Scientific,美国)、吸附柱(2.0 spin column,QIAGEN,德国)、洗涤缓冲液(Buffer PE,QIAGEN,德国)、PCR DIG Probe Synthesis Kit(Roche,瑞士)、DIG Easy Hyb Granules(Roche,德国)、尼龙膜(带正电荷)(Roche,瑞士)、CDP-Star(Roche,瑞士)、TB Green Premix Ex TaqTM(Takara,日本)、PEG 8000(Sigma,美国)、二甲基亚砜(dimethyl sulfoxide,DMSO)(Sigma,美国)、十二烷基硫酸钠(sodium dodecylsulfate,SDS)(Sigma,美国)。
1.2 方法 1.2.1 细胞培养Huh 7细胞、HepG2细胞以及HepG2-NTCP细胞均在37 ℃体积分数5% 的CO2培养箱中用DMEM培养基(含体积分数10% FBS,100 U/mL青霉素和100 μg/mg链霉素)培养;HepDES19细胞扩大培养期间在DMEM中添加Dox,使HBV preC/pgRNA不能转录复制,到达所需规模后,停止添加Dox以使HBV cccDNA积累,每隔3天换液,累积10天后收集细胞;PHH按照相关文献中5种化合物(5 chemicals,5C)培养体系进行培养[13]。
1.2.2 转染实验用转染试剂Turbofect进行转染实验,每转染1 μg质粒用2 μL的转染试剂。转染前12 h接种细胞,使进行转染实验时细胞密度处于85%~90%。以转染1 μg质粒DNA为例,在1.5 mL EP管中加入100 μL DMEM,加入1 μg质粒DNA后充分混匀,加入2 μL转染试剂Turbofect后再混匀,室温静置15 min后将其缓慢加入被转染的细胞孔板内,做好标记。12 h之后对被转染细胞进行换液。
1.2.3 HBV感染实验感染前24 h将HepG2-NTCP细胞、PHH分别接种到24孔板中,每孔约2.5×105个细胞。感染时所用的HBV病毒浓缩液来自本实验室浓缩的HepAD38细胞(受Dox调控的HBV复制细胞系[14])上清液,其滴度为2×109 IU/mL,按照感染复数(multiplicity of infection,MOI)为200进行感染。感染PHH时,参照PHH-5C的感染模型[13],每孔的感染体系为:Null medium 425 μL,0.4 g/mL PEG8000 50 μL,HBV浓缩液25 μL;感染24 h之后用磷酸盐缓冲液(phosphate buffer solution,PBS)洗涤3遍换新鲜的5C培养基进行培养,每隔3天换液,9天后收样备测HBV cccDNA。感染HepG2-NTCP细胞时每孔的感染体系为:DMEM培养基412.5 μL,0.4 g/mL PEG8000 50 μL,DMSO 12.5 μL,HBV浓缩液25 μL;感染12 h后用PBS洗涤5遍,换新鲜的DMEM(含体积分数为10% 的FBS)进行培养,每隔3天换液,9天后收样备测HBV cccDNA。
1.2.4 传统Hirt-酚/氯仿法抽提DNA以6孔板为例,从培养箱中取出的细胞以预冷PBS洗涤2遍之后,每孔加750 μL的TE buffer [10 mmol/L Tris-HCl, pH值7.5; 10 mmol/L乙二胺四乙酸(ethylenediaminetetraacetic acid,EDTA)]和50 μL的10 % SDS,轻柔混匀后室温裂解30 min。将粘性裂解液转移至1.5 mL离心管中,加200 μL 5 mol/L NaCl,上下轻柔颠倒混匀10次,4 ℃放置至少16 h后离心(4 ℃,14 500 g,30 min)。上清液转移至新2 mL离心管中,加等体积饱和酚,手动摇匀呈乳浊液,离心(4 ℃,3 500 g,10 min),接下来重复该步骤1次;上层水相转移至新2 mL离心管中,加等体积酚/氯仿,手动摇匀,离心(4 ℃,3 500 g,10 min);上层水相转移至新2 mL离心管中,加等体积异丙醇,上下颠倒混匀,-20 ℃静置过夜。离心(4 ℃,10 000 g,30 min)后弃上清液,体积分数75%乙醇洗一次,弃去乙醇,待乙醇挥发完后,ddH2O溶解沉淀。
1.2.5 改良Hirt-过柱法抽提DNA以6孔板为例,从培养箱中取出的细胞以预冷的PBS洗2遍之后,每孔加500 μL的Buffer 1(50 mmol/L Tris-HCl,pH值7.5, 10 mmol/L EDTA, 50 μg/mL RNase A)和500 μL的Buffer 2(0.012 g/mL SDS),轻摇混匀后室温孵育15 min。接着加700 μL的Buffer 3 (3 mol/L氯化铯, 1 mol/L醋酸钾, 0.67 mol/L醋酸),4 ℃孵育2 h。4 ℃下以15 000 g离心15 min。收集上清液,将其转移到吸附柱中,室温下以15 000 g离心1 min。在吸附柱中加入750 μL Washing buffer,室温下以15 000 g离心1 min。弃去收集管中的废液,室温下以15 000 g离心1 min。在吸附柱中加入60 μL ddH2O,室温下以15 000 g离心1 min。
1.2.6 Southern blot检测cccDNA抽提效果具体方法参照文献和本课题组先前报道[15-16],简述如下:配制浓度为0.01 g/mL的琼脂糖凝胶,上样,70 V电泳约2 h。取出凝胶,依次用酸变性液变性10 min、碱变性液变性1 h、中和液中和1 h。结束后,将凝胶、裁剪好的尼龙膜、滤纸浸泡入含有3 mol/L氯化钠、0.3 mol/L二水合柠檬酸三钠的转移液(20×SSC)中。转膜过夜后,将尼龙膜在2×SSC溶液中浸泡5 min,接着紫外交联2 min。将尼龙膜放入杂交管中,加入5 mL的预杂交液,42 ℃预杂交30 min。取出预先用地高辛标记好的探针,100 ℃变性5 min,迅速冰浴3 min。将探针加入新的预杂交液中,配制形成杂交液。倒出预杂交液,换成杂交液,42 ℃杂交6~8 h。高盐溶液洗膜2次,每次5 min。低盐溶液洗膜2次,每次15 min。后续洗涤、封闭和抗体孵育等步骤中杂交炉温度均设定为37 ℃。洗涤缓冲液润洗1~5 min。封闭缓冲液封闭30 min。Anti-DIG抗体稀释液孵育30 min。洗涤缓冲液洗2次,每次15 min。显色缓冲液润洗2~5 min。在尼龙膜上覆盖CDP-Star工作液,室温避光5 min,显影30 min以上。
1.2.7 qPCR检测cccDNA抽提效果利用SYBR Green法检测两种方法抽提到的rcccDNA和线粒体DNA(内参基因),使用的引物序列见表 1。反应体系如下:反应混合液(2×)10 μL,正向引物、反向引物(终浓度均为10 μmol/L)各0.3 μL,DNA样本2 μL,去离子水补足至20 μL。按如下程序进行扩增反应:95 ℃ 2 min预变性后,95 ℃ 15 s、59 ℃ 15 s、72 ℃ 20 s重复45个循环。
| Primer | Sequence(5′-3′) |
| P3 | CAAGACAGGTTTAAGGAGAC |
| P4 | GAGAGAAAGGCAAAGTGGAT |
| PcccDNA1 | CTCCCCGTCTGTGCCTTCT |
| PcccDNA2 | GCCCCAAAGCCACCCAAG |
各实验组均经独立重复实验验证,每组实验测定3个样本数据,作图采用GraphPad(Prism 6),显示测定结果的平均值及标准差(SD),并进行t检验,P≤0.05代表有统计学差异。
2 结果 2.1 HBV cccDNA转染系统中,传统Hirt-酚/氯仿法与改良Hirt-过柱法抽提效率相当选择Huh 7细胞和HepG2细胞进行转染实验,分别在这两种细胞中转染prcccDNA和pCDH-CMV-Cre,转染3天后,分别用2种抽提方法抽取DNA,之后用Southern blot和qPCR(特异性引物P3、P4)对HBV rcccDNA进行检测(见图 1A、1B)。
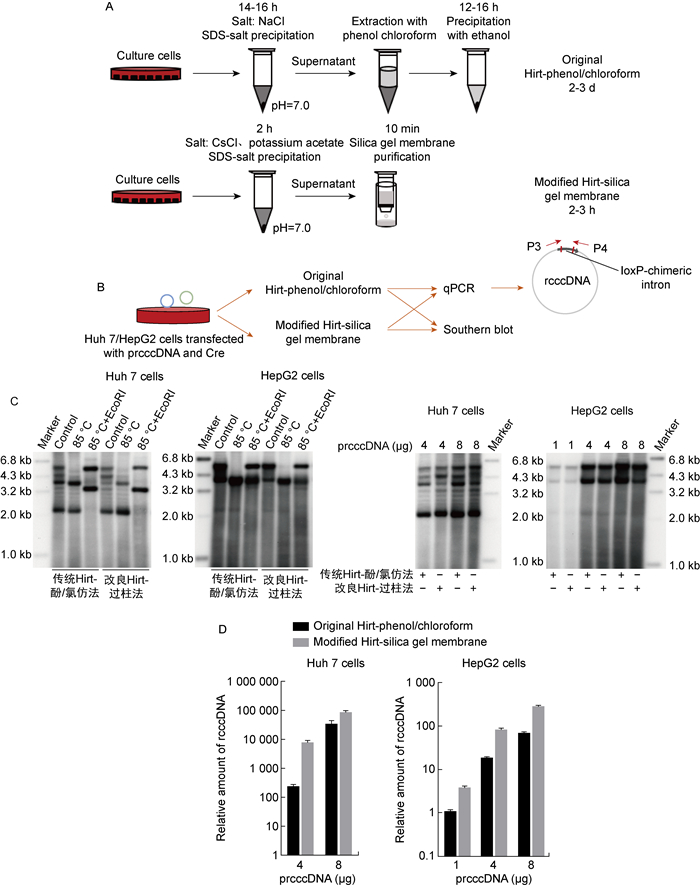
|
| A: Schematic model of two methods of HBV cccDNA extraction. B: The experimental flow of the preparation and detection of HBV rcccDNA samples in Huh 7 and HepG2 cells-based system. C: Southern blot detection of the rcccDNA extracted by the two indicated methods. D: Quantification of the extracted rcccDNA using qPCR. 图 1 两种抽提方法对HBV cccDNA转染系统来源rcccDNA样品的抽提效果比较 Fig. 1 Comparison of rcccDNA extraction efficiency and specificity between two extraction methods in HBV transfection system |
Southern blot结果显示,从转染prcccDNA和pCDH-CMV-Cre的Huh 7细胞、HepG2细胞中,分别用两种方法抽提得到的样本均可在约2.0 kb处检测到明显条带(见图 1C)。由于rcccDNA含一个EcoR I位点,样品经限制性内切酶EcoR I处理后,Southern blot显示条带均上移。同时,在prcccDNA和pCDH-CMV-Cre转染量不同的情况下,传统Hirt-酚/氯仿法与改良Hirt-过柱法抽提到的HBV rcccDNA条带大小特征无明显差异(见图 1C)。qPCR结果亦显示,两种方法均可抽提到HBV的rcccDNA(见图 1D),随着prcccDNA和pCDH-CMV-Cre转染量的增加,以引物P3、P4检测到的HBV rcccDNA含量也增加,且在同等rcccDNA转染量之下,改良Hirt-过柱法与传统Hirt-酚/氯仿法抽提所得rcccDNA样品的检测读值接近,提示两种方法的抽提效率相当(见图 1D)。
2.2 HBV复制细胞系(HepDES19)系统中,改良Hirt-过柱法抽提效率略高培养HepDES19细胞,待HBV cccDNA积累10天后,对相同细胞量的细胞分别用两种抽提方法抽提DNA后,分别用qPCR(引物PcccDNA1、PcccDNA2)和Southern blot对以上2种方式得到的DNA进行检测,比较其对HBV cccDNA的抽提效率(见图 2A)。Southern blot结果表明,传统Hirt-酚/氯仿法与改良Hirt-过柱法抽提得到的HBV rcDNA和双链线性DNA(dslDNA)条带的带型和灰度类似,但在改良Hirt-过柱法中cccDNA条带信号较强(见图 2B);进一步使用ATP依赖的DNA酶(Plasmid-SafeTM ATP-dependent DNase,PSD)对样品进行预酶切后,利用qPCR进行验证,结果表明改良Hirt-过柱法抽提得到的HBV cccDNA含量略高,但与传统Hirt-酚/氯仿法相比无统计学差异(见图 2C)。

|
| A: The experimental flow of the preparation and detection of HBV rcccDNA samples in HepDes19 cells. B: Southern blot detection of the rcccDNA extracted by the two indicated methods. C: Quantification of the extracted rcccDNA using qPCR. 图 2 两种抽提方法对HepDES19来源cccDNA样品的抽提效果比较 Fig. 2 Comparison of cccDNA extraction efficiency and specificity between two extraction methods in the HBV-replicating system, HepDES19 |
在HepG2-NTCP感染系统中,HBV感染HepG2-NTCP细胞9天(见图 3C)后,将相同数量的细胞分别用3种方法(抽取细胞核内总DNA、传统Hirt-酚/氯仿法、改良Hirt-过柱法)抽提DNA,抽提到的细胞核内总DNA取部分利用PSD消化去除非闭合环状的HBV rcDNA,之后用qPCR(引物PcccDNA1、PcccDNA2)对以上3种方法得到的DNA进行检测,比较其对HBV cccDNA的抽提效率(见图 3A)。结果表明,细胞核内总DNA抽提所得样品中HBV cccDNA拷贝数最高,经过PSD消化后拷贝数有所降低(去除了rcDNA非特异性干扰),该拷贝数与传统Hirt-酚/氯仿法与改良Hirt-过柱法抽提所得样品中的HBV cccDNA拷贝数相当,且后两种抽提方法检测值间无统计学差异(见图 3B)。

|
| A: The experimental flow of the preparation and detection of HBV rcccDNA samples in HepG2-NTCP-and PHH-based infection assay. The cells were harvested at day 9 post HBV infection. B and C: Quantification of the extracted rcccDNA using qPCR. 图 3 两种抽提方法对HBV感染细胞来源cccDNA样品的抽提效果比较 Fig. 3 Comparison of cccDNA extraction efficiency and specificity between two extraction methods in cell culture-based HBV infection systems |
在PHH-5C感染系统中,HBV感染PHH 9天后,分别用传统Hirt-酚/氯仿法与改良Hirt-过柱法抽提DNA,之后用qPCR(引物PcccDNA1、PcccDNA2)检测抽提所得样品中HBV cccDNA的含量(见图 3A)。结果表明,在PHH-5C感染系统中,2种抽提方法所测HBV cccDNA含量无统计学差异(见图 3C),提示抽提效率相当。
2.4 改良法抽提cccDNA所用硅胶膜离心柱选择范围较广鉴于市售商业化硅胶膜离心柱材质和性能的不尽相同,为比较不同制造商来源硅胶膜离心柱用于rcccDNA抽提的效率差异,本研究选用7家不同公司来源的硅胶膜离心柱,分别是QIAGEN(27106)、Axygen(AP-NW-P-250)、MACHEREY-NAGEL(740588.250)、SparkJade(AD0105)、Thermo Fisher(K0503)、TransGen Biotech(ER5001)、TIANGEN(DP105-03),括号内为货号。基于Huh 7细胞中转染prcccDNA和pCDH-CMV-Cre的研究模型,将转染细胞均分为8份,分别用传统Hirt-酚/氯仿法和使用上述7种硅胶膜离心柱的改良Hirt-过柱法抽提DNA。
Southern blot结果显示,不同制造商来源硅胶膜离心柱均可抽提获得目标rcccDNA,且抽提所得含量基本相当,其中QIAGEN、Axygen、Therm Fisher及TIANGEN等的硅胶膜离心柱对rcccDNA的抽提效率相对略高。上述结果提示,在改良Hirt-硅胶膜离心柱法抽提cccDNA时,硅胶膜离心柱有着较广的选择范围(见图 4)。
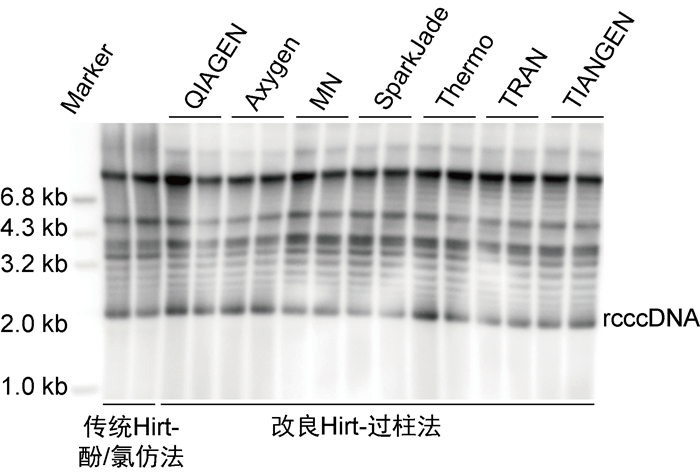
|
| Note: Southern blot detection of the rcccDNA extracted by the indicated methods. 图 4 不同来源硅胶膜离心柱用于rcccDNA抽提效率的比较 Fig. 4 Comparison of rcccDNA extraction efficiency among silica gel membranes derived from different manufacturers |
HBV cccDNA是乙型肝炎慢性化和难以治愈的“元凶”,实现对其的清除或长期功能性抑制是功能性治愈乃至根除乙型肝炎的关键。然而,目前对于HBV cccDNA的形成、调控以及代谢清除等相关机制的认识仍不十分深入,相关靶向cccDNA的手段仍在研发推进中。在HBV cccDNA研究中,准确进行HBV cccDNA检测至关重要;同时在临床,结合血清HBV病毒载量水平、抗原水平和ALT水平等常用诊断指标,HBV cccDNA检测对于乙型肝炎的病原学诊断、疾病进程分析和疗效预后的判断等具有重要意义。
Southern blot和qPCR是目前检测HBV cccDNA的两大方法,而前提是从细胞中抽提获取可供检测的cccDNA样品。传统Hirt法在TE buffer(pH值为7)环境下用SDS裂解细胞,再加入高盐溶液使蛋白质在盐析作用下发生沉淀。染色体DNA分子量大且与大量蛋白质相结合,所以会与蛋白质一同被沉淀下来。染色体外DNA因分子量小且大多仍存在于上清液中,故可对上清液进行酚/氯仿抽提,再以乙醇(或异丙醇)沉淀过夜,可获得“Hirt DNA”,整个过程需要2~3 d[4, 17-19],耗时较长。为缩短操作时间,有报道通过改变盐溶液的溶质成分使蛋白质的沉淀更加完全也更迅速,即改良Hirt法。此法去除沉淀物后得到的溶液可经硅胶膜离心柱分离抽提,相较酚/氯仿抽提进一步缩短了实验时间,但尚未被应用于HBV cccDNA抽提[6]。
本研究基于常用HBV cccDNA细胞研究模型(瞬时转染、HBV复制细胞系及感染系统),比较了改良Hirt-过柱法与传统Hirt-酚/氯仿法对cccDNA的抽提效率和所需实验时间。结果表明,两种抽提HBV cccDNA方法的得率相当,但改良Hirt-过柱法(2~3 h)与传统Hirt-酚/氯仿法(2~3 d)相比可显著节约实验时间,提高效率。需注意的是,Southern blot结果显示(见图 2B),传统Hirt-酚/氯仿法和改良Hirt-过柱法抽提得到的Hirt DNA均包括rcDNA,都无法达到对cccDNA的特异性抽提。此种情况下,Southern blot检测可通过区分条带大小和通过EcoR I酶切实验来分辨cccDNA;而在进行qPCR时,虽然采用了cccDNA特异性引物,但有研究表明仍无法避免扩增出rcDNA[20],因此建议使用PSD、T5和Exonuclease Ⅰ、Ⅲ、Ⅴ等进行预酶切[21-22],以尽量消减rcDNA对定量结果的影响。
鉴于HBV cccDNA是一种类似质粒的微小环状DNA,有报道采用质粒抽提中的“碱裂解法”对HBV cccDNA进行抽提[6, 23-24]。此方法中,rcDNA自身具有缺口,非完整闭合环状DNA,在碱变性后无法复性,而cccDNA易于复性,故该法在抽提cccDNA的过程中具有更高的特异性。但是,由于部分cccDNA在碱裂解法中也无法复性,该法更易造成样品中cccDNA的损失。cccDNA在自然感染情况下细胞内拷贝数很低,因此该法无法很好地适用于微量样品。须指出的是,本文所有实验结果都以细胞实验为主,未采用临床乙型肝炎患者的肝脏穿刺样本对两种方法进行比较。虽然,本文所得出的“两种方法抽提效能类似”的结论在理论上亦适用于临床标本,但须进一步实验评估验证。总之,本研究提示在进行细胞HBV cccDNA抽提时可选择改良Hirt-过柱法以提高实验效率,其亦适用于大规模cccDNA的抽提实验。
| [1] |
Yuen MF, Chen DS, Dusheiko GM, Janssen H, Lau D, Locarnini SA, Peters MG, Lai CL. Hepatitis B virus infection[J]. Nat Rev Dis Primers, 2018, 4: 18035.
[DOI]
|
| [2] |
Guo JT, Guo H. Metabolism and function of hepatitis B virus cccDNA: implications for the development of cccDNA-targeting antiviral therapeutics[J]. Antiviral Res, 2015, 122: 91-100.
[DOI]
|
| [3] |
宰文静, 陈捷亮, 袁正宏. HBV cccDNA转录代谢调控机制及沉默清除策略[J]. 临床肝胆病杂志, 2020, 36(5): 983-988. [DOI]
|
| [4] |
Hirt B. Selective extraction of polyoma DNA from infected mouse cell cultures[J]. J Mol Biol, 1967, 26(2): 365-369.
[DOI]
|
| [5] |
Tuttleman JS, Pourcel C, Summers J. Formation of the pool of covalently closed circular viral DNA in hepadnavirus-infected cells[J]. Cell, 1986, 47(3): 451-460.
[DOI]
|
| [6] |
Arad U. Modified Hirt procedure for rapid purification of extrachromosomal DNA from mammalian cells[J]. Biotechniques, 1998, 24(5): 760-762.
[DOI]
|
| [7] |
Hamelin C, Yelle J, D'Amours B, Chung YS. SV40 DNA extracted from persistently infected Vero cells using miniprep columns for plasmids[J]. Cell Mol Biol Res, 1993, 39(3): 289-293.
[PubMed]
|
| [8] |
Li X, Zhao J, Yuan Q, Xia N. Detection of HBV covalently closed circular DNA[J]. Viruses, 2017, 9(6): 139.
[DOI]
|
| [9] |
Guo H, Jiang D, Zhou T, Cuconati A, Block TM, Guo J T. Characterization of the intracellular deproteinized relaxed circular DNA of hepatitis B virus: an intermediate of covalently closed circular DNA formation[J]. J Virol, 2007, 81(22): 12472-12484.
[DOI]
|
| [10] |
Yan H, Zhong G, Xu G, He W, Jing Z, Gao Z, Huang Y, Qi Y, Peng B, Wang H, Fu L, Song M, Chen P, Gao W, Ren B, Sun Y, Cai T, Feng X, Sui J, Li W. Sodium taurocholate cotransporting polypeptide is a functional receptor for human hepatitis B and D virus[J]. Elife, 2012(1): e00049.
[DOI]
|
| [11] |
Qi Z, Li G, Hu H, Yang C, Zhang X, Leng Q, Xie Y, Yu D, Zhang X, Gao Y, Lan K, Deng Q. Recombinant covalently closed circular hepatitis B virus DNA induces prolonged viral persistence in immunocompetent mice[J]. J Virol, 2014, 88(14): 8045-8056.
[DOI]
|
| [12] |
朱园飞, 李改云, 常豪, 鱼康康, 高月求, 邓强. 应用微环DNA技术体外诱导和制备重组的乙型肝炎病毒共价闭合环状DNA[J]. 微生物与感染, 2017, 12(4): 229-234. [DOI]
|
| [13] |
Xiang C, Du Y, Meng G, Soon YL, Sun S, Song N, Zhang X, Xiao Y, Wang J, Yi Z, Liu Y, Xie B, Wu M, Shu J, Sun D, Jia J, Liang Z, Sun D, Huang Y, Shi Y, Xu J, Lu F, Li C, Xiang K, Yuan Z, Lu S, Deng H. Long-term functional maintenance of primary human hepatocytes in vitro[J]. Science, 2019, 364(6438): 399-402.
[DOI]
|
| [14] |
Ladner SK, Otto MJ, Barker CS, Zaifert K, Wang GH, Guo JT, Seeger C, King RW. Inducible expression of human hepatitis B virus (HBV) in stably transfected hepatoblastoma cells: a novel system for screening potential inhibitors of HBV replication[J]. Antimicrob Agents Chemother, 1997, 41(8): 1715-1720.
[DOI]
|
| [15] |
Cai D, Nie H, Yan R, Guo JT, Block TM, Guo H. A southern blot assay for detection of hepatitis B virus covalently closed circular DNA from cell cultures[J]. Methods Mol Biol, 2013, 1030: 151-161.
[PubMed]
|
| [16] |
Zhang W, Chen J, Wu M, Zhang X, Zhang M, Yue L, Li Y, Liu J, Li B, Shen F, Wang Y, Bai L, Protzer U, Levrero M, Yuan Z. PRMT5 restricts hepatitis B virus replication through epigenetic repression of covalently closed circular DNA transcription and interference with pregenomic RNA encapsidation[J]. Hepatology, 2017, 66(2): 398-415.
[DOI]
|
| [17] |
Mu D, Yan L, Tang H, Liao Y. A sensitive and accurate quantification method for the detection of hepatitis B virus covalently closed circular DNA by the application of a droplet digital polymerase chain reaction amplification system[J]. Biotechnol Lett, 2015, 37(10): 2063-2073.
[DOI]
|
| [18] |
Takkenberg RB, Zaaijer HL, Menting S, Weegink CJ, Terpstra V, Cornelissen M, Dijkgraaf MG, Jansen PL, Reesink HW, Beld MG. Detection of hepatitis B virus covalently closed circular DNA in paraffin-embedded and cryo-preserved liver biopsies of chronic hepatitis B patients[J]. Eur J Gastroenterol Hepatol, 2010, 22(8): 952-960.
[DOI]
|
| [19] |
Gao YT, Han T, Li Y, Yang B, Wang Y J, Wang FM, Jing X, Du Z. Enhanced specificity of real-time PCR for measurement of hepatitis B virus cccDNA using restriction endonuclease and plasmid-safe ATP-dependent DNase and selective primers[J]. J Virol Methods, 2010, 169(1): 181-187.
[DOI]
|
| [20] |
Lucifora J, Protzer U. Attacking hepatitis B virus cccDNA—The holy grail to hepatitis B cure[J]. J Hepatol, 2016, 64(1 Suppl): S41-S48.
[PubMed]
|
| [21] |
Qu B, Ni Y, Lempp FA, Vondran F, Urban S. T5 exonuclease hydrolysis of hepatitis B virus replicative intermediates allows reliable quantification and fast drug efficacy testing of covalently closed circular DNA by PCR[J]. J Virol, 2018, 92(23): e01117-18.
[DOI]
|
| [22] |
Gao Z, Yan L, Li W. A quantitative method for hepatitis B virus covalently closed circular DNA enables distinguishing direct acting antivirals from cytotoxic agents[J]. Antiviral Res, 2019, 168: 197-202.
[DOI]
|
| [23] |
Birnboim HC, Doly J. A rapid alkaline extraction procedure for screening recombinant plasmid DNA[J]. Nucleic Acids Res, 1979, 7(6): 1513-1523.
[DOI]
|
| [24] |
Lutze LH, Winegar RA. A quick and efficient method for the recovery of plasmid or viral DNA from mammalian cells[J]. Nucleic Acids Res, 1990, 18(20): 6150.
[DOI]
|
 2021, Vol. 16
2021, Vol. 16


