乙型肝炎病毒(hepatitis B virus, HBV)是一类有包膜的小型DNA病毒,属于嗜肝DNA病毒科[1]。成熟的HBV含有约3.2 kb的松弛环状DNA(relaxed circular DNA, rcDNA),负链DNA 5′端共价连接病毒多聚酶(polymerase,Pol)。在病毒感染的肝细胞核内,rcDNA修复为共价闭合环状DNA(covalently closed circular DNA, cccDNA),是HBV转录复制的原始模板[2-4]。以cccDNA为模板转录的前基因组RNA(pregenomic RNA, pgRNA)转运至细胞浆被核衣壳包裹,通过Pol反转录生成负链DNA,并合成不完全的正链DNA;部分双链rcDNA通过成熟的核衣壳颗粒再次转运至细胞核,完成HBV的细胞内复制周期[5]。另一方面, HBV核衣壳颗粒通过内体分选转运复合体机制,经由内体多泡体结构完成包膜化和出芽。HBV已知的天然宿主仅限于人和黑猩猩,钠牛磺胆酸共转运多肽(sodium taurcholate cotransport peptide, NTCP)被证明是HBV高亲和力受体[6-8],这极大地促进了研究者对HBV从头感染的研究。然而,HBV/NTCP感染体系依赖较高的感染复数,病毒感染和扩增效率较低,病毒粒子表面抗原与受体结合后的细胞进入机制仍需要深入研究。
重组病毒复制子通常在病毒基因组或亚基因组中插入外源报告基因,使其能够支持重组病毒进行功能性复制,具有可检测性和量化特征,如在丙型肝炎病毒、新型冠状病毒[9-13]RNA复制子中引入荧光素酶或荧光蛋白表达基因等,是病毒学体外研究的重要工具。值得注意的是,HBV 3.2 kb基因组高度压缩,4个主要阅读框相互重叠,每一个碱基都具有编码功能和潜在的顺式调控功能,因此HBV基因组难以容纳外源基因,构建重组复制子具有较大的挑战性。Hong等[14]应用包膜蛋白缺陷的HBV高复制突变株,在病毒Pol spacer编码区插入红色荧光蛋白(red fluorescent protein, RFP)基因,能够支持HBV重组病毒(recombinant HBV, rHBV)的复制;Bai等[15]基于上述突变株系统重新设计rHBV复制子,将外源报告基因插入HBV核心蛋白(HBV core, HBc)编码区,通过反式互补HBc拯救重组病毒复制。尽管如此,受限于大片段插入导致的低复制效率,上述系统并未能获得广泛应用。
内含肽(intein)可分割为氮端(IntN)和碳端(IntC)结构域[16-18],应用IntN、IntC分别融合绿色荧光蛋白(green fluorescent protein, GFP)氮端(GFPN)和碳端(GFPC)分裂蛋白,通过IntN和IntC高亲和力相互作用、催化自身移除,介导GFP分裂蛋白基于肽键的共价连接。
鉴于此,本研究基于HBV ayw3通用基因组序列,删除HBc第23~136氨基酸(amino acid, aa)编码区,构建复制缺陷的ΔHBc113载体。由于插入序列容量限制,选取intein融合的GFP作为报告基因产物。依据11个β-片层组成的二级结构,将增强绿色荧光蛋白(enhanced GFP, EGFP)分割成EGFPN1-8(aa 1~173)和EGFPC9-11(aa 174~232);或将超折叠绿色荧光蛋白(super folder GFP, sfGFP)拆分为sfGFPN1-10(aa 1~214)和sfGFPC11(aa 215~230)。应用IntC融合EGFPC或sfGFPC,将其序列插入ΔHBc113载体,在HBc反式互补条件下实现重组病毒rHBV的复制,IntC-GFPC表达产物则能够与共表达的IntN-GFPN高效拼接,产生功能性荧光蛋白。期望这一可视化的模型系统能为HBV高通量药物筛选提供实验工具,也有助于对HBV受体和受体后病毒学特征的进行深入研究。
1 材料与方法 1.1 材料 1.1.1 细胞系293T细胞系、HepG2细胞系由本课题组保存。
1.1.2 质粒pCDNA3.1、巨细胞病毒(cytomegalovirus, CMV)-HBV1.1(由CMV启动子启动的1.1拷贝的HBV基因组)、pCDH-CMV-MCS-EF1-puro(由CMV启动子启动、带有多克隆位点和筛选标记Puro的慢病毒载体)、pCDH-EGFP、pCDH-sfGFP均由本课题组保存。pCDNA3.1-IntC-EGFPC9-11(可表达intein碳端和EGFP碳端的融合蛋白)、pCDNA3.1-EGFPN1-8-IntN(可表达EGFP氮端和intein氮端的融合蛋白)、pCDNA3.1-IntC-sfGFPC11(可表达intein碳端和sfGFP碳端的融合蛋白)、pCDNA3.1-sfGFPN1-10-IntN(可表达sfGFP氮端和intein氮端的融合蛋白)均由上海桑尼生物科技有限公司合成。
1.1.3 仪器和试剂主要仪器有聚合酶链反应(polymerase chain reaction, PCR)仪(Eppendorf公司)、二氧化碳细胞培养箱(Thermo公司)、荧光倒置显微镜(Nikon公司)、pH计(上海天达仪器有限公司)、化学发光成像系统ChemiDocXRS+(Bio-Rad公司)。主要试剂有PrimeSTAR Max DNA Polymerase(TaKaRa公司)、Seamless Cloning试剂盒(碧云天生物技术有限公司)、无内毒素质粒小提中量试剂盒[天根生化科技(北京)有限公司]、凝胶DNA小量回收试剂盒(美基生物有限公司)、EZ Trans细胞转染试剂(上海李记生物科技有限公司)、DMEM培养基(Gibco公司)、胎牛血清(Gibco公司)、Trypsin-EDTA(Gibco公司)。所用的引物由北京擎科新业生物技术有限公司合成,引物序列见表 1。
| Name | Sequence (5′-3′) |
| sfGFPN1-10-IntN-F | TAGAAGATTCTAGAGCTAGCGAATTCGCCACCATGGTGA |
| sfGFPN1-10-IntN-R | CGAACTGAGGGTGGCTCCAGGATCCCTAGGCGGTGGCC |
| IntC-sfGFPC11-F | GGCAGTACATCAAGTGTATC |
| IntC-sfGFPC11-R | CGTCTAACAACAGTAGTCTCCGGAAGTGTTGATAGGATA |
| EGFPN1-8-IntN-F | TAGAAGATTCTAGAGCTAGCGAATTCGCCACCATGGTGA |
| EGFPN1-8-IntN-R | CGAACTGAGGGTGGCTCCAGGATCCCTAGGCGGTGGCC |
| IntC-EGFPC9-11-F | CGTTTTTGCCTTCTGACTTCGTCAAAATCGTCAGACGTAG |
| IntC-EGFPC9-11-R | GTCTAACAACAGTAGTCTCCGGAAGTGTTGATAGGATAG |
| ΔHBc113-F | GGCAGTACATCAAGTGTATC |
| ΔHBc113-R | GTCGTCTAACAACAGTAGTCTCCGGAAGTGTTGATAGGATAGGGGCATG- AAGTCAGAAGGCAAAAACG |
以pCMV-HBV1.1(HBV ayw3,GenBank N°V01460)为模板,ΔHBc113-F和ΔHBc113-R为引物进行PCR,利用Seamless Cloning试剂盒,将PCR产物无缝连接在pCMV-HBV1.1载体NdeⅠ和BspEⅠ酶切位点之间,获得HBV1.1-ΔHBc113质粒。
通过基因合成获得念珠藻类(Nostoc punctiforme PCC73102, Npu)intein编码序列。应用常规PCR扩增获得IntC-EGFPC9-11、EGFPN1-8-IntN、IntC-sfGFPC11以及sfGFPN1-10-IntN融合蛋白编码DNA,分别克隆至真核表达质粒pCDNA3.1(+)。以相应质粒为模板,应用IntC-EGFPC9-11-F和IntC-EGFPC9-11-R,或IntC-sfGFPC11-F和IntC-sfGFPC11-R为引物分别进行PCR扩增,将PCR产物连接在HBV1.1-ΔHBc113载体NdeⅠ和BspEⅠ酶切位点之间,构建质粒分别命名为ΔHBc113/EGFPC9-11和ΔHBc113/sfGFPC11。以EGFPN1-8-IntN-F和EGFPN1-8-IntN-R,或sfGFPN1-10-IntN-F和sfGFPN1-10-IntN-R为引物,分别扩增EGFPN1-8-IntN和sfGFPN1-10-IntN融合蛋白编码序列,将PCR产物克隆至pCDH-puro载体EcoRⅠ和BamHⅠ酶切位点间,构建慢病毒质粒pCDH-EGFPN1-8-IntN和pCDH-sfGFPN1-10-IntN。
1.2.2 细胞培养和细胞转染将HepG2和293T细胞置于5% CO2培养箱37 ℃培养,培养基为DMEM(含10%胎牛血清、100 U/mL青霉素和100 μg/mL链霉素)。将处于生长对数期的HepG2或293T细胞接种到六孔板中,24 h后细胞密度达到80%~90%准备转染。将质粒按每孔3 μg总量加至125 μL DMEM无血清培养基中,另外将9 μL EZ Trans细胞转染试剂加至125 μL DMEM无血清培养基中,充分混匀DNA溶液和转染试剂溶液,静置20 min。弃掉细胞培养液,换入2 mL新鲜无血清DMEM培养基。将质粒脂质体混合液加入细胞中,轻轻混匀后放入细胞培养箱培养。6~8 h后,弃掉培养液,加入新鲜含血清DMEM培养基,放入培养箱中培养,以供后续实验。
1.2.3 荧光信号检测实验HepG2和293T细胞经EZ Trans转染试剂转染质粒24 h后,弃去培养基,每孔加入2 mL新鲜的含血清DMEM培养液,放入培养箱中继续培养。24 h后,在荧光倒置显微镜下观察细胞绿色荧光信号,拍照记录结果。
1.2.4 提取HBc颗粒内DNAHepG2细胞转染质粒3~4 d后,弃上清,用预冷的磷酸盐缓冲液(phosphate buffered saline, PBS)洗2次。六孔板每孔加入400 μL裂解液,在37 ℃培养箱放置30 min。将裂解液14 000 g(下同)离心5 min,弃沉淀物,收集上清液,加入4 μL 1 mol/L MgCl2和8 μL 10 mg/mL DNase Ⅰ,37 ℃水浴消化30 min。然后用100 μL 35% PEG 8 000沉淀病毒颗粒,4 ℃过夜放置,离心10 min,沉淀用100 μL DNase Ⅰ溶液重悬,37 ℃水浴消化30 min。用蛋白酶K消化去除病毒核衣壳,37 ℃水浴消化过夜。用等体积的苯酚抽提1次,振荡后离心10 min,取上清水相,加入2 μL 20 mg/mL糖原、十分之一体积3 mol/L醋酸钠(pH 5.2)溶液和等体积的异丙醇,混匀后置于-20 ℃沉淀过夜,离心15 min弃上清,沉淀用75%乙醇洗2次并尽弃乙醇,静置待乙醇挥发,用20 μL去离子水溶解,-80 ℃保存。
1.2.5 提取细胞上清HBc颗粒内DNAHepG2细胞经EZ Trans转染试剂转染质粒24 h后,收集培养上清液,2 000 g离心5 min,去除细胞碎片,用孔径0.45 μm的滤膜过滤。加入PEG 8 000溶液至终浓度7%,4 ℃放置过夜。取浓缩后的400 μL上清液,重复1.2.4步骤,提取HBc颗粒内DNA。
1.2.6 DNA印迹法(Southern blotting)检测rHBV载体的细胞内复制能力配制160 mL 1%琼脂糖凝胶,加入抽提的HBV胞内和上清DNA样品,80 V电泳3 h。电泳结束后,将凝胶用变性液洗2次,每次15 min,用去离子水润洗后,再用中性液中和2次,每次15 min。将凝胶再次用去离子水清洗后,放入20×SSC浸泡。将尼龙膜、滤纸完全浸湿,按照吸水纸、滤纸、尼龙膜、凝胶、滤纸由下至上的顺序进行搭桥过夜。转膜结束后,取出尼龙膜,用2×SSC浸泡5 min,再置于2张滤纸之间,于80 ℃烘烤2 h。尼龙膜放入杂交管,用10 mL预杂交液浸润,在杂交炉42 ℃预杂交30 min。倒出预杂交液,将预热好的探针放入杂交管中,42 ℃旋转过夜孵育。回收探针,尼龙膜用10 mL高盐溶液常温洗2次,每次15 min,接着用预热的68 ℃低盐溶液洗2次,每次15 min。洗液润洗尼龙膜2 min后用吸水纸吸干,加入封闭液室温孵育30 min,再用anti-DIG antibody溶液室温孵育30 min。洗液室温润洗尼龙膜3次,每次10 min。先用显色液室温平衡膜2~5 min,加入CSPD显色液避光常温孵育5 min后,用化学发光检测仪器检测,记录结果。
2 结果 2.1 基于HBV1.1-ΔHBc113载体的重组病毒复制子的构建基于HBV ayw基因型通用序列,设计构建HBc缺失的HBV复制子载体HBV1.1-ΔHBc113(图 1A)。在HBc表达质粒共转染条件下,HBV1.1-ΔHBc113载体显示出良好的HBV DNA细胞内复制信号(图 1B)。由于HBV1.1-ΔHBc113载体HBc部分序列被删除,所以与野生型相比,HBV1.1-ΔHBc113复制子形成的rcDNA和双链线性DNA(double-stranded linear DNA, dslDNA)条带更靠前。基于上述载体进一步构建携带外源报告基因的重组复制子(图 1C),由独立的ATG位点翻译起始外源基因的表达,3′端则设计T2A断裂肽引导HBV Pol蛋白的表达。然而,引入全长GFP、RFP以及荧光素酶等报告基因,都未能有效支持重组病毒复制。
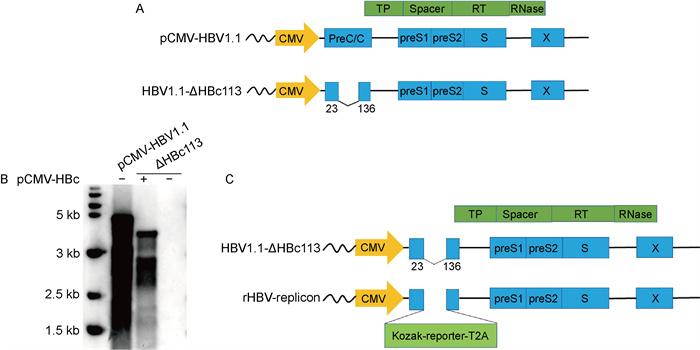
|
| A: Schematic representation of HBV1.1 replicon and ΔHBc113 mutant. B: Southern blotting of HBV intracellular replication. ΔHBc113 vector was transfected with or without pCMV-HBc in HepG2 cells. HBV1.1 replicon was served as a positive control. C: Schematic diagram of rHBV construct bearing a reporter gene. The foreign gene is initiated from an optimized Kozak signal. An in-frame T2A coding sequence is designed to facilitate HBV Pol gene expression. 图 1 重组乙型肝炎病毒复制子构建示意图 Fig. 1 Schematic diagram of rHBV replicon constructions |
Intein分割为IntC和IntN,IntC和IntN侧翼分别融合GFPN和GFPC分裂蛋白,通过IntN和IntC高亲和力相互作用、催化自身移除,介导GFP分裂蛋白基于肽键的共价连接(图 2A)。首先将EGFP分成aa 1~173(EGFPN1-8)和aa 174~232(EGFPC9-11)两部分(图 2A),基于pCDNA3.1和pCDH-puro质粒,设计构建EGFPC9-11-IntC和EGFPN1-8-IntN真核表达载体。结果显示,在体外培养的293T细胞中,单独转染EGFPC9-11-IntC或EGFPN1-8-IntN质粒未能观察到绿色荧光;当共转染2个互补质粒时,则能够观察到显著的绿色荧光信号,但弱于完整EGFP质粒转染细胞的荧光强度(图 2B)。在缺乏intein基序融合时,EGFPC9-11/EGFPN1-8表达质粒共转染,同样能够诱导GFP产生,但荧光强度显著减弱(图 2C)。
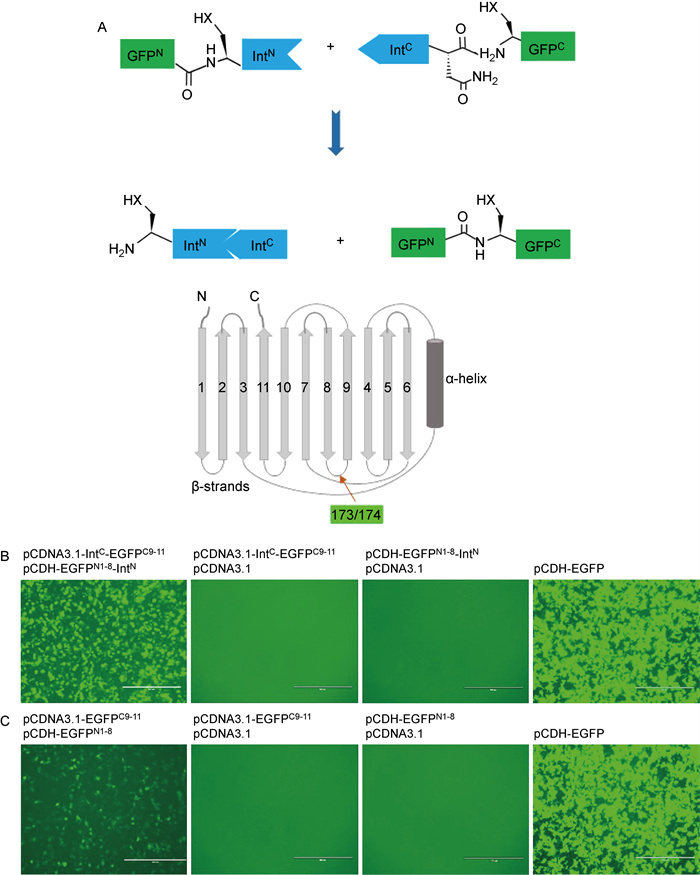
|
| A: Schematic diagram of split-GFP through intein trans-splicing pathway and consecutive β-strand numbering was shown in the EGFP structure diagram. Arrow indicates the split site (between aa 173 and 174). B: 293T cells transfected with pcDNA3.1/pCDNA3.1-IntC-EGFPC9-11, pcDNA3.1/pCDH-EGFPN1-8-IntN, or pCDNA3.1-IntC-EGFPC9-11/pCDH-EGFPN1-8-IntN, respectively. pCDH-EGFP was served as control. Scale bars, 400 μm (the same below). C: Green fluorescence signals reduced greatly in split-EGFPN1-8/C9-11 system in the absence of intein-mediated splicing. 图 2 内含肽介导的EGFPN1-8/C9-11分裂系统的构建 Fig. 2 Development of intein-mediated split-EGFPN1-8/C9-11 system |
为获得较短的GFP碳端分裂肽,本研究尝试应用sfGFP,即所谓精良折叠版本的GFP(其对融合蛋白结构具有更好的耐受性)。将sfGFP拆分为aa 1~214(sfGFPN1-10,214 aa)和aa 215~230(sfGFPC11,16 aa)(图 3A),共转染实验结果显示sfGFPC11-IntC与sfGFPN1-10-IntN具有良好的匹配,能够诱导高效拼接并产生高亮度荧光蛋白(图 3B)。同样,sfGFPC11-IntC与sfGFPN1-10-IntN质粒单独转染均不能产生荧光信号,sfGFPC11/sfGFPN1-10的拼接效率显著弱于sfGFPC11-IntC/sfGFPN1-10-IntN(数据未展示)。
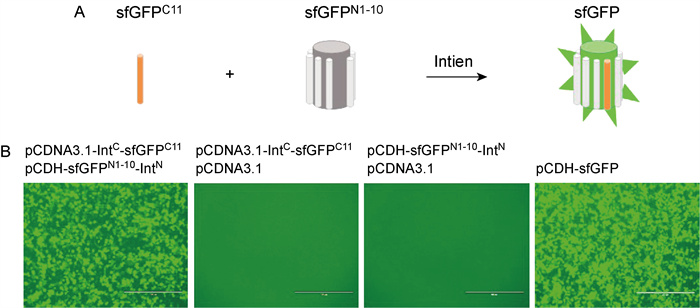
|
| A: Schematic representation of split-sfGFP system. B: 293T cells transfected with pcDNA3.1/pCDNA3.1-IntC-sfGFPC11, pcDNA3.1/pCDH-sfGFPN1-10-IntN, or pCDNA3.1-IntC-sfGFPC11/pCDH-sfGFPN1-10-IntN, respectively. pCDH-sfGFP was served as control. Scale bars, 400 μm. 图 3 内含肽介导的sfGFPN1-10/C11分裂系统的构建 Fig. 3 Development of intein-mediated split-sfGFPN1-10/C11 system |
将IntC-EGFPC9-11或IntC-sfGFPC11编码序列分别插入ΔHBc113载体,基于图 1 C构建ΔHBc113/EGFPC9-11和ΔHBc113/sfGFPC11两种rHBV复制子。如图 4A所示,在肝肿瘤细胞来源的HepG2细胞中,应用两种rHBV复制子载体与相应的EGFPN1-8-IntN或sfGFPN1-10-IntN表达质粒共转染,观察到了荧光,证明成功诱导了功能性GFP的拼接。
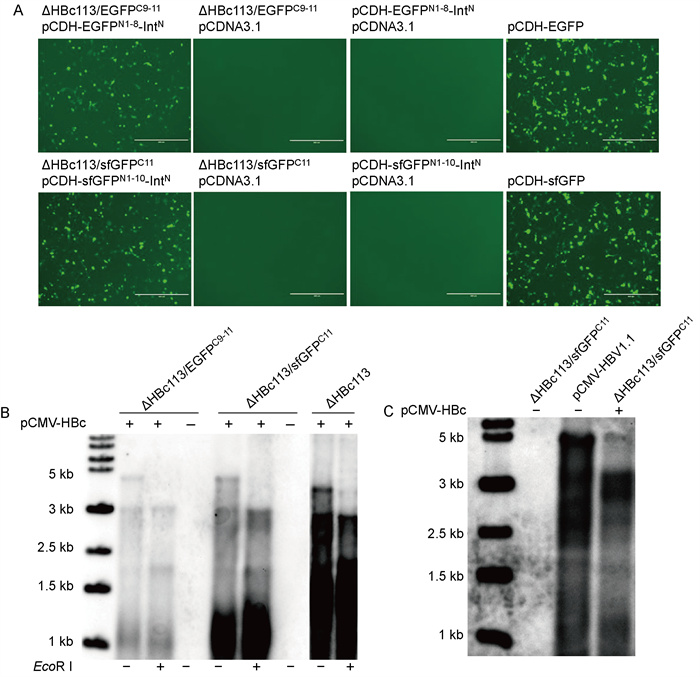
|
| A: Fluorescence analysis of HepG2 cells co-transfected with ΔHBc113/EGFPC9-11 or ΔHBc113/sfGFPC11, and their counterpart plasmids as indicated, respectively. Scale bars, 400 μm. B: Southern blotting of HepG2 cells transfected with ΔHBc113/EGFPC9-11or ΔHBc113/sfGFPC11, with or without pCMV-HBc co-transfection. rHBV DNA intermediates were validated by EcoR Ⅰ digestion. C: HepG2 cells were transfected with indicated plasmids. Virions concentrated from cell culture supernatants were subjected to Southern blotting. 图 4 ΔHBc113/sfGFPC11在HBc互补条件下诱导重组病毒的有效复制 Fig. 4 ΔHBc113/sfGFPC11accomplished functional viral replication upon HBc complementation |
在HepG2转染细胞中,应用Southern blotting验证两种rHBV载体的细胞内复制能力。如图 4B所示,单独转染ΔHBc113/EGFPC9-11或ΔHBc113/sfGFPC11质粒均不能诱导重组病毒细胞内复制。在共转染HBc表达质粒的条件下,ΔHBc113/EGFPC9-11仅显示非常弱的病毒DNA复制中间体信号,外源基因较短的ΔHBc113/sfGFPC11病毒复制信号则显著增加,但仍弱于ΔHBc113空载体。进一步用酶切方式验证重组病毒复制中间体,如图 4B所示,由于rcDNA部分双链序列中含有EcoRⅠ限制酶切位点,酶切处理后形成3.2 kb (电泳位置)的线性DNA,提示ΔHBc113/sfGFPC11载体具有可靠的细胞内复制能力。
将ΔHBc113/sfGFPC11和pCMV-HBc共转染HepG2细胞,通过PEG 8000沉淀、浓缩培养细胞上清。将pCMV-HBV1.1转染细胞上清作为阳性对照,单独转染ΔHBc113/sfGFPC11载体作为阴性对照,Southern blotting证实ΔHBc113/sfGFPC11重组复制子能够在HBc回补条件下产生子代病毒粒子(图 4C)。
3 讨论本研究实际包含两个层次的蛋白分裂系统,即GFP分裂系统和intein分裂系统。基于GFP突变衍生的EGFP和sfGFP由11个围绕中心α-螺旋的反平行β-折叠组成,可应用于分裂蛋白的研究[19, 22]。GFPN和GFPC依赖内在相互作用,非共价结合形成功能性GFP复合物,使得GFP分割位置有较大限制,严格依赖两端分裂肽的构象可塑性和亲和力。我们依据GFP蛋白二级结构中的11个β-片层结构,尝试了多种分割方案,GFPC9-11是其中最短的碳端分裂肽(数据未展示)。Zhao等[9]最近应用intein系统,获得IntN和IntC之间高亲和力连接EGFP分裂肽(N1-7/C8-11, aa 1~157/aa 158~238),显示了高效的拼接效率,且拼接后intein可自身移除。参考上述研究,本研究将intein分裂系统引入EGFPN1-8/C9-11,证实其能够显著增加EGFP分裂蛋白的拼接效率。进一步尝试应用intein策略分割EGFPN1-10/C11,但并未获成功,推测与EGFPN1-10构象可塑性较弱有关。因此,本研究尝试应用具有改良折叠构象的sfGFP,发现共表达sfGFPN1-10/C11分裂肽能够互补形成功能性荧光蛋白,但效率较低,与早先报道一致[19, 20, 23]。非常有意思的是,引入intein分裂系统显著增加了sfGFPN1-10/C11分裂肽的功能性拼接(图 3B),推测sfGFPN1-10/C11分裂肽具有较好的构象可塑性,能够稳定表达,融合intein分裂肽则能显著增加它们之间的亲和力。
HBV 3.2 kb基因组高度压缩,每一个碱基都具有病毒蛋白编码功能以及可能的顺式调控功能,后者涉及HBV cccDNA转录调控、负链DNA合成中的蛋白引物转位以及rcDNA形成过程中正链DNA的延伸和转位等。另一方面,HBc富含精氨酸的碳端结构域精确调控病毒RNA基因组的包装,过长的病毒基因组则易造成核衣壳不稳定[24-25]。rHBV复制子通常依赖反向遗传学策略,删除已知的、非顺式调控区的部分序列,并插入大小合适的外源报告基因,删除的基因可通过反式互补而拯救重组病毒复制。本研究在HBV RNA加尾信号下游删除约339 bp的HBc蛋白编码序列,构建了HBV1.1-ΔHBc113载体。ΔHBc113载体实际上代表了一种HBc缺陷的病毒基因组,由于大片段缺失(339 bp),病毒Pol编码基因阅读框向5′端大幅前移,更加接近转录起始点。该载体在HBc互补条件下,能够在转染细胞中形成高效的病毒DNA复制(图 1B)。在ΔHBc113载体中插入可用的报告基因则复杂得多,本研究首先在病毒Pol基因上游融合T2A编码序列,使得Pol与上游外源基因融合,通过断裂肽获得独立表达。尽管如此,大片段插入完整的荧光素酶或GFP序列使得ΔHBc113融合载体完全失去复制能力。有意思的是,当插入小片段(约 < 360 bp)GFP部分编码序列,Southern blotting显示明显的单链DNA(single-stranded DNA, ssRNA)形成(数据未展示)。这些结果提示,外源基因容量是rHBV细胞内复制的重要限制因素。
本研究利用intein/GFP分裂系统寻求功能性的GFP分裂短肽,以构建可视化的rHBV复制子。结果表明,ΔHBc113/sfGFPC11携带的外源序列仅编码IntC(39 aa)融合的sfGFPC11(16 aa)以及下游的T2A(21 aa),在HBc互补条件下能支持细胞内重组病毒的功能性复制(ssRNA + rcDNA),并产生子代病毒粒子(图 4)。应该指出的是,ΔHBc113/sfGFPC11荧光强度弱于野生病毒复制子,故仍有较大的改进空间。综上所述,本研究设计构建了一种GFPC分裂蛋白融合的rHBV复制子,能够在共表达HBc以及GFPN的细胞中建立病毒复制,并产生功能性荧光蛋白。未来的工作将基于ΔHBc113/sfGFPC11子代病毒粒子从头感染sfGFPN1-10表达细胞,建立HBV可视化细胞培养模型,期望可用于进一步深入研究HBV复制机制以及抗病毒药物筛选。
| [1] |
Seeger C, Mason WS. Hepatitis B virus biology[J]. Microbiol Mol Biol Rev, 2000, 64(1): 51-68.
[DOI]
|
| [2] |
Mason WS, Seal G, Summers J. Virus of Pekin ducks with structural and biological relatedness to human hepatitis B virus[J]. J virol, 1980, 36(3): 829-836.
[DOI]
|
| [3] |
Robinson WS, Clayton DA, Greenman RL. DNA of a human hepatitis B virus candidate[J]. J Virol, 1974, 14(2): 384-391.
[DOI]
|
| [4] |
Summers J, Mason WS. Replication of the genome of a hepatitis B-like virus by reverse transcription of an RNA intermediate[J]. Cell, 1982, 29(2): 403-415.
[DOI]
|
| [5] |
Beck J, Nassal M. Hepatitis B virus replication[J]. World J Gastroenterol, 2007, 13(1): 48-64.
[DOI]
|
| [6] |
Yan H, Zhong G, Xu G, He W, Jing Z, Gao Z, Huang Y, Qi Y, Peng B, Wang H, Fu L, Song M, Chen P, Gao W, Ren B, Sun Y, Cai T, Feng X, Sui J, Li W. Sodium taurocholate cotransporting polypeptide is a functional receptor for human hepatitis B and D virus[J]. Elife, 2012, 1: e00049..
[DOI]
|
| [7] |
Ni Y, Lempp FA, Mehrle S, Nkongolo S, Kaufman C, Fälth M, Stindt J, Königer C, Nassal M, Kubitz R, Sültmann H, Urban S. Hepatitis B and D viruses exploit sodium taurocholate co-transporting polypeptide for species-specific entry into hepatocytes[J]. Gastroenterology, 2014, 146(4): 1070-1083.
[DOI]
|
| [8] |
Yan H, Peng B, He W, Zhong G, Qi Y, Ren B, Gao Z, Jing Z, Song M, Xu G, Sui J, Li W. Molecular determinants of hepatitis B and D virus entry restriction in mouse sodium taurocholate cotransporting polypeptide[J]. J Virol, 2013, 87(14): 7977-7991.
[DOI]
|
| [9] |
Zhao F, Zhao T, Deng L, Lv D, Zhang X, Pan X, Xu J, Long G, Diamond M S. Visualizing the essential role of complete virion assembly machinery in efficient hepatitis C virus cell-to-cell transmission by a viral infection-activated split-intein-mediated reporter system[J]. J Virol, 2017, 91(2): e01720-16.
[DOI]
|
| [10] |
Ogando NS, Dalebout TJ, Zevenhoven-Dobbe JC, Limpens RWAL, van der Meer Y, Caly L, Druce J, de Vries JJC, Kikkert M, Bárcena M, Sidorov I, Snijder EJ. SARS-coronavirus-2 replication in Vero E6 cells: replication kinetics, rapid adaptation and cytopathology[J]. J Gen Virol, 2020, 101(9): 925-940.
[DOI]
|
| [11] |
Fernandes RS, Freire MCLC, Bueno RV, Godoy AS, Gil LHVG, Oliva G. Reporter replicons for antiviral drug discovery against positive single-stranded RNA viruses[J]. Viruses, 2020, 12(6): 598.
[DOI]
|
| [12] |
Li JQ, Deng CL, Gu D, Li X, Shi L, He J, Zhang QY, Zhang B, Ye HQ. Development of a replicon cell line-based high throughput antiviral assay for screening inhibitors of Zika virus[J]. Antiviral Res, 2018, 150: 148-154.
[DOI]
|
| [13] |
Hou YJ, Okuda K, Edwards CE, Martinez DR, Asakura T, Dinnon KH 3rd, Kato T, Lee RE, Yount BL, Mascenik TM, Chen G, Olivier KN, Ghio A, Tse LV, Leist SR, Gralinski LE, Schäfer A, Dang H, Gilmore R, Nakano S, Sun L, Fulcher ML, Livraghi-Butrico A, Nicely NI, Cameron M, Cameron C, Kelvin DJ, de Silva A, Margolis DM, Markmann A, Bartelt L, Zumwalt R, Martinez FJ, Salvatore SP, Borczuk A, Tata PR, Sontake V, Kimple A, Jaspers I, O'Neal WK, Randell SH, Boucher RC, Baric RS. SARS-CoV-2 reverse genetics reveals a variable infection gradient in the respiratory tract[J]. Cell, 2020, 182(2): 429-446.
[DOI]
|
| [14] |
Hong R, Bai W, Zhai J, Liu W, Li X, Zhang J, Cui X, Zhao X, Ye X, Deng Q, Tiollais P, Wen Y, Liu J, Xie Y. Novel recombinant hepatitis B virus vectors efficiently deliver protein and RNA encoding genes into primary hepatocytes[J]. J Virol, 2013, 87(12): 6615-6624.
[DOI]
|
| [15] |
Bai W, Cui X, Chen R, Tao S, Hong R, Zhang J, Zhang J, Wang Y, Xie Y, Liu J. Re-designed recombinant hepatitis B virus vectors enable efficient delivery of versatile cargo genes to hepatocytes with improved safety[J]. Viruses, 2016, 8(5): 129.
[DOI]
|
| [16] |
Iwai H, Züger S, Jin J, Tam PH. Highly efficient protein trans-splicing by a naturally split DnaEintein from Nostoc punctiforme[J]. FEBS lett, 2006, 580(7): 1853-1858.
[DOI]
|
| [17] |
Perler FB. InBase, the intein database[J]. Nucleic Acids Res, 2000, 28(1): 344-345.
[DOI]
|
| [18] |
Dassa B, London N, Stoddard BL, Schueler-Furman O, Pietrokovski S. Fractured genes: a novel genomic arrangement involving new split inteins and a new homing endonuclease family[J]. Nucleic Acids Res, 2009, 37(8): 2560-2573.
[DOI]
|
| [19] |
Cabantous S, Terwilliger TC, Waldo GS. Protein tagging and detection with engineered self-assembling fragments of green fluorescent protein[J]. Nat Biotechnol, 2005, 23(1): 102-107.
[DOI]
|
| [20] |
Pedelacq JD, Cabantous S. Development and applications of superfolder and split fluorescent protein detection systems in biology[J]. Int J Mol Sci, 2019, 20(14): 3479.
[DOI]
|
| [21] |
Pedelacq JD, Cabantous S, Tran T, Terwilliger TC, Waldo GS. Engineering and characterization of a superfolder green fluorescent protein[J]. Nat Biotechnol, 2006, 24(1): 79-88.
[DOI]
|
| [22] |
Ghosh I, Hamilton AD, Regan L. Antiparallel leucine zipper-directed protein reassembly: Application to the green fluorescent protein[J]. J Am Chem Soc, 2000, 122(23): 5658-5659.
[DOI]
|
| [23] |
Cabantous S, Waldo GS. In vivo and in vitro protein solubility assays using split GFP[J]. Nat Methods, 2006, 3(10): 845-854.
[DOI]
|
| [24] |
Ludgate L, Liu K, Luckenbaugh L, Streck N, Eng S, Voitenleitner C, Delaney WE 4th, Hu J. Cell-free hepatitis B virus capsid assembly dependent on the core protein C-terminal domain and regulated by phosphorylation[J]. J Virol, 2016, 90(12): 5830-5844.
[DOI]
|
| [25] |
Guo F, Zhao Q, Sheraz M, Cheng J, Qi Y, Su Q, Cuconati A, Wei L, Du Y, Li W, Chang J, Guo JT. HBV core protein allosteric modulators differentially alter cccDNA biosynthesis from de novo infection and intracellular amplification pathways[J]. PLoS Pathog, 2017, 13(9): e1006658.
[DOI]
|
 2021, Vol. 16
2021, Vol. 16


