2. 上海市宝山区友谊社区街道卫生服务中心检验科,上海 201999;
3. 国家老年疾病临床医学研究中心(复旦大学附属华山医院),上海 200040
2. Community Health Service Center of Youyi Community, Baoshan District, Shanghai 201999, China;
3. National Clinical Research Center for Aging and Medicine (Huashan Hospital Affiliated to Fudan University), Shanghai 200040, China
肠道是与外界接触的最大器官之一,寄居其中的肠道菌群易受到外界环境的影响[1]。研究证明,内外环境变化引起的肠道菌群失调与多种疾病的发生发展有关[2-4]。肠道微生态紊乱最直观的改变是肠道菌群结构的改变,表现为整体细菌数量或部分菌属丰度的改变,因此对肠道菌群中关键或重要组成菌群进行精确定量,对于了解疾病机理具有重要的意义。
已有文献证实,肠道菌群中的优势菌群与人体健康有着密切的联系[5]。肠球菌属作为哺乳动物正常肠道菌群的重要组成部分,具有较强的生存竞争能力以及对多种抗生素的内在抗性,因此也被认为是引起院内和社区感染的主要原因之一[6-7]。近期发表在Nature杂志的研究指出,粪肠球菌的异常扩张可导致非酒精性脂肪肝[8]。而健康人群肠道内的另一优势菌属是双歧杆菌属,其被证明与肠道黏膜免疫激活、肠道蠕动以及酸碱度的稳定维持有关,其含量是评估肠道健康的重要指标[9]。另一共生于肠道中的优势菌科为肠杆菌科,其中许多菌种是导致人类感染的重要病原菌和机会致病菌,不同种的肠杆菌科细菌可转移耐药基因,导致高度耐药的碳青霉烯耐药肠杆菌(carbapenem-resistant Enterobacteriaceae, CRE)的产生[10]。吴仲文等[11]提出,粪便中双歧杆菌与肠杆菌的数量比值(B/E值)可以作为肠道微生物定植抗力指标,评估肠道菌群的健康程度并预测感染疾病发生的风险。因此,针对重要菌群肠球菌属、双歧杆菌属、肠杆菌科以及肠道总菌群建立快速准确的定量方法,可以实现对菌群健康及稳定程度的快速评估,并建立针对疾病早期的无创辅助诊断方法体系。
迄今针对肠道菌群进行绝对定量的技术十分有限。由于肠道中细菌数量庞大、种类繁杂,且80%以上都不具有分离培养的技术,传统的细菌计数方法并不适用于肠道菌群定量[12]。变性梯度凝胶电泳和荧光原位杂交在一定程度上实现了对部分细菌的绝对定量,但因其较高的样本处理要求和较低的灵敏度,在肠道微生物组定量分析中应用有限[13]。高通量测序技术能够在多个层面上更精确地研究菌群结构,其中16S rDNA扩增子测序的检测范围几乎可覆盖肠道中所有菌属,但只能实现定性和相对丰度的检测。宏基因组测序实现了种水平的精确定量,但受限于较高的起始模板量和较长的检测周期[14],并不适用于常规临床检测。目前,标准化定量菌群的方法主要基于SYBR Green或Pico Green染料法的实时定量PCR(qPCR)来实现,但由于染料与DNA双链分子的结合是非特异性的,因此无法在单个反应体系同时检测多个目的基因[15]。TaqMan探针多重qPCR可以在同一反应体系内完成对多种目标基因的检测,在病毒谱快速诊断中已经得到了广泛应用[16-18],并已用于临床实践,但尚未应用于肠道共生菌的定量。因此,本研究建立对肠道菌群重要菌属的TaqMan探针多重实时荧光定量PCR法,期望对测序结果验证、菌群特征谱的临床应用推广以及前瞻性菌群相关研究等发挥积极作用。
1 材料和方法 1.1 材料 1.1.1 菌株长双歧杆菌GDMCC 1.1575购于广东省科学院微生物研究所; 大肠杆菌DH5 α购于日本Takara公司; 粪肠球菌菌株分离自健康人粪便,经16S rDNA测序及同源性比对后确认。
1.1.2 培养基BBL液体培养基(双歧杆菌液体培养基)、双歧杆菌琼脂、肠球菌液体培养基购于青岛海博生物有限公司; LB(Luria-Bertani,溶菌肉汤)培养基(胰蛋白胨、酵母提取物、氯化钠)购于上海生工生物技术有限公司。
1.1.3 试剂细菌基因组提取试剂盒Axygen AxyPrep bacteria gDNA kit购于美国CORNING公司; 粪便菌群总DNA提取试剂盒DNeasy PowerSoil kit购于德国QIAGEN公司; 质粒小提试剂盒TIANPrep mini plasmid kit购于北京天根生化科技有限公司; 胶回收试剂盒Thermo Scientific GeneJET Gel Extraction kit购于美国ThermoFisher公司; pET-28a(+)质粒、限制性内切酶Ncol及XhoI、去磷酸化酶rSAP均购于美国NEB公司; T4克隆连接试剂盒、GoTaq©qPCR Probe Master Mix均购于美国Promega公司。
1.1.4 引物和探针合成引物和探针序列由深圳华大基因科技公司合成并标记。引物和探针合成采用固相亚磷酰胺三酯法,将DNA固定在固相载体上完成DNA链的合成,由待合成引物的3 ′端向5 ′端合成延伸,相邻的核苷酸通过3 ′ →5 ′磷酸二酯键连接,在探针的5 ′端和3 ′端分别标记报告荧光基团和淬灭荧光基团,最后使用高效液相色谱法(high performance liquid chromatography,HPLC)对引物和探针进行纯化。
1.2 方法 1.2.1 细菌培养长双歧杆菌GDMCC 1.1575冻干粉用无菌水溶解稀释后,均匀涂布在双歧杆菌琼脂平板上,待长出单菌落后,挑取单菌落于BBL液体培养基中。肠球菌使用肠球菌液体培养基培养,二者均在37 ℃恒温厌氧培养箱中静置培养48 h。大肠杆菌DH5 α使用LB液体培养基,200 r/min 37 ℃恒温摇床培养12 h。
1.2.2 细菌基因组DNA的提取取1~5 mL增菌培养后的长双歧杆菌、肠球菌以及大肠杆菌菌液,低速离心(3 000 g, 3 min)去除培养基沉淀后采取高速离心(12 000 g, 3 min)获得菌体。配制裂解缓冲液(20 mmol/L Tris, pH 8.0;2 mol/L EDTA; 1.2% Triton; 20 mg/mL溶菌酶)对长双歧杆菌、肠球菌37 ℃温浴,消化细胞壁制备原生质球。按照AxyPrep bacteria gDNA kit说明,使用离心法提取细菌基因组DNA,并使用Nanodrop-1000紫外分光光度计测定DNA的浓度和纯度。
1.2.3 粪便总菌群基因组DNA的提取在粪便标本中加入磷酸缓冲盐溶液(phosphate buffered saline, PBS)缓冲液,并在震荡混匀器上以最大速度(3 200 r/min)震荡10 min,对其进行均质化处理后,使用QIAGEN DNeasy PowerSoil试剂盒,按照说明提取粪便总菌群基因组DNA,并使用Nanodrop1000紫外分光光度计测定DNA的浓度和纯度。
1.2.4 引物和探针设计通过NCBI(National Center for Biotechnology Information,https://www.ncbi.nlm.nih.gov/genome/)公布的肠球菌属、双歧杆菌属细菌的16S rDNA, 使用Snapgene对其进行比对,针对菌属特异性的保守序列设计引物和探针。通过检索国内外的相关文献[19-20],参考肠杆菌科特异性检测引物,并根据Primer Blast结果设计探针。对所有引物和探针进行优化,使所有引物退火温度接近,且不形成引物二聚体和发卡结构,经Primer-Blast/Nucleotide-Blast验证,确保该引物和探针可以最大限度检测所有可检索到16S rRNA基因序列的目标菌属/科的目标序列。根据Thermo Fisher Quant Studio 5荧光定量PCR仪检测通道,标记探针荧光基团。
1.2.5 引物和探针特异性验证以探针序列及其互补链引物序列,合成探针特异性验证引物。以肠球菌为例,探针特异性引物命名为“Enterococcus-p”,序列为5 ′ -TGGTTCTCTCCGAAATAGCTTTA-GGGCTA-3 ′及5 ′ -CAGTGCTCTACCTCCATCA-TT-3 ′。以细菌基因组DNA及粪便总菌群基因组DNA为模板进行PCR扩增,扩增条件如表 1所示,并进行琼脂糖凝胶电泳,琼脂糖浓度为2%,电泳条件为110 V恒压20 min。
| Steps | Number of cycles | Temperature(℃) | Duration |
| Predegeneration | 1 | 95 | 10 min |
| Denaturation | 95 | 30 s | |
| Annealing | 30 | 52 | 30 s |
| Extension | 72 | 30 s | |
| Extension | 1 | 72 | 3 min |
首先,进行目的片段扩增,为保证后续重组质粒的顺利构建,另设计黏性末端引物。以肠球菌为例,对原引物进行改造,添加限制性内切酶NcoI和XhoI酶切位点和保护碱基,以获得黏性末端引物,序列如下:F: 5’-CCCTCGA-GGGATGCTGGTGTGGAAGAGA-3’ (XhoI); R: 5’-TACCATGGTGCTCGCGTCCACTATCC-AGT-3’ (NcoI)。PCR扩增产物于2%琼脂糖凝胶中,110 V恒压电泳20 min。在紫外凝胶成像仪下,使用干净的手术刀片将含有DNA片段的凝胶切成薄片,尽可能切近DNA,以减少凝胶体积。此过程应尽可能迅速,避免紫外线长时间照射损坏DNA。使用Thermo Scientific GeneJET Gel Extraction试剂盒,按操作说明回收目的片段,并测定其浓度和纯度。由于目的片段较小(< 500 bp),凝胶溶解后加入同等体积的异丙醇以沉淀DNA,并减少多糖对后续步骤的影响。
然后,进行重组质粒的构建。使用限制性内切酶NcoI及XhoI、去磷酸化酶rSAP对Pet-28a(+)载体进行消化,产物经琼脂糖凝胶电泳及胶回收后,使用T4克隆连接试剂盒与目的片段连接,4 ℃过夜。将连接完成的质粒转入大肠杆菌DH5 α,经卡那霉素以及蓝白斑筛选后,挑取白色单菌落接种于5 mL卡那霉素抗性的LB培养基中,于37 ℃恒温摇床震荡培养16 h。使用TIANGEN TIAN Prep mini plasmid试剂盒提取质粒,作为模板进行PCR扩增验证及测序鉴定,扩增条件如表 2所示。
| Steps | Number of cycles | Temperature(℃) | Duration |
| Predegeneration | 1 | 95 | 10 min |
| Denaturation | 95 | 30 s | |
| Annealing | 30 | 57 | 30 s |
| Extension | 72 | 30 s | |
| Extension | 1 | 72 | 3 min |
最后,制备标准品。将成功构建的阳性载体转入大肠杆菌感受态细胞,200 r/min 37 ℃恒温摇床培养12 h并抽提质粒,测定产物浓度并转换为拷贝数(copies/μL),计算公式如下。
| $ C=c \times 10^{-9} \times \mathrm{NA} / \mathrm{MW} $ |
其中:C为拷贝数,单位为copies/μL; c为质量浓度,单位为ng/μL; NA为阿伏伽德罗常数,取6.02×1023 copies/mol; MW为摩尔质量,单位为g/mol。
将质粒模板进行10倍比稀释,冻存于-20 ℃待用。
1.2.7 反应条件的优化按GoTaq©Probe qPCR Master Mix的说明进行TaqMan探针实时定量PCR,分别用不同的引物终浓度(100 nmol/L、150 nmol/L、200 nmol/L、250 nmol/L、300 nmol/L、350 nmol/L、400 nmol/L)、探针终浓度(50 nmol/L、75 nmol/L、100 nmol/L、125 nmol/L、150 nmol/L、175 nmol/L、200 nmol/L)和循环数(30、35、40)对反应体系进行优化。
1.2.8 标准曲线和回归方程的建立以浓度为1~1012copies/μL的标准品作为阳性模板,以nuclease-free H2O作为阴性对照,按照优化好的反应条件组合进行TaqMan探针实时定量PCR,一式3份。绘制lgC-Ct标准曲线,并获得相应的回归方程。
1.2.9 重复性实验选择104copies/μL、105copies/μL、106copies/μL的标准模板进行重复性实验,一式3份。每隔3 d重复1次实验。计算组内重复和组间重复变异系数CV,公式如下。
| $ \mathrm{CV}=\mathrm{SD} / \mathrm{MN} \times 100 \% $ |
其中,SD为标准差,MN为平均值。
1.2.10 临床样本的检测为评估本方法是否适用于临床检测,随机取不同年龄段健康人群粪便标本各12例,包括青年人(44岁以下)、中年人(45~59岁)、普通老年人(60~79岁)、高龄老年人(80岁及以上)。志愿者来自上海市宝山区友谊街道社区、宁波某体检机构和复旦大学等,经血液生化检查、尿检和粪检等常规体检项目筛查,未患有基础代谢障碍、免疫缺陷以及慢性消耗性疾病,且近3个月内无任何抗生素使用史。
提取粪便标本总菌群基因组DNA用以检测,取浓度为1~1012 copies/μL的标准品用于标准曲线绘制,以双歧杆菌、肠球菌和大肠杆菌混菌液提取的gDNA为阳性模板,以nuclease-free H2O作为阴性对照,按照优化的反应条件组合,进行TaqMan探针多重实时定量PCR,并以回归方程为参考,获得各目标菌属/科及总肠道菌群的含量,以探讨本方法在临床实际标本中的应用效果。
1.3 统计学方法采用Graphpad Prism(version 8.4.0)对数据进行统计分析。多组比较使用ordinary one way ANOVA检验。ns代表P>0.05,表明差异不具有统计学意义; *代表P < 0.05,* *代表P < 0.01,* * *代表P < 0.001,表明差异具有统计学意义。
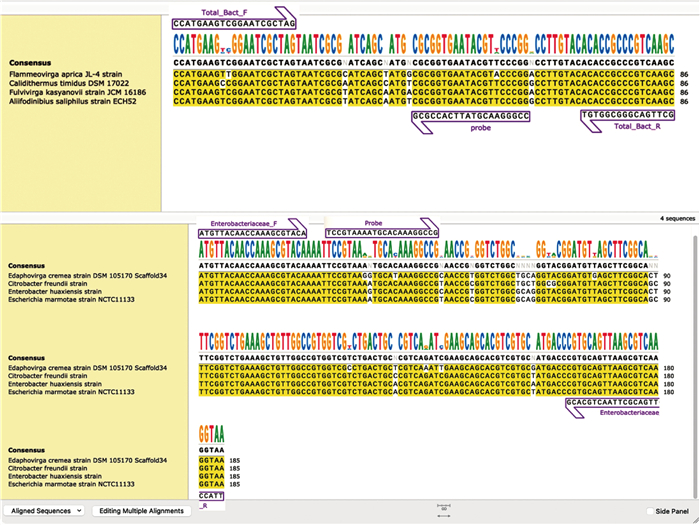
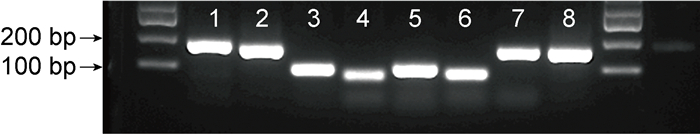
2 结果 2.1 引物和探针特异性设计引物和探针序列如表 3所示。Primer Blast以及Nucleotide Blast结果显示,引物和探针能够与其对应菌属/科的16S rRNA基因序列相结合(见图 1)。PCR和琼脂糖凝胶电泳结果显示,引物和探针验证引物的扩增产物均显示为单一、明亮的条带,大小与预期片段大小一致,无非特异性条带产生(见图 2),证明设计的引物和探针具有良好的特异性。
| Target bacteria | sequence(5′-3′) | |
| All bacteria[19] | F- | CCATGAAGTCGGAATCGCTAG |
| R- | GCTTGACGGGCGGTGT | |
| Probe- | 5′-FAM-CCGGGAACGTATTCACCGCG-BHQ1-3′ | |
| Enterococcus | F- | AGAAATTCCAAACGAACTTGGAG |
| R- | CAGTGCTCTACCTCCATCATT | |
| Probe- | 5′-ROX-TGGTTCTCTCCGAAATAGCTTTAGGGCTA-BHQ2-3′ | |
| Bifidobacterium | F- | ATGCTGGTGTGGAAGAGA |
| R- | TGCTCGCGTCCACTATCCAGT | |
| Probe- | 5′-HEX-TCAAACCACCACGCG CCA-BHQ1-3′ | |
| Enterobacteriaceae[20] | F- | ATGTTACAACCAAAGCGTACA |
| R- | TTACCTTGACGCTTAACTGCACG | |
| Probe- | 5′-CY5-TCCGTAAAATGCACAAAGGCCG-BHQ2-3′ |
|
| Binding sites of primers and probes on template DNA of total gut microbiota (top) and Enterobacteriaceae (bottom). 图 1 引物和探针结合位置示意 Fig. 1 Schematic diagram of binding position of primer and prob |
|
| Each primer and probe verification primer can amplify the product as a single, and bright bands with the same size are as expected. 1.Bifidobacterium; 2.Bifidobacterium-probe; 3.16S; 4.16S-probe; 5.Enterococcus; 6.Enterococcus-probe; 7.Enterobacteriaceae; 8.Enterobacteriaceae-probe. 图 2 引物和探针的特异性 Fig. 2 High specificity of primers and probes |
以重组质粒为模板进行PCR扩增验证,结果显示条带大小与目的片段大小一致,经测序,结果与所需片段同源性一致,表示成功构建阳性质粒,最终获得浓度梯度为1~1012copies/μL的标准模板。
使用不同的引物浓度、探针浓度和循环数进行TaqMan探针多重实时荧光定量PCR,当循环数为40且在表 4所示的最优反应浓度条件下,各引物扩增效率趋于一致且荧光强度最大。确定各目标细菌的检测限为:双歧杆菌46 copies/μL、肠球菌37 copies/μL、肠杆菌51 copies/μL、总菌群7.5×102 copies/μL。
| Target bacteria | Primer concentration (nmol/L) | Probe concentration (nmol/L) |
| Bifidobacterium | 200 | 100 |
| Enterococcus | 150 | 75 |
| Enterobacteriaceae | 150 | 75 |
| All bacteria | 100 | 50 |
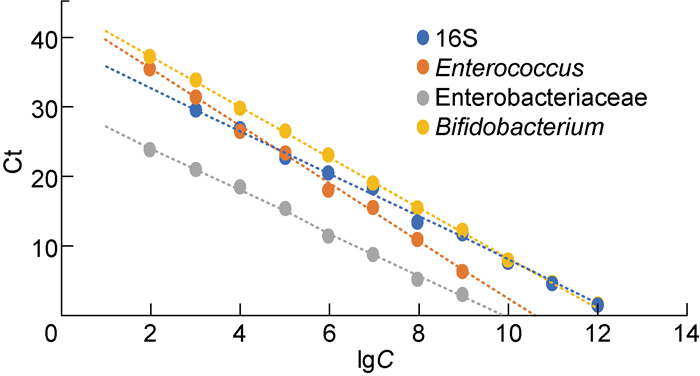
如表 5所示,起始模板量在一定范围内时,拷贝数对数值(lg C)与Ct值具有线性关系,标准曲线拟合度良好,R2值在0.995以上(见图 3)。
| Target bacteria | Regression equation | Linear range (copies/μL) | R2 |
| Enterococcus | y=-4.206 3x+43.741 | 5.9×102~5.9×109 | 0.995 6 |
| Bifidobacterium | y=-3.542 5x+43.779 | 3.1×102~3.1×109 | 0.999 2 |
| Enterobacteriaceae | y=-3.306 2x+41.033 | 6.3×102~6.3×109 | 0.998 8 |
| All bacteria | y=-3.088 8x+38.835 | 2.9×103~2.9×1012 | 0.998 7 |
|
| The linearity of different bacterial standard curves was high with coefficient of determination (R2≥0.995). 图 3 标准曲线 Fig. 3 Standard curves |
组间重复和组内重复实验结果显示,各变异系数均在1.32%以下,表示本方法具有较高的可重复性,结果可信(见表 6)。
| Target bacteria | Starting template amount (copies/μL) | Repeat within group | Repeat between groups | |||||
| SD (copies/μL) | Mean (copies/μL) | CV(%) | SD (copies/μL) | Mean (copies/μL) | CV(%) | |||
| Enterococcus | 5.9×106 | 4.31×104 | 5.82×106 | 0.74 | 5.96×104 | 5.84×106 | 1.02 | |
| 5.9×105 | 3.99×103 | 5.88×105 | 0.68 | 5.50×103 | 5.91×105 | 0.93 | ||
| 5.9×104 | 4.68×102 | 5.93×104 | 0.79 | 5.75×102 | 5.87×104 | 0.98 | ||
| Bifidobacterium | 3.1×106 | 1.41×104 | 3.13×106 | 0.45 | 1.65×104 | 3.18×106 | 0.52 | |
| 3.1×105 | 2.68×103 | 3.05×105 | 0.88 | 3.32×103 | 3.07×105 | 1.08 | ||
| 3.1×104 | 2.92×102 | 3.21×104 | 0.91 | 3.57×102 | 3.22×104 | 1.11 | ||
| Enterobacteriaceae | 6.3×106 | 5.87×104 | 6.24×106 | 0.94 | 8.24×104 | 6.29×106 | 1.31 | |
| 6.3×105 | 2.09×103 | 6.33×105 | 0.33 | 3.87×103 | 6.35×105 | 0.61 | ||
| 6.3×104 | 1.52×102 | 6.32×104 | 0.24 | 2.27×102 | 6.31×104 | 0.36 | ||
| All bacteria | 2.9×106 | 3.34×104 | 2.96×106 | 1.13 | 2.95×104 | 2.98×106 | 0.99 | |
| 2.9×105 | 3.54×103 | 2.83×105 | 1.25 | 3.64×103 | 2.84×105 | 1.28 | ||
| 2.9×104 | 2.74×102 | 2.85×104 | 0.96 | 3.43×102 | 2.86×104 | 1.28 | ||
| SD:standard deviation; CV:coefficient of variation | ||||||||
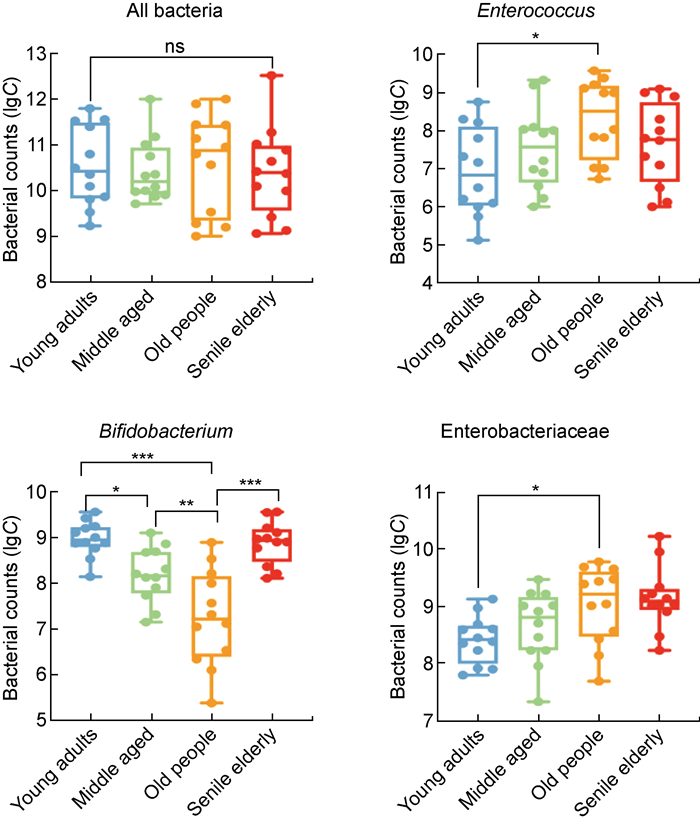
在健康增龄人群中,运用建立的TaqMan探针法检测的肠道菌群总量在各组间无显著差异。肠球菌和肠杆菌含量随增龄呈增长趋势,且老年组显著高于青年组(肠球菌, P=0.017;肠杆菌, P=0.017),在高龄老年人未发现肠球菌数量增加。双歧杆菌含量随增龄减少,且各组间具有显著性差异(青年组和准老年组,P=0.033; 青年组和老年组,P < 0.000 1;准老年组和老年组,P=0.008 2;老年组和高龄老年组,P < 0.000 1;青年组和高龄老年组,P=0.991),在高龄老年人中也未观察到双歧杆菌含量显著减少(见图 4)。
|
|
Using the regression equation of each target bacteria as a reference, the total intestinal flora of each group and the content of Enterococcus, Bifidobacterium, and Enterobacteriaceae were calculated based on the Ct value. From left to right, the abscissas are corresponding to the youth group, the middle aged group, the general elderly group, and the senile elderly group. Ordinary one way ANOVA test is used to test differences between groups, ns means P>0.05, no statistical difference; *means P < 0.05, * *means P < 0.01, * * *means P < 0.001, and the difference is statistically significant. C: copy number, unit: copies/g. 图 4 健康增龄人群肠道细菌绝对含量比较 Fig. 4 Comparison of the absolute quantification of bacteria in healthy aging groups |
本研究主要建立基于TaqMan探针多重qPCR的肠道总菌群以及重要共生菌群绝对定量方法。结果显示:目标菌属/科间不具有交叉反应; 灵敏度高,检测限约为染料法qPCR的10%;时效短,在2 h内即可完成对3类肠道共生菌和总菌群的检测; 具有良好的可重复性,变异系数低于1.32%,与其他基于TaqMan探针qPCR的研究结果相似[21-23]。Hu等[21]采用TaqMan探针qPCR检测194份粪便标本中的福氏志贺菌,发现该方法敏感性达97%,特异性达99%。此外,既往针对肠道菌群定量的研究大多采用细菌计数或染料法qPCR。Sharma等[22]使用平板计数法对婴儿肠道中的需氧菌和厌氧菌进行定量,结果显示菌落计数范围为104~1013 CFU/g粪便; Yao等[23]通过SYBR法qPCR对粪便中拟杆菌、梭菌、梭状芽孢杆菌、双歧杆菌和法氏杆菌16S rDNA的检测实现了细菌定量,检测限为102~103 copies/μL。但此类研究具有一定的局限性,平板计数法对培养基选择、细菌培养条件等方面有较高的要求,且方法的准确性和可重复性低,灵敏度不及qPCR; SYBR法qPCR单孔内只能检测1种靶基因,且直接以DNA为模板不能消除多拷贝的影响。本研究通过建立TaqMan探针多重qPCR法,构建重组质粒作为标准模板,在除去细菌不同基因拷贝数影响的同时,保证实验结果的准确性和稳定性。
在本方法建立的过程中,考虑了各种影响实验准确性的可能因素,其中引物和探针的有效性是保证试验成功的关键。通过Blast以及PCR扩增,本研究验证了引物和探针的特异性,并且对引物序列进行优化,减少因退火温度不同引起的扩增效率差异。此外,由于同一体系内多对引物对底物(酶、Mg2+、dNTP等)的竞争,某些高丰度的目标基因的荧光信号可能出现过早,在其他基因扩增前已达到线性和平台期,则可能使其他基因面临试剂耗竭,从而导致扩增效果较差。在本实验中,通过调整引物和探针的浓度,在最大程度上保证各引物扩增速率的一致性。
本研究利用建立的TaqMan探针多重qPCR法对临床标本进行检测,结果显示健康人群粪便标本中双歧杆菌、肠球菌及肠杆菌的丰度随增龄的变化趋势与文献基本一致[24-27]。双歧杆菌随着增龄而逐渐减少,肠杆菌轻度增多,B/E值的降低可能与老年人肠道菌群的定植抗性降低以及对病原的易感有关[28-29]。由于并非来自同一人群的队列研究,而是较小规模的横断面研究,对高龄老年组的菌群变化与普通老年组不同的合理解释,可能并非源自增龄相关的菌群变化规律,而更可能由高龄老年潜在的长寿影响因素决定。既往针对长寿老人的研究发现,他们肠道中的双歧杆菌、阿克曼菌和毛螺菌等“有益菌”含量较普通老年人高,而“有害菌”含量更少,多样性更高[30]。本研究在一定程度上支持了这一结论。
总体而言,本研究建立的肠道共生菌群定量检测方法适用于实际标本,可以提高主要菌群定量检测的时效和灵敏度,并有效减少标本用量,特别是珍贵的临床标本。根据本方法的思路,可继续开发针对其他目标菌群的定量方法,例如:肠道中有重要研究意义但培养条件严苛的厌氧菌; 可用于总菌含量更低的标本,如血液、唾液、鼻咽拭子等。另外,TaqMan探针qPCR的多重分析特性,有助于在细菌定量研究中实现样本的高通量检测。2016年《柳叶刀》杂志报道的一项病例对照研究,使用该方法对11 400例粪便标本完成32种肠道病原菌的定量检测[31]。基于此特性,本研究建立的方法可作为高通量测序结果的实验室补充验证手段。另外,未来或许可以开发针对肠道共生菌检测的TaqMan芯片。然而,本研究建立的方法也具有一定的局限性,单孔内的检测靶标越多,引物间干扰越大,结果偏差出现的风险也越高。因此,可能须对单重扩增和多重扩增进行差异分析比较,以确保方法的可信度。
综上所述,本研究建立了一种简单有效的菌群定量方法,适用于测序结果的验证以及临床推广,旨在实现对菌群稳定性和健康程度的快速评估。
| [1] |
Adak A, Khan MR. An insight into gut microbiota and its functionalities[J]. Cell Mol Life Sci, 2019, 76(3): 473-493.
[DOI]
|
| [2] |
Angelucci F, Cechova K, Amlerova J, Hort J. Antibiotics, gut microbiota, and Alzheimer's disease[J]. J Neuroinflammation, 2019, 16(1): 108.
[DOI]
|
| [3] |
Pickard JM, Zeng MY, Caruso R, Núñez G. Gut microbiota: role in pathogen colonization, immune responses, and inflammatory disease[J]. Immunol Rev, 2017, 279(1): 70-89.
[DOI]
|
| [4] |
Illiano P, Brambilla R, Parolini P. The mutual interplay of gut microbiota, diet and human disease[J]. FEBS J, 2020, 287(5): 833-855.
[DOI]
|
| [5] |
Gomaa EZ. Human gut microbiota/microbiome in health and diseases: a review[J]. Antonie Van Leeuwenhoek, 2020, 113(12): 2019-2040.
[DOI]
|
| [6] |
van Hal SJ, Willems RJL, Gouliouris T, Ballard SA, Coque TM, Hammerum AM, Hegstad K, Pinholt M, Howden BP, Malhotra-Kumar S, Werner G, Yanagihara K, Earl AM, Raven KE, Corander J, Bowden R, Enterococcal Study Group. The interplay between community and hospital Enterococcus faecium clones within health-care settings: a genomic analysis[J]. Lancet Microbe, 2022, 3(2): e133-e141.
[DOI]
|
| [7] |
Cattoir V, Giard JC. Antibiotic resistance in Enterococcus faecium clinical isolates[J]. Expert Rev Anti Infect Ther, 2014, 12(2): 239-248.
[DOI]
|
| [8] |
Duan Y, Llorente C, Lang S, Brandl K, Chu H, Jiang L, White RC, Clarke TH, Nguyen K, Torralba M, Shao Y, Liu J, Morales AH, Lessor L, Rahman IR, Miyamoto Y, Ly M, Gao B, Sun WZ, Kiesel R, Hutmacher F, Lee S, Cots MV, Padilla FB, Verna EC, Abraldes JG, Brown RS, Vargas V, Altamirano J, Caballería J, Shawcross DL, Ho SB, Louvet A, Lucey MR, Mathurin P, Tsao GG, Bataller R, Tu XM, Eckmann L, Donk WAV, Young R, Lawley TD, Stärkel P, Pride D, Fouts DE, Schnabl B. Bacteriophage targeting of gut bacterium attenuates alcoholic liver disease[J]. Nature, 2019, 575(7783): 505-511.
[DOI]
|
| [9] |
Luo J, Li Y, Xie J, Goo L, Liu L, Ou S, Chen L, Peng X. The primary biological network of Bifidobacterium in the gut[J]. FEMS Microbiol Lett, 2018, 365(8).
[DOI]
|
| [10] |
Zhang Y, Wang Q, Yin Y, Chen H, Jin L, Gu B, Xie L, Yang C, Ma X, Li H, Li W, Zhang X, Liao K, Man S, Wang S, Wen H, Li B, Guo Z, Tian J, Pei F, Liu L, Zhang L, Zou C, Hu T, Cai J, Yang H, Huang J, Jia X, Huang W, Cao B, Wang H. Epidemiology of carbapenem-resistant Enterobacteriaceae infections: report from the China CRE Network[J]. Antimicrob Agents Chemother, 2018, 62(2): e01882-17.
[DOI]
|
| [11] |
吴仲文, 李兰娟, 马伟杭, 俞云松, 陈亚岗. 肠道微生物定植抗力的新指标——B/E值[J]. 浙江预防医学, 2000(7): 4-5. [CNKI]
|
| [12] |
Karthiga N, Chee LK. Molecular biology-based methods for quantification of bacteria in mixed culture: perspectives and limitations[J]. Appl Microbiol Biotechnol, 2014, 98(16): 6907-6919.
[DOI]
|
| [13] |
Muyzer G. DGGE/TGGE a method for identifying genes from natural ecosystems[J]. Current Opinion in Microbiology, 1999, 2(3): 317-322.
[DOI]
|
| [14] |
Reuter JA, Spacek DV, Snyder MP. High-throughput sequencing technologies[J]. Mol Cell, 2015, 58(4): 586-597.
[DOI]
|
| [15] |
陈旭, 齐凤坤, 康立功, 李景富. 实时荧光定量PCR技术研究进展及其应用[J]. 东北农业大学学报, 2010, 41(8): 148-155. [CNKI]
|
| [16] |
Huang X, Chen J, Yao G, Guo Q, Wang J, Liu G. A TaqMan-probe-based multiplex real-time RT-qPCR for simultaneous detection of porcine enteric coronaviruses[J]. Appl Microbiol Biotechnol, 2019, 103(12): 4943-4952.
[DOI]
|
| [17] |
Xu X, Yang F, Zhang Q, Xu Y, Huang J, Fu M, Zhang W. Development of a multiplex TaqMan qPCR assay for simultaneous detection and differentiation of four DNA and RNA viruses from clinical samples of sheep and goats[J]. J Virol Methods, 2019, 266: 58-64.
[DOI]
|
| [18] |
Carossino M, Barrandeguy ME, Erol E, Li Y, Balasuriya UBR. Development and evaluation of a one-step multiplex real-time TaqMan RT-qPCR assay for the detection and genotyping of equine G3 and G14 rotaviruses in fecal samples[J]. Virol J, 2019, 16(1): 49.
[DOI]
|
| [19] |
Vandeputte D, Kathagen G, D'hoe K, Vieira-Silva S, Colomer MV, Sabino J, Wang J, Tito RY, Commer LD, Darzi Y, Vermeire S, Falony G, Raes J. Quantitative microbiome profiling links gut community variation to microbial load[J]. Nature, 2017, 551(7681): 507-511.
[DOI]
|
| [20] |
Takahashi H, Saito R, Miya S, Tanaka Y, Miyamura N, Kuda T, Kimura B. Development of quantitative real-time PCR for detection and enumeration of Enterobacteriaceae[J]. Int J Food Microbiol, 2017, 246: 92-97.
[DOI]
|
| [21] |
Hu Z, Benkoulouche M, Barel AL, Heiget GL, Imeddourene AB, Guen YL, Monties N, Guerreiro C, Remaud-Siméon M, Moulis C, André I, Mulard LA. Convergent chemoenzymatic strategy to deliver a diversity of Shigella flexneri serotype-specific o-antigen segments from a unique lightly protected tetrasaccharide core[J]. J Org Chem, 2021, 86(3): 2058-2075.
[DOI]
|
| [22] |
Sharma N, Chaudhry R, Panigrahi P. Quantitative and qualitative study of intestinal flora in neonates[J]. J Glob Infect Dis, 2012, 4(4): 188-192.
[DOI]
|
| [23] |
Yao P, Cui M, Wang H, Gao H, Wang L, Yang T, Cheng Y. Quantitative analysis of intestinal flora of uygur and han ethnic chinese patients with ulcerative colitis[J]. Gastroenterol Res Pract, 2016, 9186232.
[DOI]
|
| [24] |
Arboleya S, Watkins C, Stanton C, Ross RP. Gut Bifidobacteria populations in human health and aging[J]. Front Microbiol, 2016, 7: 1204.
[DOI]
|
| [25] |
Mueller S, Saunier K, Hanisch C, Norin E, Alm L, Midtvedt T, Cresci A, Silvi S, Orpianesi C, Verdenelli MC, Clavel T, Koebnick C, Zunft HJ, Doré J, Blaut M. Differences in fecal microbiota in different European study populations in relation to age, gender, and country: a cross-sectional study[J]. Appl Environ Microbiol, 2006, 72(2): 1027-1033.
[DOI]
|
| [26] |
Tedim AP, Ruiz-Garbajosa P, Corander J, Rodríguez CM, Cantón R, Willems RJ, Baquero F, Coque TM. Population biology of intestinal enterococcus isolates from hospitalized and nonhospitalized individuals in different age groups[J]. Appl Environ Microbiol, 2015, 81(5): 1820-1831.
[DOI]
|
| [27] |
Mei LH, Zheng WX, Zhao ZT, Meng N, Zhang QR, Zhu WJ, Li RD, Liang XL, Li QY. A pilot study of the effect of Lactobacillus casei obtained from long-lived elderly on blood biochemical, oxidative, and inflammatory markers, and on gut microbiota in young volunteers[J]. Nutrients, 2021, 13(11): 3891.
[DOI]
|
| [28] |
Yu HJ, Jing C, Xiao N, Zang XM, Zhang CY, Zhang X, Qu YN, Li Y, Tan QW. Structural difference analysis of adult's intestinal flora basing on the 16S rDNA gene sequencing technology[J]. Eur Rev Med Pharmacol Sci, 2020, 24(24): 12983-12992.
|
| [29] |
Vemuri R, Gundamaraju R, Shastri MD, Shukla SD, Kalpurath K, Ball M, Stephen T, Shankar EM, Ahuja K, Eri R. Gut microbial changes, interactions, and their implications on human lifecycle: an ageing perspective[J]. Biomed Res Int, 2018, 4178607.
[DOI]
|
| [30] |
Kong F, Deng F, Li Y, Zhao J. Identification of gut microbiome signatures associated with longevity provides a promising modulation target for healthy aging[J]. Gut microbes, 2019, 10(2): 210-215.
[DOI]
|
| [31] |
Liu J, Platts-Mills JA, Juma J, Kabir F, Nkeze J, Okoi C, Operario DJ, Uddin J, Ahmed S, Alonso PL, Antonio M, Becker SM, Blackwelder WC, Breiman RF, Faruque AS, Fields B 8, Gratz J, Haque R, Hossain A, Hossain MJ, Jarju S, Qamar F, Iqbal NT, Kwambana B, Mandomando I, McMurry TL, Ochieng C, Ochieng JB, Ochieng M, Onyango C, Panchalingam S, Kalam A, Aziz F, Qureshi S, Ramamurthy T, Roberts JH, Saha D, Sow SO, Stroup SE, Sur D, Tamboura B, Taniuchi M, Tennant SM, Toema D, Wu Y, Zaidi A, Nataro JP, Kotloff KL, Levine MM, Houpt ER. Use of quantitative molecular diagnostic methods to identify causes of diarrhoea in children: a reanalysis of the GEMS case-control study[J]. Lancet, 2016, 388(10051): 1291-1301.
[DOI]
|
 2022, Vol. 17
2022, Vol. 17


