鼠肝炎病毒(mouse hepatitis virus,MHV)即鼠冠状病毒(murine coronavirus),属于乙型冠状病毒属(Betacoronavirus,β -CoV),与严重急性呼吸综合征冠状病毒(severe acute respiratory syndrome coronavirus,SARS-CoV)、中东呼吸综合征冠状病毒(Middle East respiratory syndrome coronavirus,MERS-CoV)和严重急性呼吸综合征冠状病毒2(severe acute respiratory syndrome coronavirus 2,SARS-CoV-2)为同一属的不同谱系。MHV不感染人类,对实验人员具有很高的安全性,是目前研究冠状病毒的模式病毒。该病毒于1949年首次被报道[1],根据组织嗜性不同,可分为呼吸株[respiratory strain; 又称多嗜株(polytropic strain)]和肠嗜株(enterotropic strain)[2]。实验室中研究较多的A59毒株是从患有白血病的小鼠体内分离出来的,具有弱的神经毒性和温和的嗜肝性,属于呼吸株[3]。
MHV是正链RNA病毒,基因组全长约31 kb,具有5’帽子结构和poly(A)尾。基因组的前2/3是开放阅读框(open reading frame,ORF)1a和1b,翻译出多聚蛋白(polyprotein,pp)1a和1ab,编码病毒RNA复制和转录所需的16个非结构蛋白[4]。基因组后1/3包含7个ORF,其中ORF2a、ORF4和ORF5a分别编码种属特异性的辅助蛋白ns2、ns4和ns5a[5-7]。ORF2b编码血凝素酯酶(hemagglutinin esterase,HE),具有唾液酸结合和乙酰酯酶活性[8]。ORF3编码的刺突蛋白(spike protein,S)可与细胞受体癌胚抗原相关细胞黏附分子1(carcinoembryonic antigen-related cell adhesion molecule 1,CEACAM-1)结合,并决定病毒的细胞嗜性[9]。ORF5b、ORF6和ORF7分别编码包膜蛋白(envelope protein,E)、膜蛋白(membrane protein,M)和核衣壳蛋白(nucleocapsid protein,N)[10]。在N基因内还含有编码I蛋白(internal protein)的ORF[11]。
反向遗传操作系统是研究病毒基因组功能、构建重组病毒的重要工具,能帮助更好地理解病毒的生物学特性,亦可用作基因传递载体和表达系统[12-13]。1992年,Masters等首先基于RNA靶向重组技术和病毒温度敏感表型,对不耐热的亲本病毒进行识别筛选,开发出了MHV反向遗传系统[14-15]。2000年,Kuo等[16]利用病毒的宿主嗜性,通过病毒S基因的替换改进了这一系统。2002年,Yount等[17]基于体外连接技术,开发了一种由7个片段构成全长感染性cDNA克隆的MHV反向遗传系统,此系统需要构建质粒并在大肠埃希菌中表达,但病毒基因组中含有毒性序列,高拷贝数时对大肠埃希菌有毒性。2005年,Coley等[18]使用痘苗病毒作为真核表达系统,构建了MHV反向遗传系统,并且可通过痘苗病毒介导的同源重组进行突变。最近,Thi Nhu Thao等[19]建立了基于酵母人工染色体(yeast artificial chromosome,YAC)系统的RNA病毒拯救平台并拯救出MHV,此系统利用了酿酒酵母体内高效的同源重组系统,无须考虑酶切位点的选择。在特定的实验环境下,上述反向遗传系统各有优劣,须综合考虑实验因素选择合适的病毒拯救策略。
在众多反向遗传系统中,体外连接是最经典和相对简单、直接的方法。本研究优化了Yount等构建的反向遗传操作系统,利用ⅡS类限制性内切酶BsmBⅠ和BsaⅠ,成功构建了MHV A59毒株的六质粒系统,通过观察细胞病变效应(cytopathic effect,CPE)和生长曲线等,证明拯救的重组病毒与原始毒株的生物学特性一致。相较于之前的七质粒系统,本研究优化的六质粒系统未对病毒基因组序列进行任何同义突变,完整保留了原始毒株的序列,简化了实验操作流程,也提高了连接和拯救效率; 还构建了含ZsGreen(Zoanthus sp. green fluorescent protein)或Nluc(NanoLuc luciferase)基因的报告病毒,为后续研究MHV的生物学特性、测试抗病毒药物等提供了强有力的工具。
1 材料和方法 1.1 材料 1.1.1 细胞和质粒BHK-R(稳定表达MHV受体CEACAM-1的BHK细胞)和L2细胞均为美国宾夕法尼亚大学医学院Susan Weiss教授馈赠。MHV原始毒株A59(wtA59)由本单位测序并保存[20]。大肠埃希菌Stabl3感受态细胞购自Thermo公司。pSMART载体质粒购自美国Lucigen公司。
1.1.2 试剂主要试剂有NEBNext Ultra II Q5®预混液(北京NEB公司,M0544L)、限制性内切酶BsmBⅠ-v2(北京NEB公司,R0739L)、限制性内切酶BsaⅠ-HF v2(北京NEB公司,R3733L)、T4 DNA连接酶(北京NEB公司,M0202L)、总RNA提取试剂盒[天根生化科技(北京)有限公司,DP419]、普通琼脂糖凝胶DNA回收试剂盒[天根生化科技(北京)有限公司,DP209]、无内毒素质粒大提试剂盒[天根生化科技(北京)有限公司,DP120],QIAquick Gel Extraction Kit(德国QIAGEN公司,28706)、In-Fusion® HD Cloning Kit(TaKaRa公司,639650)、1 st Strand cDNA Synthesis Kit(日本TaKaRa公司,6110A)、AxyPrep质粒DNA小量试剂盒[康宁生命科学(吴江)有限公司,AP-MN-P-250]、mMESSAGE mMACHINETM T7 Transcription Kit[英潍捷基(上海)贸易有限公司,AM1344]、辣根过氧化物酶标记山羊抗小鼠IgG(美国Sigma公司,A4416-1ML)、TrueBlueTM Peroxidase Substrate(英国SeraCare公司,5510-0050)、Nano-Glo® Luciferase Assay System(美国Promega公司,N1110)。小鼠抗MHV-N抗体由本单位制备。
1.2 方法 1.2.1 MHV全基因组分段扩增用总RNA提取试剂盒抽提MHV原始毒株A59(wtA59)细胞培养上清液中的病毒RNA,并用PrimeScriptTM 1 st Strand cDNA Synthesis Kit将病毒RNA反转录为cDNA,于-80 ℃保存。根据全基因组序列、酶切位点分布及DNA聚合酶适宜扩增长度,将全基因组分为6个片段进行扩增。在A片段5’端引物中引入T7启动子序列,F2片段3’端引物中引入poly(A)尾,相邻引物间使用“No See ′ m”的BsmBⅠ或BsaⅠ酶切位点[17]。经过反转录聚合酶链式反应(reverse transcription-polymerase chain reaction,RT-PCR),获得A、B、C、D、E、F片段(扩增程序见表 1),产物经琼脂糖凝胶电泳后,用普通琼脂糖凝胶DNA回收试剂盒纯化。
| Steps | Temperature (℃) | Time | Cycles |
| Initial denaturation | 98 | 30 s | 1 |
| Denaturation | 98 | 10 s | |
| Annealing | 56 | 30 s | 30 |
| Extension | 72 | 3 min | |
| Final extension | 72 | 2 min | 1 |
通过PCR扩增pSMART质粒获得线性化载体。用In-Fusion® HD Cloning Kit将目的基因片段和载体片段无缝连接,获得分别含A、B、C、D、E、F片段的6个质粒。构建的质粒经PCR鉴定为阳性后送苏州金唯智生物科技有限公司进行测序。
1.2.3 MHV全长cDNA克隆的构建将构建的6个质粒分别用BsmBⅠ或BsaⅠ酶切线性化(详见图 1),经琼脂糖凝胶电泳后,用QIAquick Gel Extraction Kit进行胶回收,回收产物用T4 DNA连接酶进行体外连接,用氯仿-异丙醇-乙醇抽提法抽提核酸,获得A59全长感染性cDNA克隆(infectious cDNA clone of A59,icA59)。
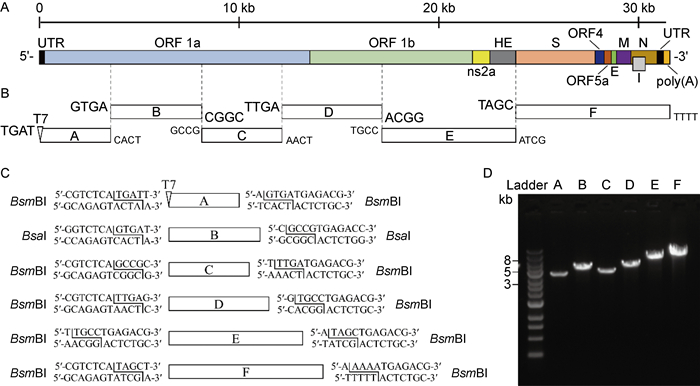
|
| A: Genome structure of MHV A59. B: Strategy for in vitro assembly of an infectious cDNA clone of MHV A59. C: Diagram of the terminal sequences of each cDNA fragment recognized by BsmBI and BsaI. D: Gel analysis of the six purified cDNA fragments. The 1-kb DNA ladders are indicated. 图 1 MHV A59毒株全长感染性克隆的构建框架 Fig. 1 Schematic of the construction of full-length infectious clone of MHV A59 strain |
以构建的F质粒为骨架,去掉ORF4前的3对核苷酸,获得SalⅠ酶切位点,在ORF4内第281位核苷酸后引入NotⅠ酶切位点[21]。将该质粒酶切后,分别与ZsGreen和Nluc基因片段进行无缝连接,获得带有ZsGreen或Nluc基因的重组F-ZsGreen和F-Nluc质粒。按照1.2.3中的方法进行后续酶切和连接,获得icA59-ZsGreen和icA59-Nluc全长cDNA。
1.2.5 病毒的拯救本实验使用mMESSAGE mMACHINETM T7 Transcription Kit对cDNA进行体外转录,获得基因组全长RNA,电转BHK-R细胞。每天观察拯救病毒感染细胞后的CPE,当50%的细胞出现CPE时,收集上清并保存于-80 ℃。
1.2.6 噬斑和CPE实验将原始毒株和拯救的重组病毒按照感染复数(multiplicity of infection,MOI)为0.01分别感染6孔板中的L2细胞,感染后2 h每孔加入3 mL 2%低熔点琼脂糖与2×DMEM的混合物,室温放置20 min后放入培养箱孵育。48 h后,观察CPE,并用结晶紫染色15 min,观察病毒噬斑的大小及形态。
1.2.7 病毒滴度测定采用斑点形成实验(focus forming assay,FFA)测定病毒滴度[22],具体步骤如下。将L2细胞铺于96孔细胞培养板中,待细胞汇合度达到100%时,将病毒用含2%胎牛血清(fetal bovine serum,FBS)的培养基进行10倍梯度稀释,每个稀释度进行3个重复,每孔加100 μL病毒液,在培养箱中孵育2 h后,每孔加入125 μL甲基纤维素溶液与2×DMEM的混合物,继续放回培养箱中孵育16 h,用4%组织细胞固定液固定30 min。与1∶40 000稀释的小鼠抗MHV-N抗体孵育2 h后,用磷酸缓冲盐溶液(phosphate buffered saline, PBS)洗3遍,然后与1∶2 500稀释的辣根过氧化物酶标记山羊抗小鼠IgG孵育2 h,用PBS洗3遍,最后用TrueBlueTM Peroxidase Substrate染色30 min,在显微镜下对蓝色斑点计数。
1.2.8 多步生长曲线的测定将原始毒株和拯救的重组病毒按照MOI为0.01分别感染12孔板中的L2细胞,感染后2 h弃去含病毒的上清,用培养基洗3遍,每孔加1 mL培养基。收集感染后2、8、12、16和24 h的上清,按照1.2.7中的方法测定病毒滴度,每个时间点均有3个复孔,绘制病毒多步生长曲线。
1.2.9 药物抑制实验将L2细胞铺到96孔板中,用药物处理2 h后加入报告病毒,在感染后12 h和24 h分别检测绿色荧光强度和荧光素酶活性。检测荧光素酶活性时,弃去病毒上清,加入30 μL含2% FBS的培养基和30 μL Nano-Glo® Luciferase Reagent,室温孵育15 min,将孔中的液体全部转移至白色不透明板中,用Flex Station 3多功能微孔板读板机进行读值。
1.3 统计学分析使用GraphPad Prism 8软件,采用ANOVA或unpaired t-test进行组间差异分析,P < 0.05为有统计学差异,P < 0.005为有显著的统计学差异,P < 0.001为有极显著的统计学差异。
2 结果 2.1 成功构建MHV全长cDNA克隆从原始病毒wtA59感染的细胞上清中提取RNA,作为RT-PCR模板,制备cDNA片段。通过设计构建6个连续的cDNA片段,以覆盖整个病毒基因组(见图 1B),这些cDNA片段全部通过RT-PCR制备,然后克隆到pSMART质粒载体中。每个cDNA片段两侧都有一个ⅡS类限制性内切酶位点(BsmBⅠ或BsaⅠ),以产生特异的黏性末端(见图 1C),然后正确连接成全长cDNA。本研究采用一种称为“No See ′ m”的“隐形克隆”策略[17],使回收片段不含内切酶识别位点。图 1C描述了这6个片段的详细信息:A(T7启动子序列加1~3 513位核苷酸)、B(3 514~8 095位核苷酸)、C(8 096~12 109位核苷酸)、D(12 110~17 132位核苷酸)、E(17 133~23 850位核苷酸)和F [23 851~31 333位核苷酸加poly(A)序列]。
将6个cDNA片段分别克隆到质粒载体中,并对它们进行测序以确保序列正确。然后用BsmBⅠ或BsaⅠ酶切这6个质粒,将得到的DNA片段通过琼脂糖凝胶回收(见图 1D),在体外连接成全长基因组cDNA,纯化后进行体外转录,生成病毒全长基因组RNA。
2.2 重组病毒与原始毒株复制特性相似为了从icA59中拯救出重组A59(recombinant A59,rA59)毒株,本研究直接将体外转录的基因组RNA电转至BHK-R细胞中。有报道称,N蛋白可增强冠状病毒RNA转录物的感染性[17, 23-24],但本研究未同时电转编码A59 N蛋白的RNA。细胞在电转后12 h就出现了以合胞体为典型特征的CPE,成功拯救出有感染性的重组病毒。对比重组病毒(rA59)与原始毒株(wtA59)的复制特性,rA59形成的CPE和噬斑形态与wtA59相似(见图 2A和2B),且在L2细胞中表现出相似的生长曲线(见图 2C)。以上结果表明,体外转录的全长RNA在电转进入细胞后具有感染性,且重组病毒重现了原始毒株在L2细胞中的复制特性。
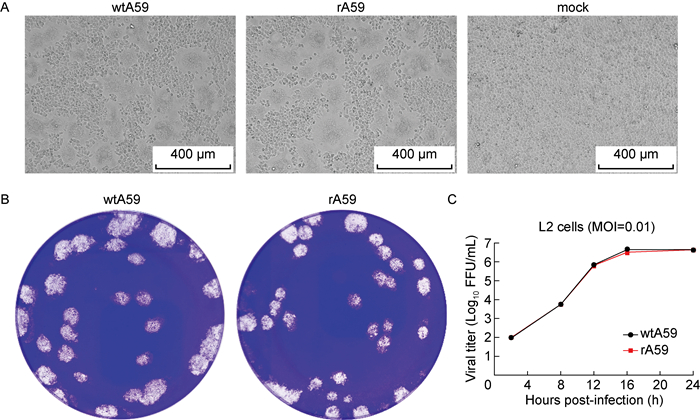
|
| A: Bright-field images of the original wild-type wtA59 and the recombinant rA59. B: Plaque morphology of the original wtA59 and the recombinant rA59. C: Growth curve of the original wtA59 and the recombinant rA59 in L2 cells at an MOI of 0.01. 图 2 重组病毒(rA59)与原始毒株(wtA59)复制特性比较 Fig. 2 Comparison of replication characteristics between recombinant virus (rA59) and original wild-type virus (wtA59) |
表达报告基因(即荧光蛋白或化学发光)的报告病毒是研究病毒复制、组织嗜性、发病机制以及筛选药物的有力工具。为了构建报告病毒,本研究将ZsGreen和Nluc基因分别插入病毒基因组的ORF4中(见图 3A)。采用与上述icA59相同的体外连接方法制备icA59-ZsGreen和icA59-Nluc全长cDNA克隆,经体外转录和电转基因组RNA,获得rA59-ZsGreen和rA59-Nluc重组病毒。为了检查报告基因是否抑制了病毒复制,本研究比较了携带报告基因的病毒与其原始重组病毒在L2细胞中的复制特性。结果显示,rA59-ZsGreen和rA59-Nluc报告病毒感染L2细胞后,产生了与原始重组病毒rA59相似的CPE和噬斑形态(见图 3B和3C)。rA59-Nluc与rA59的多步生长曲线几乎完全一致,而rA59-ZsGreen与rA59的多步生长曲线略有差异,但不显著(见图 3D)。以上结果表明,rA59-ZsGreen和rA59-Nluc病毒所携带的报告基因未显著影响病毒复制,可以很好地反映rA59病毒的复制特性。
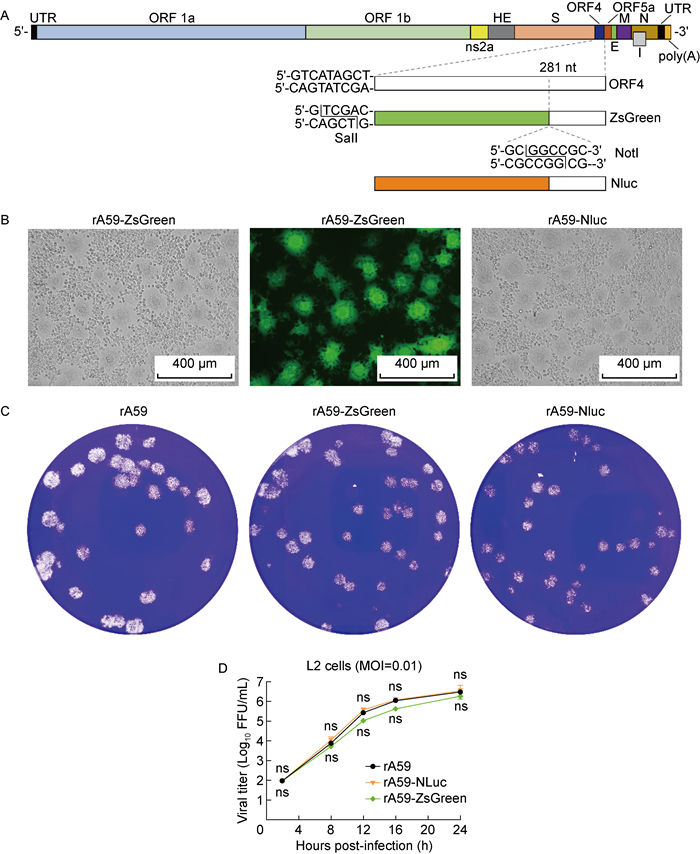
|
| A: Assembly of the rA59-ZsGreen and rA59-Nluc cDNA. The ORF4 sequence is replaced by ZsGreen or Nluc gene. B: Fluorescence microscopy and light microscopy of virus-infected cells. Representative images are shown. C: Plaque morphology of the rA59-ZsGreen and rA59-Nluc viruses. D: Growth curve of the rA59, reporter viruses rA59-ZsGreen and rA59-Nluc in L2 cells at an MOI of 0.01. 图 3 rA59-ZsGreen和rA59-Nluc报告病毒的拯救 Fig. 3 Rescue of reporter viruses rA59-ZsGreen and rA59-Nluc |
为了探索rA59-ZsGreen和rA59-Nluc的应用价值,使用这两种病毒验证瑞德西韦(remdesivir)的抗病毒活性。瑞德西韦是一种核苷酸类似物前体药物,在细胞培养和小鼠模型中对人冠状病毒和人畜共患冠状病毒表现出广谱的抗病毒活性[25]。用10 μmol/L瑞德西韦预处理L2细胞2 h,加入病毒(药物一直存在),在感染后12 h和24 h分别进行检测(见图 4A)。与对照组二甲基亚砜(dimethyl sulfoxide,DMSO)比较,rA59-ZsGreen感染后的荧光细胞可被药物明显抑制(见图 4B)。药物处理同样显著抑制了荧光素酶活性,表明rA59-Nluc病毒的复制水平降低(见图 4C)。与传统的噬斑测定相比,报告病毒检测更加快速便捷。以上结果表明,rA59-ZsGreen和rA59-Nluc可用于高效的抗病毒药物筛选平台。
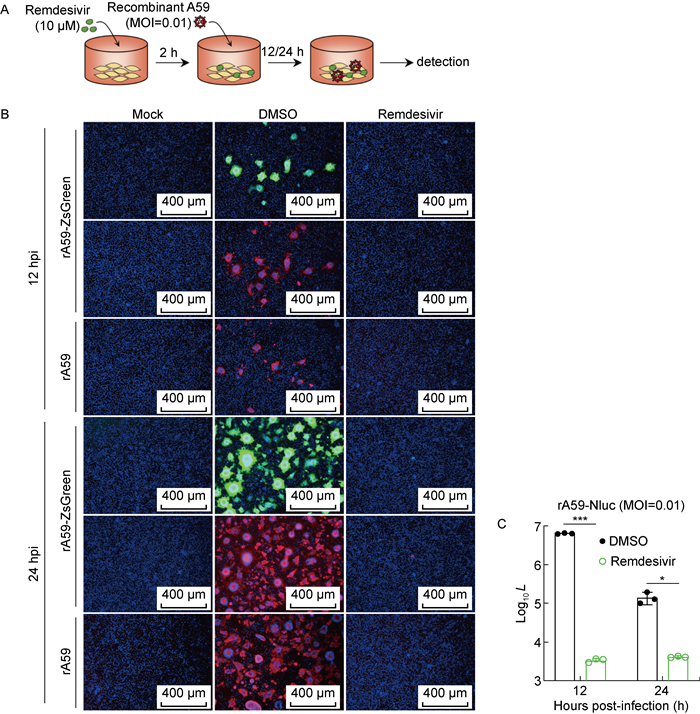
|
| A: Schematic diagram of remdesivir treatment. B: Representative fluorescent images of virus-infected L2 cells after remdesivir treatment. Color representations are as follows: green, ZsGreen; red, N protein; blue, nucleus. C: Luciferase signal produced from virus-infected L2 cells after remdesivir treatment. L: Luminescence. 图 4 rA59-ZsGreen和rA59-Nluc验证瑞德西韦的抗病毒活性 Fig. 4 Validation of antiviral activity of remdesivir using reporter viruses rA59-ZsGreen and rA59-Nluc |
小鼠不仅是研究炎症、免疫反应和感染的经典模型,而且可用于开发新的诊断、预防和治疗方法。MHV是天然感染小鼠的冠状病毒,是研究冠状病毒嗜性、毒力和免疫应答等发病机制的优秀模型,已被广泛用作模型冠状病毒来研究人类肝炎和多发性硬化症(multiple sclerosis,MS)等脱髓鞘疾病[26-27]。MHV ns2和MERS-CoV NS4b都具有2 ′,5 ′ -磷酸二酯酶(2 ′, 5 ′ -phosphodiesterase,2 ′, 5 ′ -PDE)活性,可切割2 ′,5 ′ -寡腺苷酸(2 ′, 5 ′ -oligoadenylate,2-5A),从而阻止核糖核酸酶L(ribonuclease L,RNase L)的激活[28]。同属于β -CoV的MHV和SARS-CoV-2含有相似的基因组结构,且编码相似的蛋白,特别是非结构蛋白。虽然两者的受体利用存在差异,但MHV受体的细胞表达谱与SARS-CoV-2类似,MHV可感染肺部并诱导与SARS-CoV-2相似的炎症反应和病理变化[29]。因此,MHV也被用作研究MERS-CoV和SARS-CoV-2的模型。此外,操作MHV只需要二级生物安全防护,而操作SARS-CoV-2等高致病性病毒需要三级生物安全防护。因此,深入研究MHV-A59病毒为更好地了解高致病性冠状病毒提供了有力帮助。
MHV A59毒株基因组内含有毒性序列,高拷贝数时对大肠埃希菌有毒性,可能导致序列突变和缺失,所以构建质粒时必须要考虑毒性序列所在区域。此外,之前的七质粒系统须对MHV A59毒株基因组序列原有的3处BsmBⅠ酶切位点进行同义突变,而本研究优化的六质粒反向遗传操作系统直接利用了基因组序列中的2处BsmBⅠ酶切位点,并使用BsaⅠ来避开基因组序列中剩余1处BsmBⅠ酶切位点在酶切质粒时的影响,从而完整保留了原始毒株的基因组序列,简化了实验操作流程,也提高了连接和拯救的效率。
本研究成功构建了基于六质粒的MHV A59毒株全长感染性克隆。与原始毒株相比,重组病毒rA59在噬斑形态和生长曲线等方面没有差异。因此,该重组病毒可替代原始毒株,也可通过操作基因组突变病毒来研究突变对病毒的影响。本研究还拯救了rA59-ZsGreen和rA59-Nluc两株报告病毒,通过测定ZsGreen和Nluc的表达情况以反映病毒的复制能力。Nluc能定量测定其催化反应的化学发光值,而且在细胞内没有背景,可检测到非常微弱的表达,灵敏度很高,适用于高通量筛选[30],还可用于研究病毒的小鼠组织器官嗜性和感染路径。不过,rA59-Nluc携带的Nluc需要额外的底物才能激活,因此不适合在显微镜下观察细胞间的病毒复制,而rA59-ZsGreen携带的ZsGreen荧光标记可清楚直观地观察病毒的感染扩散情况。用瑞德西韦处理后,两种报告病毒都可快速有效地验证抗病毒效果。因此,本研究中构建的rA59-ZsGreen和rA59-Nluc可满足不同实验场景下的使用需求。
有研究报道,冠状病毒的基因组可容纳10%~20%的外源基因插入[31-32],而基因组中20%~30%的区域可被替换,甚至仅须复制酶基因就可介导基因组mRNA转录[33-34]。因此,本研究建立的MHV A59反向遗传操作系统不仅可构建报告病毒,用作高通量药物筛选和中和抗体测定的工具,还可作为模式病毒,研究MHV及其他冠状病毒的基因组功能和生物学特性,甚至可作为病毒载体,在受体细胞和体内高效表达异源蛋白来研究其功能。
| [1] |
Cheever FS, Daniels JB, Pappenheimer AM, Bailey OT. A murine virus (JHM) causing disseminated encephalomyelitis with extensive destruction of myelin[J]. J Exp Med, 1949, 90(3): 181-210.
[DOI]
|
| [2] |
Kyuwa S, Ohsawa K, Sato H, Urano T. Replication of enterotropic and polytropic murine coronaviruses in cultured cell lines of mouse origin[J]. Exp Anim, 2000, 49(4): 251-257.
[DOI]
|
| [3] |
Manaker RA, Piczak CV, Miller AA, Stanton MF. A hepatitis virus complicating studies with mouse leukemia[J]. J Natl Cancer Inst, 1961, 27: 29-51.
[PubMed]
|
| [4] |
Lee HJ, Shieh CK, Gorbalenya AE, Koonin EV, La Monica N, Tuler J, Bagdzhadzhyan A, Lai MM. The complete sequence (22 kilobases) of murine coronavirus gene 1 encoding the putative proteases and RNA polymerase[J]. Virology, 1991, 180(2): 567-582.
[DOI]
|
| [5] |
Schwarz B, Routledge E, Siddell SG. Murine coronavirus nonstructural protein ns2 is not essential for virus replication in transformed cells[J]. J Virol, 1990, 64(10): 4784-4791.
[DOI]
|
| [6] |
Weiss SR, Zoltick PW, Leibowitz JL. The ns4 gene of mouse hepatitis virus (MHV), strain A59 contains two ORFs and thus differs from ns4 of the JHM and S strains[J]. Arch Virol, 1993, 129(1/2/3/4): 301-309.
[PubMed]
|
| [7] |
Yokomori K, Lai MM. Mouse hepatitis virus S RNA sequence reveals that nonstructural proteins ns4 and ns5a are not essential for murine coronavirus replication[J]. J Virol, 1991, 65(10): 5605-5608.
[DOI]
|
| [8] |
Langereis MA, Van Vliet AL, Boot W, De Groot RJ. Attachment of mouse hepatitis virus to O-acetylated sialic acid is mediated by hemagglutinin-esterase and not by the spike protein[J]. J Virol, 2010, 84(17): 8970-8974.
[DOI]
|
| [9] |
Tsai JC, Zelus BD, Holmes KV, Weiss SR. The N-terminal domain of the murine coronavirus spike glycoprotein determines the CEACAM1 receptor specificity of the virus strain[J]. J Virol, 2003, 77(2): 841-850.
[DOI]
|
| [10] |
Weiss SR, Leibowitz JL. Pathogenesis of murine coronavirus infection[M]// Perlman S, Gallagher T, Snijder EJ. Nidoviruses. Washington, DC: ASM Press, 2007: 259-278.
|
| [11] |
Fischer F, Peng D, Hingley ST, Weiss SR, Masters PS. The internal open reading frame within the nucleocapsid gene of mouse hepatitis virus encodes a structural protein that is not essential for viral replication[J]. J Virol, 1997, 71(2): 996-1003.
[DOI]
|
| [12] |
Ma Z, Li Z, Dong L, Yang T, Xiao S. Reverse genetic systems: rational design of coronavirus live attenuated vaccines with immune sequelae[J]. Adv Virus Res, 2020, 107: 383-416.
[PubMed]
|
| [13] |
Masters PS. The molecular biology of coronaviruses[J]. Adv Virus Res, 2006, 66: 193-292.
|
| [14] |
Koetzner CA, Parker MM, Ricard CS, Sturman LS, Masters PS. Repair and mutagenesis of the genome of a deletion mutant of the coronavirus mouse hepatitis virus by targeted RNA recombination[J]. J Virol, 1992, 66(4): 1841-1848.
[DOI]
|
| [15] |
Masters PS, Koetzner CA, Kerr CA, Heo Y. Optimization of targeted RNA recombination and mapping of a novel nucleocapsid gene mutation in the coronavirus mouse hepatitis virus[J]. J Virol, 1994, 68(1): 328-337.
[DOI]
|
| [16] |
Kuo L, Godeke GJ, Raamsman MJ, Masters PS, Rottier PJ. Retargeting of coronavirus by substitution of the spike glycoprotein ectodomain: crossing the host cell species barrier[J]. J Virol, 2000, 74(3): 1393-1406.
[DOI]
|
| [17] |
Yount B, Denison MR, Weiss SR, Baric RS. Systematic assembly of a full-length infectious cDNA of mouse hepatitis virus strain A59[J]. J Virol, 2002, 76(21): 11065-11078.
[DOI]
|
| [18] |
Coley SE, Lavi E, Sawicki SG, Fu L, Schelle B, Karl N, Siddell SG, Thiel V. Recombinant mouse hepatitis virus strain A59 from cloned, full-length cDNA replicates to high titers in vitro and is fully pathogenic in vivo[J]. J Virol, 2005, 79(5): 3097-3106.
[DOI]
|
| [19] |
Thi Nhu Thao T, Labroussaa F, Ebert N, V'kovski P, Stalder H, Portmann J, Kelly J, Steiner S, Holwerda M, Kratzel A, Gultom M, Schmied K, Laloli L, Hüsser L, Wider M, Pfaender S, Hirt D, Cippà V, Crespo-Pomar S, Schröder S, Muth D, Niemeyer D, Corman VM, Müller MA, Drosten C, Dijkman R, Jores J, Thiel V. Rapid reconstruction of SARS-CoV-2 using a synthetic genomics platform[J]. Nature, 2020, 582(7813): 561-565.
[DOI]
|
| [20] |
Yao Q, Masters PS, Ye R. Negatively charged residues in the endodomain are critical for specific assembly of spike protein into murine coronavirus[J]. Virology, 2013, 442(1): 74-81.
[DOI]
|
| [21] |
Das Sarma J, Scheen E, Seo SH, Koval M, Weiss SR. Enhanced green fluorescent protein expression may be used to monitor murine coronavirus spread in vitro and in the mouse central nervous system[J]. J Neurovirol, 2002, 8(5): 381-391.
[DOI]
|
| [22] |
Zhu Y, Feng F, Hu G, Wang Y, Yu Y, Zhu Y, Xu W, Cai X, Sun Z, Han W, Ye R, Qu D, Ding Q, Huang X, Chen H, Xu W, Xie Y, Cai Q, Yuan Z, Zhang R. A genome-wide CRISPR screen identifies host factors that regulate SARS-CoV-2 entry[J]. Nat Commun, 2021, 12(1): 961.
[DOI]
|
| [23] |
Curtis KM, Yount B, Baric RS. Heterologous gene expression from transmissible gastroenteritis virus replicon particles[J]. J Virol, 2002, 76(3): 1422-1434.
[DOI]
|
| [24] |
Yount B, Curtis KM, Fritz EA, Hensley LE, Jahrling PB, Prentice E, Denison MR, Geisbert TW, Baric RS. Reverse genetics with a full-length infectious cDNA of severe acute respiratory syndrome coronavirus[J]. Proc Natl Acad Sci U S A, 2003, 100(22): 12995-13000.
[DOI]
|
| [25] |
Li Z, Wang X, Cao D, Sun R, Li C, Li G. Rapid review for the anti-coronavirus effect of remdesivir[J]. Drug Discov Ther, 2020, 14(2): 73-76.
[DOI]
|
| [26] |
Macphee PJ, Dindzans VJ, Fung LS, Levy GA. Acute and chronic changes in the microcirculation of the liver in inbred strains of mice following infection with mouse hepatitis virus type 3[J]. Hepatology, 1985, 5(4): 649-660.
[DOI]
|
| [27] |
Singh M, Khan RS, Dine K, Das Sarma J, Shindler KS. Intracranial inoculation is more potent than intranasal inoculation for inducing optic neuritis in the mouse hepatitis virus-induced model of multiple sclerosis[J]. Front Cell Infect Microbiol, 2018, 8: 311.
[DOI]
|
| [28] |
Thornbrough JM, Jha BK, Yount B, Goldstein SA, Li Y, Elliott R, Sims AC, Baric RS, Silverman RH, Weiss SR, Palese P, Perlman S, Hogue BG. Middle east respiratory syndrome coronavirus NS4b protein inhibits host RNase L activation[J]. mBio, 2016, 7(2): e00258-00216.
[DOI]
|
| [29] |
Körner RW, Majjouti M, Alcazar MAA, Mahabir E. Of mice and men: the coronavirus mhv and mouse models as a translational approach to understand SARS-CoV-2[J]. Viruses, 2020, 12(8): 880.
[DOI]
|
| [30] |
Nguyen VT, Morange M, Bensaude O. Firefly luciferase luminescence assays using scintillation counters for quantitation in transfected mammalian cells[J]. Anal Biochem, 1988, 171(2): 404-408.
[DOI]
|
| [31] |
de Haan CAM, van Genne L, Stoop JN, Volders H, Rottier PJM. Coronaviruses as vectors: position dependence of foreign gene expression[J]. J Virol, 2003, 77(21): 11312-11323.
[DOI]
|
| [32] |
Cervantes-Barragan L, Züst R, Maier R, Sierro S, Janda J, Levy F, Speiser D, Romero P, Rohrlich P-S, Ludewig B, Thiel V, Palese P. Dendritic cell-specific antigen delivery by coronavirus vaccine vectors induces long-lasting protective antiviral and antitumor immunity[J]. mBio, 2010, 1(4): e00171-10.
[DOI]
|
| [33] |
Thiel V, Herold J, Schelle B, Siddell SG. Viral replicase gene products suffice for coronavirus discontinuous transcription[J]. J Virol, 2001, 75(14): 6676-6681.
[DOI]
|
| [34] |
Thiel V, Karl N, Schelle B, Disterer P, Klagge I, Siddell SG. Multigene RNA vector based on coronavirus transcription[J]. J Virol, 2003, 77(18): 9790-9798.
[DOI]
|
 2022, Vol. 17
2022, Vol. 17


