2. 复旦大学上海医学院基础医学院,教育部/卫健委医学分子病毒学重点实验室,上海 200032;
3. 堪培拉大学生物医学系理学部,澳大利亚首都直辖区,2617
2. Key Laboratory of Medical Molecular Virology (MOE/NHC), School of Basic Medical Sciences, Shanghai Medical College, Fudan University, Shanghai 200032, China;
3. Faculty of Science and Technology, University of Canberra, Australia Capital Territory 2617, Australia
乙型肝炎病毒(hepatitis B virus, HBV)慢性感染是全球公共卫生问题。截至2019年,全球有超过2.96亿的HBV慢性感染者,而这些感染者最终有发展为终末期肝病的风险,如肝硬化、肝衰竭和原发性肝癌等[1]。HBV感染肝细胞后,在肝细胞核内形成稳定存在的cccDNA池,是HBV持续性感染的主要原因[2-3]。持续性沉默cccDNA是HBV功能性治愈的主要目标[4]。HBV在复制过程中整合到宿主基因组的HBV DNA也可以转录形成HBV mRNAs,严重干扰cccDNA来源RNA(episome-derived RNA, eRNA)的检测,尤其是经过长期抗病毒治疗或HBeAg阴性的患者,整合频率较高[5-6]。所以,对HBV转录本来源的特异区分是目前研究的难点和热点。
目前,关于HBV感染患者肝组织内HBV病毒学参数(HBV DNA, HBV RNA, cccDNA)的研究主要通过生化抽提再进行总体聚合酶链反应(polymerase chain reaction,PCR)定量的方式,获得的是大量细胞的平均水平,无法提供单细胞水平HBV病毒学参数的信息。本课题组前期在组织水平建立了检测HBV核酸的单细胞分辨率原位杂交技术(in situ hybridization, ISH),揭示治疗后HBV DNA或cccDNA阳性细胞的变化情况[7]。但ISH无法准确定量单细胞内的HBV核酸,灵敏度有待提高[8]。单细胞测序技术(single-cell sequencing)能够获得单个细胞内RNA或DNA的总体情况,灵敏度高于ISH。有研究结合单细胞测序技术和ISH影像研究揭示了单个细胞内cccDNA的转录模式[9]。但单细胞测序技术主要用于测序转录组,当研究特定基因的表达情况时,成本昂贵,总体效费比低。由此可见,单细胞研究肝组织内HBV靶标的技术须兼具高灵敏度和针对特定靶标的能力。
微滴数字PCR(droplet digital PCR, ddPCR)具有PCR扩增的偏好性,可以达到单拷贝灵敏度,结合单细胞分离技术可用于肝组织内HBV的单细胞研究,但此技术仍在发展阶段,尚不完善。有研究结合单细胞激光捕获显微切割(single-cell laser capture microdissection, scLCM)和ddPCR,发现长期抗病毒治疗后cccDNA的转录活性下降[10]。但scLCM的成本昂贵,操作烦琐,易损坏细胞,每个样本均需要大量的核酸抽提和ddPCR反应,不适合大样本量临床标本的检测。重要的是,须将单细胞裂解产物分成多等份,经相应核酸酶处理后才能检测不同核酸,可能无法检测到低拷贝核酸的信号。
鉴于上述方法的固有缺陷,本研究以优化肝组织内HBV病毒学活动的单细胞研究方法为目的,基于ddPCR开发了一套单细胞水平检测HBV DNA和RNA的单细胞微滴数字PCR方法(single-cell droplet digital PCR, sc-ddPCR),通过水相-油相乳化技术将单个细胞分散到含低凝胶度琼脂糖的微滴中,在琼脂糖冷却形成凝胶后,通过细胞裂解和蛋白酶消化保留总核酸,随后进行传统ddPCR检测:第2次油包水微滴生成、对多种靶标进行PCR扩增和荧光检测,获得单细胞水平特定核酸的定量数据。在肝癌细胞系中对该方法进行验证和优化,评测此方法检测HBV DNA的线性和检测下限。进一步设计针对eRNA的特异性引物和探针体系,验证此方法在检测RNA方面的线性和检测下限,以此建立一套相对简单、可针对多种特定靶标、高灵敏度和高通量的sc-ddPCR方法。
1 材料和方法 1.1 材料 1.1.1 细胞HepAD38细胞系由美国布隆格研究所郭巨涛教授惠赠,此细胞系整合有1.1拷贝的HBV基因组,CMV-tet启动子调控其复制开关。当多西环素(doxycycline)存在时,HBV复制关闭,抑制前基因组RNA(pregenomic RNA, pgRNA)的转录;当多西环素消失时,HBV复制开启,约2周之后,病毒复制能力稳定,处于高水平状态。人肝癌亚历山大细胞系(PLC/PRF/5)由本实验室保存,该细胞系模拟HBV自然整合状态,可以分泌乙肝病毒表面抗原(Hepatitis B surface protein, HBsAg)和乙型肝炎X蛋白(Hepatitis B X protein, HBx)。HepG2细胞系由本实验室保存,来自人肝癌组织。
1.1.2 仪器和试剂主要仪器包括MicroDrop-100A样本制备仪(广州Forevergen),微孔板封膜仪(广州Forevergen),PCR仪(X50S,德国Eppendorf),MicroDrop-20B生物芯片分析仪(广州Forevergen),显微镜(CIL,法国Nikon)。主要试剂包括DMEM培养基(美国Gibco),多聚赖氨酸(美国Sigma-Aldrich),胎牛血清(澳洲BI),胰蛋白酶(澳洲BI),谷氨酰胺(美国Invitrogen),青霉素和链霉素(澳洲BI),多西环素(上海Sangon Biotech),低凝胶度琼脂糖(A5030-5G,美国Sigma-Aldirich),乳化油(美国Ran biotechnologies),吐温-20(上海Sangon Biotech),蛋白酶K(美国Sigma-Aldrich),十二烷基硫酸钠SDS(深圳BBI),乙二胺四乙酸二钠盐二水EDTA(深圳BBI),三羟甲基氨基甲烷Tris(深圳BBI),盐酸HCL(上海国药公司),无水乙醇(上海国药公司),样本制备通用耗材(广州Forevergen),微滴式数字PCR用反应预混液(广州Forevergen),微滴检测油(广州Forevergen),一步法逆转录微滴式PCR用反应预混液(广州Forevergen),可拆分96孔PCR板(德国Eppendorf),96孔板可穿刺热封膜(德国Eppendorf),八联排管(德国Eppendorf)。
1.2 方法 1.2.1 引物和探针合成由上海生工生物科技公司合成。引物和探针序列如表 1所示。
| Name | Sequence(5’-3’) |
| HBV DNA-F | CGTCTGTGCCTTCTCRTCTG |
| HBV DNA-R | AGTCCAAGAGTYCTCTTATGYAAGACCTT |
| HBV DNA-P | FAM-CCGTGTGCACTTCGCTTCACCTCTGC-BHQ |
| amelegenin-F | CCCTGGGCTCTGTAAAGAA |
| amelegenin-R | ATCAGAGCTTAAACTGGGAAGCTG |
| amelegenin-P | VIC-CCCAGATGTTTCTCAAGTGGT-MGB |
| eRNA-F | GGAGGCTGTAGGCATAAATTGGT |
| eRNA-R | TTTTTTTTTTTTTTTTTTTTTMTAGCT |
| eRNA-P | FAM-ACTKTTCAAGCCTCCAAGCTGTGCCTT-BHQ |
HepG2细胞、HepAD38细胞和PLC细胞均在含有体积分数1%青霉素(10 000 U/mL)和链霉素(10 mg/mL)、体积分数1%谷氨酰胺和体积分数10%胎牛血清的DMEM培养基中培养,培养条件:37 ℃,体积分数5% CO2。如果要关闭HepAD38细胞的复制,按1∶1 000的比例加入多西环素(doxycycline),约2周后复制关闭。
1.2.3 单细胞琼脂糖微滴的制备裂解缓冲液制备:PBS,体积分数1%SDS,200 mmol/L EDTA,20 mmol/L Tris-HCL(pH值为8.0)。3%质量分数低凝胶度琼脂糖制备:将低凝胶度琼脂糖加入PBS中制成3%琼脂糖溶液,微波炉中加热至完全溶解,70 ℃水浴锅中静置>1 h,室温保存。实验前准备:将乳化油在40 ℃金属浴中预热1 h以上;将3%低凝胶度琼脂糖于微波炉中完全融化,70 ℃水浴锅中放置>0.5 h,然后移至40 ℃金属浴中。制备单细胞琼脂糖微滴:将1.5 mL离心管放入40 ℃金属浴中,加入75 μ L预热的5×106~107/mL的细胞悬液和等体积3%低凝胶度琼脂糖,吹打混匀。加入150 μ L乳化油,充分吹打混匀至上层相形成厚厚的乳白色层,40 ℃静置5 min。转移到冰上,静置1 h以上,琼脂糖冷却凝胶,细胞被稳定包裹在微滴中。使用100 μ mol/L细胞过滤筛过滤微滴,用15 mL预冷PBS冲洗细胞过滤筛,4 ℃,500 g,离心3 min,弃掉上清液,用预冷PBS重悬微滴至0.5 mL。细胞裂解和消化:加入0.5 mL裂解缓冲液和10 μ L蛋白酶K,37 ℃过夜12 h。洗涤:10 mL 2% Tween-20(PBS),轻柔吹打混匀,1 000 g离心3 min。10 mL无水乙醇,轻柔吹打混匀,1 000 g离心3 min。10 mL 0.02% Tween-20(PBS)洗涤5次,1 000 g离心3 min,尽可能弃掉上清液。
1.2.4 sc-dd PCR检测HBV DNA按照广东永诺医疗科技有限公司的微滴数字PCR检测系统的说明书进行操作。人amelogenin基因为内参基因,通过计算双阳性微滴比例,推算出HBV DNA单细胞阳性率。反应体系:微滴式数字PCR用反应预混液10 μ L,HBV DNA-F 0.9 μ mol/L,HBV DNA-R 0.9 μ mol/L,HBV DNA-P 0.25 μ mol/L,amelogenin-R 0.9 μ mol/L,amelogenin-F 0.9 μ mol/L,amelogenin-P 0.25 μ mol/L,单细胞琼脂糖微滴4.9 μ L,总体积20 μ L。ddPCR反应条件:95 ℃变性10 min,94 ℃ 30 s,60 ℃ 1 min,40个循环,98 ℃ 10 min,25 ℃ hold。
1.2.5 sc-dd PCR检测eRNA按照广东永诺医疗科技有限公司的微滴数字PCR检测系统的说明书进行操作。人amelogenin基因为内参基因,通过计算双阳性微滴比例,推出eRNA单细胞阳性率。反应体系:一步法逆转录微滴式数字PCR用反应预混液10 μ L,逆转录酶0.5 μ L,eRNA-F 0.9 μ mol/L,eRNA-R 0.9 μ mol/L,eRNA-P 0.25 μ mol/L,amelogenin-R 0.9 μ mol/L,amelogenin-F 0.9 μ mol/L,amelogenin-P 0.25 μ mol/L,单细胞琼脂糖微滴4.9 μ L,总体积20 μ L。ddPCR反应条件:46 ℃逆转录1 h,95 ℃变性10 min,94 ℃ 30 s,60 ℃ 1 min,40个循环,98 ℃ 10 min,25 ℃ hold。
1.2.6 统计学分析GraphPad Prism 8.0用于作图及数据统计分析,P < 0.05被认为具有统计学意义。将sc-ddPCR获得的双阳性微滴比例和实际HBV阳性细胞比例转换为对数值,通过线性回归分析两者的相关性,并计算直线的相关系数R2。通过非对称t检验,检测HBV阳性细胞比例和sc-ddPCR获得的双阳性微滴比例在统计学上的差异性。
2 结果 2.1 sc-ddPCR方法的建立本研究建立的sc-ddPCR方法包括两部分:单细胞琼脂糖微滴的制备和ddPCR检测。低凝胶温度的琼脂糖具有较低的凝胶点和熔点,能极好地进行凝胶内酶促反应和克隆分析,被广泛应用于琼脂糖微滴的制备[11-13]。本研究利用水相-油相乳化技术将待测细胞悬液分散到独立的低凝胶度琼脂糖微滴中,使细胞相互独立,互不干扰。通过低温处理使琼脂糖冷却凝胶,增加微滴包裹细胞的稳定性,经细胞裂解和蛋白酶K消化后,释放的核酸原位保留在微滴中,随后进行传统ddPCR的微滴生成、PCR扩增和荧光检测,具体流程如图 1所示。

|
| The detailed process of single-cell ddPCR (sc-ddPCR) can be found in the methods section. 图 1 sc-ddPCR流程 Fig. 1 Flow chart of sc-ddPCR |
为了验证单细胞琼脂糖微滴包裹细胞的效率,本研究选取了不同的肝癌细胞系制备单细胞琼脂糖微滴。通过显微镜观察发现,平均有43.26%的细胞被成功包裹于琼脂糖微滴中,其中包裹单细胞的微滴数和总细胞微滴数的比例为90.15%(见图 2A,表 2)。结果表明此方案可以成功捕获单细胞,生成的单细胞琼脂糖微滴数高,能够满足实验需要。
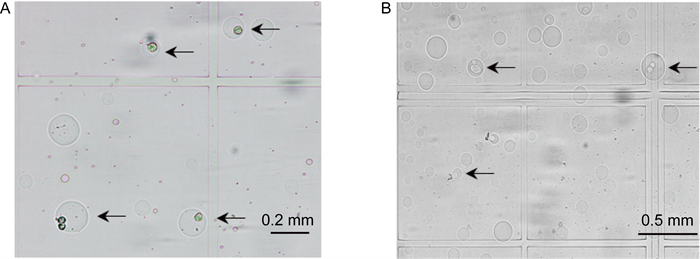
|
| A. Before lysis and digestion, the complete cell structure in the agarose droplets can be clearly seen. B. After lysis and digestion, the cell structure disappears. The arrow points to the droplets. The surrounding transparent layer is agarose, and the white opaque layer in the middle is the space left after the cell structure disappeared. 图 2 单细胞琼脂糖微滴裂解和消化前后的对比 Fig. 2 Comparison of single-cell agarose droplets before and after lysis and digestion |
| Cell | 1 | 2 | 3 | 4 | 5 | 6 |
| Input cell(/mL) | 6.19×106 | 6.19×106 | 6.19×106 | 6.19×106 | 6.19×106 | 6.17×106 |
| Cell droplet(/mL) | 6.5×105 | 4.67×105 | 4.0×105 | 4.0×105 | 2.44×105 | 2.5×105 |
| Single-cell droplet/cell droplet (%) | 90.92 | 100 | 100 | 70 | 100 | 80 |
| Encapsulation efficiency*(%) | 69.95 | 50.22 | 43.05 | 43.05 | 26.32 | 26.99 |
| Average(%) | 43.26 | |||||
| *The ratio of cell droplets and input cells. | ||||||
为了验证琼脂糖微滴中的细胞是否完全被裂解和消化,通过显微镜观察发现,在细胞裂解和消化后,包裹于微滴中的细胞结构消失(见图 2B),说明细胞已经被裂解和消化,能够成功释放核内待测核酸。
2.2 针对HBV DNA的sc-ddPCR方法的线性和灵敏度为了验证sc-ddPCR方法的线性和灵敏度,通过混合不同比例的HBV阳性[HepAD38 DOX(+) 细胞] 细胞和阴性(HepG2细胞)细胞模拟HBV慢性感染下的肝脏状态,使用HBV DNA引物和探针进行sc-ddPCR检测。HepAD38 DOX(+)细胞的比例依次为0%(HepG2细胞)、0.16%、0.8%、4.0%、20%和100%,其中0.16%、0.8%、4.0% HepAD38 DOX(+)细胞进行4次重复检测,其余组进行3次重复检测。通过线性回归分析,发现此方法的标准曲线展现出良好的线性,相关系数R2=0.998 7(见图 3B)。通过非对称t检验分析,计算出0.16% HBV阳性细胞测得的双阳性微滴比例和HepG2细胞的双阳性微滴比例的显著差异(P < 0.05),说明此方法的最低检测限度小于0.16%,有较好的灵敏度。
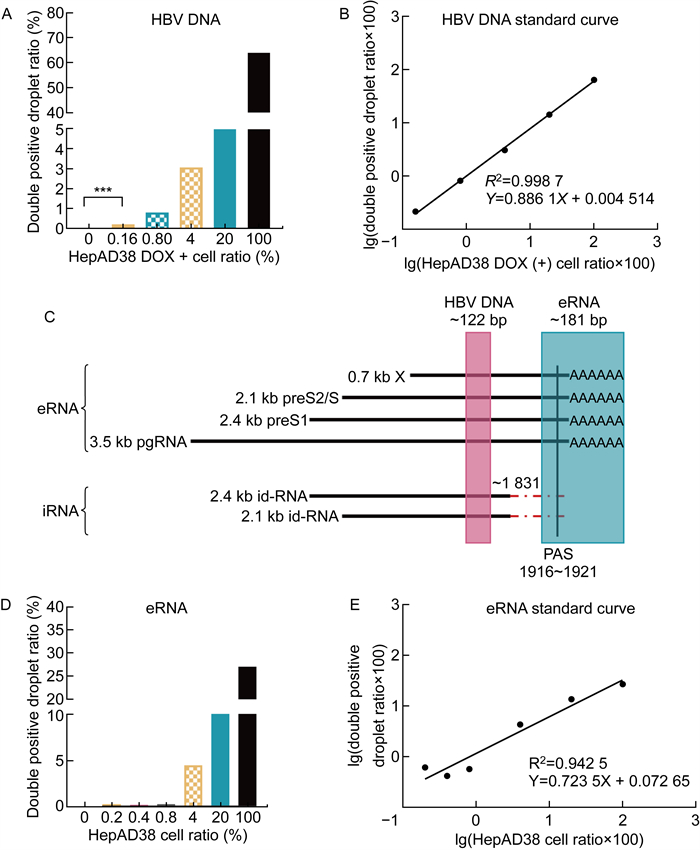
|
| A. The double positive droplet ratio in HepAD38 DOX(+) cells in different concentration gradients with HBV DNA primers and probe. The difference in double positive droplet ratios of HepG2 and 0.16% HepAD38 DOX(+)cells, the results showed significant differences (P < 0.05). B. The standard curve constructed by HBV DNA using sc-ddPCR technique. C. The differences in 3'ends of eRNAs and iRNAs. eRNAs have the same 3' end and are catalyzed by terminal PAS to form polyadenylate tails. The 3' ends of iRNAs could not be induced polyadenylated by PAS and are joined to the host genome. Horizontal lines represent eRNAs and iRNAs, dashed lines represent virus-host gene chimeric sequences, vertical lines represent PAS, and pink and blue boxes represent the amplified regions of HBV DNA and eRNA primers, respectively. PAS, polyadenylation signal. D. The double positive droplet ratio in HepAD38 cells in different concentration gradients with eRNA primers and probe. E. The standard curve constructed by eRNA using sc-ddPCR technique. 图 3 sc-ddPCR的标准曲线和灵敏度以及eRNA和iRNA的差异 Fig. 3 Standard curve and sensitivity of sc-ddPCR and differences between eRNA and iRNA |
HBV的总转录谱不仅包含cccDNA转录产生的mRNA(episome-derived RNA,eRNA),还包含来源于整合HBV DNA的转录产物(integrant-derived RNA, iRNA),针对eRNA设计特异性的引物探针体系具有较大的技术难度。cccDNA可以转录形成5种类型的mRNA(3.5 kb pregenomic RNA, 3.5 kb precore RNA, 2.4 kb preS1 RNA, 2.1 kb preS2/S RNA, 0.7kb X RNA),所有eRNA具有相同的3’末端,终止于多聚腺苷酸尾(polyadenylation, polyA)[14-15]。双链线性DNA(double-stranded linear DNA, dslDNA)是HBV整合的主要前体,由于其结构特点,导致整合HBV DNA产生病毒-宿主嵌合转录本,缺乏eRNA共有的3’末端(见图 3C)[16-17]。根据HBV转录本3’末端的结构差异,本研究设计了针对eRNA的特异性引物和探针,序列如表 1所示。
为了评估eRNA引物探针的特异度,使用sc-ddPCR方法检测PLC/PRF/5细胞(整合有部分HBV基因组,模拟HBV整合状态)、HepAD38细胞(HBV转录复制打开)、HepAD38 DOX(+)细胞(HBV转录复制受到抑制)和HepG2细胞的eRNA信号。结果如表 4所示,HepAD38细胞的双阳性微滴比例显著高于HepAD38 DOX(+)细胞,和PLC/PRF/5细胞获得的检测值之比为158(PLC/PRF/5细胞:0.24%,HepAD38细胞:38.12%,HepAD38 DOX(+)细胞:2.83%),说明eRNA引物探针可特异区分eRNA和iRNA,具有高特异度,可应用于本研究中。
| Single-cell isolation methods | Gradient dilution | scLCM | Droplet method |
| Spatial information | None | Yes | None |
| Operation | Simple | Complex | Simple |
| Cost | Low | High | High |
| Single-cell isolation | Error-prone | Error-prone, severe cell damage | High precision |
| Batch detect | Yes | No | Yes |
| Droplet | Water | HepG2 | HepAD38 DOX(+) | HepAD38 | PLC |
| FAM positive droplet | 0 | 0 | 143 | 790 | 14 |
| VIC positive droplet | 0 | 922 | 812 | 522 | 1 240 |
| FAM & VIC positive droplet | 0 | 0 | 23 | 199 | 3 |
| FAM & VIC positive droplet ratio (%) | 0.00 | 2.83 | 38.12 | 0.24 |
为了验证sc-ddPCR检测eRNA的线性和灵敏度,按不同比例混合HepAD38 DOX(-)细胞和HepG2细胞,HepAD38 DOX(-)细胞比例依次为0%(HepG2细胞)、0.2%、0.4%、0.8%、4.0%、20%和100%,使用eRNA引物和探针进行检测,其中0.2%、0.4%、0.8%、4.0% HepAD38 DOX(-)细胞进行4次重复检测,其余比例的细胞进行3次重复检测。通过线性回归分析,发现标准曲线具有良好的线性,相关系数R2=0.942 5(见图 3E)。当HepAD38 DOX(-)细胞的比例为0.2%时,4次重复实验有2次检测到双阳性微滴信号,说明此方法检测eRNA的最低检测下限为0.2%,具有较好的灵敏度。
3 讨论HBV是危害人类健康的重要病原体之一。在HBV感染患者的肝组织内,不同肝细胞间的HBV复制水平存在高度异质性,因此,研究总体水平的方法不能准确提供单细胞层面HBV核酸的信息。然而,目前肝组织内HBV的单细胞研究方法较少,仍处于开发阶段,需要进一步的优化和改善。
现有的研究肝组织内HBV相关参数的sc-ddPCR技术须结合单细胞分离技术,主要有梯度稀释法和scLCM技术。有文章将有限稀释的肝细胞核悬液分离至独立的微量管后进行核酸抽提和ddPCR检测,研究单细胞内cccDNA的拷贝数[18]。此方法的成本低,操作简单,但易分离细胞错误或丢失。也有研究团队通过结合scLCM和ddPCR检测肝组织内的cccDNA转录活性和整合来源转录本[10, 19-20]。此方法不仅提高了单细胞方面检测HBV靶标的准确度,还保留了空间信息,但操作烦琐,容易受主观意识影响和损坏细胞;须对单个样本做大量的核酸抽提和ddPCR反应,成本昂贵,耗费人力,不适用于大量临床肝标本的检测。
已有报道将反向引物包被的磁珠和细胞混合物制备成油包水微滴或琼脂糖微滴,使每个细胞分散至独立的微滴中,经PCR扩增后读取微滴或磁珠的荧光信号[11, 21]。上述方法以微滴作为捕获单细胞和反应的载体,成本低,捕获效率高,但微滴比较脆弱,不能承受复杂的实验处理,否则容易破坏微滴,导致微滴融合,降低实验的准确度,不适合需要经过DNA纯化才能释放核内核酸的细胞。相较于传统的微滴法,本研究利用琼脂糖微滴可转变为凝胶状态的特性,使琼脂糖微滴能够承受后续蛋白酶消化等处理,并稳定束缚细胞内的大分子,适用性更加广泛。在本研究中,一份样品获得的单细胞琼脂糖微滴可以用于多种靶标的检测,使样品得到充分的利用,有利于获得全面的HBV病毒学活动的相关信息。也有文献利用琼脂糖凝胶的特点,在琼脂糖微滴捕获单细胞后低温处理,经细胞裂解和消化后进行检测,但是操作方式和应用领域各不相同[12, 22-23]。结合本研究的结果来看,使用琼脂糖微滴捕获单个细胞的方案是可行的,这个方案提高了微滴的稳定性和分离细胞的准确度,具有良好的应用前景。
cccDNA的持续性沉默有利于HBV的功能性治愈,是目前的研究重点,但是HBV转录本也可以来源于整合HBV DNA,所以HBV转录本的特异性区分是十分重要的。目前鉴别HBV转录本来源的方法主要有Alu-PCR, RNA测序(RNA-sequencing)等,操作烦琐,成本高,具有较高的定量误差[24-25]。本研究建立的方法,操作简单,成本低,而且设计的eRNA引物探针体系具有高特异度,可特异区分eRNA和iRNA。
在肝标本应用方面,本课题组用该方法检测到了HBV DNA和eRNA信号。前期预实验结果显示,sc-ddPCR测得的HBV DNA阳性率和患者病毒载量呈正相关,说明该方法在肝标本应用方面是可行的。对于整合HBV DNA,可以通过设计特异性针对环状HBV DNA的引物和探针,间接估算线性HBV DNA的含量,由于线性HBV DNA主要为整合HBV DNA,所以可以用于估计HBV的整合频率[26]。但是,本课题组设计的环状HBV DNA引物和探针在检测PLC细胞系时,发现该引物的特异度还不能满足目前的要求。关于整合HBV DNA的研究,本课题组还在进一步开展中。
总之,本研究建立了一套简单易用的单细胞琼脂糖包裹-消化-ddPCR检测系统(sc-ddPCR),能够对肝癌细胞系统中的DNA和RNA进行检测,并将单细胞琼脂糖微滴结合ddPCR的方法首次应用于HBV病毒学参数的相关研究中,为进一步研究肝组织内HBV病毒学活动提供了创新性的技术平台,在HBV治疗策略和药物研发方面有良好的临床应用价值。
| [1] |
Lee WM. Hepatitis B virus infection[J]. N Engl J Med, 1997, 337(24): 1733-1745.
[DOI]
|
| [2] |
Nassal M. HBV cccDNA: viral persistence reservoir and key obstacle for a cure of chronic hepatitis B[J]. Gut, 2015, 1972-1984.
|
| [3] |
Ko C, Chakraborty A, Chou WM, Hasreiter J, Wettengel JM, Stadler D, Bester R, Asen T, Zhang K, Wisskirchen K, Mckeating JA, Ryu WS, Protzer U. Hepatitis B virus genome recycling and de novo secondary infection events maintain stable cccDNA levels[J]. J Hepatol, 2018, 69(6): 1231-1241.
[DOI]
|
| [4] |
Martinez MG, Boyd A, Combe E, Testoni B, Zoulim F. Covalently closed circular DNA: the ultimate therapeutic target for curing HBV infections[J]. J Hepatol, 2021, 75(3): 706-717.
[DOI]
|
| [5] |
Rydell GE, Larsson SB, Prakash K, Andersson M, Norder H, Hellstrand K, Norkrans G, Lindh M. Abundance of noncircular intrahepatic hepatitis B virus DNA may reflect frequent integration into human DNA in chronically infected patients[J]. J Infect Dis, 2022, 225(11): 1982-1990.
[DOI]
|
| [6] |
Meier MA, Calabrese D, Suslov A, Terracciano LM, Heim MH, Wieland S. Ubiquitous expression of HBsAg from integrated HBV DNA in patients with low viral load[J]. J Hepatol, 2021, 75(4): 840-847.
[DOI]
|
| [7] |
Zhang X, Lu W, Zheng Y, Wang W, Bai L, Chen L, Feng Y, Zhang Z, Yuan Z. In situ analysis of intrahepatic virological events in chronic hepatitis B virus infection[J]. J Clin Invest, 2016, 126(3): 1079-1092.
[DOI]
|
| [8] |
Bustamante-Jaramillo LF, Fingal J, Blondot ML, Rydell GE, Kann M. Imaging of hepatitis B virus nucleic acids: current advances and challenges[J]. Viruses, 2022, 14(3): 557.
[DOI]
|
| [9] |
Peng B, Jing Z, Zhou Z, Sun Y, Guo G, Tan Z, Diao Y, Yao Q, Ping Y, Li X, Ren T, Li B, Li W. Nonproductive hepatitis B virus covalently closed circular DNA generates HBx-related transcripts from the HBx/enhancer Ⅰ region and acquires reactivation by superinfection in single cells[J]. J Virol, 2023, 97(1): e0171722.
[DOI]
|
| [10] |
Balagopal A, Hwang HS, Grudda T, Quinn J, Sterling RK, Sulkowski MS, Thio CL. Single hepatocyte hepatitis B virus transcriptional landscape in HIV coinfection[J]. J Infect Dis, 2020, 221(9): 1462-1469.
[DOI]
|
| [11] |
Zhu Z, Zhang W, Leng X, Zhang M, Guan Z, Lu J, Yang CJ. Highly sensitive and quantitative detection of rare pathogens through agarose droplet microfluidic emulsion PCR at the single-cell level[J]. Lab Chip, 2012, 12(20): 3907-3913.
[DOI]
|
| [12] |
Geng T, Novak R, Mathies RA. Single-cell forensic short tandem repeat typing within microfluidic droplets[J]. Anal Chem, 2014, 86(1): 703-712.
[DOI]
|
| [13] |
Leng X, Zhang W, Wang C, Cui L, Yang CJ. Agarose droplet microfluidics for highly parallel and efficient single molecule emulsion PCR[J]. Lab Chip, 2010, 10(21): 2841-2843.
[DOI]
|
| [14] |
Seeger C, Mason WS. Molecular biology of hepatitis B virus infection[J]. Virology, 2015, 479/480: 672-686.
[DOI]
|
| [15] |
Stadelmayer B, Diederichs A, Chapus F, Rivoire M, Neveu G, Alam A, Fraisse L, Carter K, Testoni B, Zoulim F. Full-length 5'RACE identifies all major HBV transcripts in HBV-infected hepatocytes and patient serum[J]. J Hepatol, 2020, 73(1): 40-51.
[DOI]
|
| [16] |
Mason WS, Low HC, Xu C, Aldrich CE, Scougall CA, Grosse A, Clouston A, Chavez D, Litwin S, Peri S, Jilbert AR, Lanford RE. Detection of clonally expanded hepatocytes in chimpanzees with chronic hepatitis B virus infection[J]. J Virol, 2009, 83(17): 8396-8408.
[DOI]
|
| [17] |
Mason WS, Liu C, Aldrich CE, Litwin S, Yeh MM. Clonal expansion of normal-appearing human hepatocytes during chronic hepatitis B virus infection[J]. J Virol, 2010, 84(16): 8308-8315.
[DOI]
|
| [18] |
Zhang YY, Zhang BH, Theele D, Litwin S, Toll E, Summers J. Single-cell analysis of covalently closed circular DNA copy numbers in a hepadnavirus-infected liver[J]. Proc Natl Acad Sci U S A, 2003, 100(21): 12372-12377.
[DOI]
|
| [19] |
Grudda T, Hwang HS, Taddese M, Quinn J, Sulkowski MS, Sterling RK, Balagopal A, Thio CL. Integrated hepatitis B virus DNA maintains surface antigen production during antiviral treatment[J]. J Clin Invest, 2022, 132(18): e161818.
[DOI]
|
| [20] |
Balagopal A, Grudda T, Ribeiro RM, Saad YS, Hwang HS, Quinn J, Murphy M, Ward K, Sterling RK, Zhang Y, Perelson AS, Sulkowski MS, Osburn WO, Thio CL. Single hepatocytes show persistence and transcriptional inactivity of hepatitis B[J]. JCI Insight, 2020, 5(19): e140584.
[DOI]
|
| [21] |
Kumaresan P, Yang CJ, Cronier SA, Blazej RG, Mathies RA. High-throughput single copy DNA amplification and cell analysis in engineered nanoliter droplets[J]. Anal Chem, 2008, 80(10): 3522-3529.
[DOI]
|
| [22] |
Novak R, Zeng Y, Shuga J, Venugopalan G, Fletcher DA, Smith MT, Mathies RA. Single-cell multiplex gene detection and sequencing with microfluidically generated agarose emulsions[J]. Angew Chem Int Ed Engl, 2011, 50(2): 390-395.
[DOI]
|
| [23] |
Clark IC, Wheeler MA, Lee HG, Li Z, Sanmarco LM, Thaploo S, Polonio CM, Shin SW, Scalisi G, Henry AR, Rone JM, Giovannoni F, Charabati M, Akl CF, Aleman DM, Zandee SEJ, Prat A, Douek DC, Boritz EA, Quintana FJ, Abate AR. Identification of astrocyte regulators by nucleic acid cytometry[J]. Nature, 2023, 614(7947): 326-333.
[DOI]
|
| [24] |
Erken R, Loukachov V, Van Dort K, Van Den Hurk A, Takkenberg RB, De Niet A, Jansen L, Willemse S, Reesink H, Kootstra N. Quantified integrated hepatitis B virus is related to viral activity in patients with chronic hepatitis B[J]. Hepatology, 2022, 76(1): 196-206.
[DOI]
|
| [25] |
Van Buuren N, Ramirez R, Soulette C, Suri V, Han D, May L, Turner S, Parvangada PC, Martin R, Chan HLY, Marcellin P, Buti M, Bui N, Bhardwaj N, Gaggar A, Li L, Mo H, Feierbach B. Targeted long-read sequencing reveals clonally expanded HBV-associated chromosomal translocations in patients with chronic hepatitis B[J]. JHEP Rep, 2022, 4(4): 100449.
[DOI]
|
| [26] |
Rydell GE, Larsson SB, Prakash K, Andersson M, Norder H, Hellstrand K, Norkrans G, Lindh M. Abundance of non-circular intrahepatic hepatitis B virus DNA may reflect frequent integration into human DNA in chronically infected patients[J]. J Infect Dis, 2022, 225(11): 1982-1990.
[DOI]
|
 2023, Vol. 18
2023, Vol. 18


