侵袭性肺曲霉菌病(invasive pulmonary aspergillosis,IPA)是曲霉菌菌丝侵入肺实质引发的一种感染性疾病,是肺部曲霉菌感染中最常见的类型。我国侵袭性真菌感染负担极其严重,根据估算的数据,我国每年IPA的发病人数约为118万[1]。IPA不仅发生于免疫低下(如:血液系统恶性肿瘤、器官移植、恶性肿瘤化疗后、服用大剂量激素或免疫抑制剂)或合并基础疾病(如:慢性阻塞性肺疾病、支气管扩张)的人群,而且免疫正常人群因真菌过度暴露也可出现IPA[2-3]。侵袭性真菌感染的病死率高达30%~40%,提早诊断能够显著降低该病的死亡率[4]。
对于IPA而言,部分患者缺乏特异性征象,临床诊断较为困难,往往需要联合病原体培养、G/半乳甘露聚糖(glactomannan,GM)试验、分子生物学检测等多种手段,以提高该病的检出率[5]。涂片镜检操作简便,出结果快,但其敏感性极差。真菌培养可提供药物敏感性信息,但某些真菌类型生长缓慢,且阳性率偏低。GM试验,主要检测曲霉菌细胞壁特有的GM抗原,是严重免疫抑制/粒细胞缺乏患者诊断IPA的有效标志物。对于非粒细胞缺乏患者,血清GM实验的敏感性明显下降,且干扰因素较多[6]。
近年来,以宏基因组二代测序(metagenomic next-generation sequencing,mNGS)为代表的高通量测序技术,在感染性疾病诊断中的应用不断增多。相较于传统的真菌检测方法,mNGS无须培养,节省时间,且可以精确区分真菌类型,具有更高的敏感性和特异性[7]。然而,mNGS技术价格昂贵,易受人源背景或背景微生物的干扰,限制了其在临床的广泛使用。靶向下一代测序(targeted next-generation sequencing, tNGS)是通过针对目标病原微生物的基因序列设计特异性引物进行超多重PCR扩增,从而进行高通量测序以实现病原体鉴定的一种技术。重庆医科大学附属第二医院的一项研究收集了102名肺部感染患者的合格下呼吸道样本,同时进行mNGS和tNGS检测,纳入153种病原体的tNGS阳性检出率为82.17%,与mNGS的病原体检出率相当(86.51%),两者在病原体检出率方面相当,但tNGS更具有卫生经济学价值[8]。另一项国内的研究以培养法作为标准,tNGS检测肺泡灌洗液的敏感性和特异性分别为84.4%和97.7%,表明tNGS检出结果有较高的临床可信度[9]。此外,在47份肺泡灌洗液样本中,tNGS检出34份混合感染,其中多为细菌/病毒混合感染。然而,国内外尚未有研究系统性探讨tNGS技术在IPA诊断中的价值。
本研究以209例临床怀疑IPA的患者为研究对象,系统地评估tNGS技术在IPA诊断中的价值,包括与传统检测方法的比较、诊断混合感染的能力、曲霉菌种属的分型。此外,本文还回顾性搜集本检验中心15 583份肺泡灌洗液样本的数据,进一步分析tNGS技术对于曲霉菌及混合感染的鉴别能力。
1 资料与方法 1.1 一般资料本研究纳入2023年5月—2023年7月收治的疑似肺部真菌感染住院患者为研究对象,共209例。患者男性111例,女性98例;年龄6~89岁,平均45.72±16.80岁;炎症指标:白细胞升高的有165例,C反应蛋白升高的有151例,降钙素原升高的有115例;合并肺部基础疾病:支气管哮喘11例,慢性阻塞性肺病8例,肺结核5例;临床表现:咳嗽195例,发热113例,咳痰142例,呼吸困难65例。本研究经伦理委员会批准,伦理编号[2024154],所有患者或家属均签署外送样本知情同意书。
1.2 纳入与排除标准纳入标准:①有相关临床表现,接受胸部CT等检查,疑似肺部真菌感染的患者;②同意采用纤维支气管镜检查采集肺泡灌洗液,且同意接受tNGS检测的患者;③支气管肺泡灌洗液样品及检测过程均通过质量控制;④医疗资料记录完整的患者。以上条件须同时满足。
排除标准:①接受β内酰胺类抗生素、葡萄糖酸制剂、血液制品等治疗的患者;②临床资料不完整的患者。符合其中一条即排除。
1.3 试剂和仪器病原微生物DNA提取或纯化试剂盒、病原微生物RNA提取或纯化试剂盒、病原微生物核酸文库快速构建试剂盒购自广州金圻睿生物科技有限责任公司,PCR扩增仪购自美国Thermo Fisher公司,Qsep100全自动核酸蛋白分析仪购自Bioptic公司,Illumina MiniSeq Dx-CN测序仪购自美国Illumina公司。GM试剂盒购自天津丹娜生物,GM仪器全自动酶联免疫分析仪购自嘉兴科瑞迪医疗器械有限公司。革兰氏染色剂购自珠海贝索生物技术有限公司,光学显微镜为奥林巴斯生产的双目显微镜。
1.4 方法 1.4.1 tNGS检测将肺泡灌洗液样本彻底混匀,取650 μL原始样本进行液化、破壁,然后按照标准操作流程进行核酸提取。使用病原微生物核酸文库快速构建试剂盒构建文库。详细操作如下:①cDNA合成。样本处理:根据核酸浓度取样(浓度≥10 ng/μL取100 ng;浓度不足则取14 μL直接建库);引物退火:加入cDNA合成引物,65 ℃孵育2 min;合成反应:加入一链合成缓冲液和酶,运行程序(25 ℃ 5 min→37 ℃ 45 min→85℃ 5 s)。②目标区域富集(多重PCR)反应体系:向多重PCR预混液(M100)中加入9.5 μL cDNA产物;PCR程序:28个循环扩增(95 ℃ 30 s→60 ℃ 30 s→72 ℃ 30 s);产物纯化:磁珠纯化后取11.5 μL用于文库扩增。③文库扩增。扩增体系:向文库扩增预混液中加入纯化产物;PCR程序:10个循环扩增(参数同上);纯化与质控:磁珠纯化后测定文库浓度(要求≥0.5 ng/μL),检测片段大小(250~350 bp)。④文库Pooling:等质量混合文库(20~60 ng/样本),检测混合文库的浓度和片段分布。使用Qsep100全自动核酸蛋白分析仪质控文库,将制备好的文库加载到测序试剂中,使用Illumina MiniSeq Dx-CN测序仪进行测序。然后再与数据库中微生物核酸序列进行比对鉴定,并按照细菌、真菌、病毒、寄生虫、支原体\衣原体\螺旋体\立克次氏体进行分类,结合《临床微生物学手册(第十一版)》《高通量测序技术在分枝杆菌病诊断中的应用专家共识》《靶向高通量测序(tNGS)在感染性疾病中应用与实践专家共识》等进行致病微生物解读[10-12]。数据质量控制要求:Q30≥75%;原始序列数≥50 kb;内参序列数≥200。结果判定:对于曲霉菌、结核分枝杆菌复合群、非结核分枝杆菌、诺卡菌、隐球菌等,将特异性序列数>50条且覆盖≥50%扩增子靶点序列作为阳性阈值判定标准,当特异性序列数为10~50条,须重新复查tNGS,复查tNGS的特异性序列数>10条且非单一靶点序列,也判定为阳性;对于其他细菌、真菌、支原体或衣原体,将特异性序列数>50条且覆盖≥50%扩增子靶点序列作为阳性阈值判定标准;对于病毒类,将特异性序列>100条且覆盖≥50%扩增子靶点序列作为阳性阈值判定标准。
1.4.2 半乳甘露聚糖(GM)试验使用酶联免疫夹心法对tNGS检测提示曲霉菌阳性的灌洗液样本进行GM试验,并使用特异性抗体检测曲霉菌半乳甘露聚糖抗原。检测步骤具体如下。①检测时取出室温中放置至少30 min后的试剂盒板条,剩余板条于自封袋中密封并置于4 ℃冰箱中保存。②加样: 分别设置阴性和阳性质控品组、Cut-off质控品组、待测样本组、空白对照,各取100 μL加入酶标板中,在37 ℃下孵育60 min。③洗涤:揭开封板膜,洗涤酶标板。④加酶标抗体:于各组酶标板中分别加入100 μL酶标抗体,在37 ℃下孵育30 min。⑤洗涤:揭开封板膜,洗涤酶标板。⑥显色:洗涤结束后,每孔加入底物溶液100 μL,在37 ℃下孵育25~30 min,并避光保存。⑦终止:每孔加入终止液50 μL,5 min内在450 nm波长处读取吸光度值(参考波长620/630 nm)。
数据处理:指数(I)是待测样本OD值与Cut-off质控品OD值平均值的比值。指数(I)用于判定待测样本中是否含有半乳甘露聚糖抗原。结果判定:对于曲霉菌高危感染人群,若I<0.5,则判定为阴性,表示感染曲霉菌的风险较低;若I>0.5,则判定为阳性,表示感染曲霉菌的风险较高。2019年发布的第3版《EORTC/MSG指南》提出,采用肺部灌洗液进行GM试验来诊断侵袭性曲霉病的推荐阈值为I≥1.0,本文推荐肺部灌洗液进行GM试验的阳性阈值为I≥1.0[13]。
1.4.3 革兰氏染色涂片按照革兰氏染色涂片的操作规范实施操作,确认样本中是否存在曲霉菌属菌丝。
1.5 统计学方法将数据导入SPSS22.0,对资料进行分析处理,计量资料符合正态分布检验则采用均值±标准差(x±s)的形式来表示,采用t检验;计数资料采用(%)表示,采用卡方检验,P<0.05表示差异有统计学意义。
2 结果 2.1 tNGS和GM试验对疑似IPA患者的肺泡灌洗液样本中曲霉菌的检出情况比较对209例疑似IPA患者的肺泡灌洗液样本进行检测,tNGS技术共检出曲霉菌183例,阳性率为87.6%。以BALF样本GM试验≥1.0为标准,GM试验检出阳性样本147例,阳性率为70.3%。在敏感性方面,tNGS技术优于GM试验(P<0.001,见表 1)。无论tNGS技术还是GM试验,其对曲霉菌的检出率均远高于革兰氏染色涂片(42例,20.1%)。联合tNGS技术、GM试验和革兰氏染色涂片,可将曲霉菌的检出率提升至92.8%。
| tNGS | GM test | Χ2 | P值 | ||
| Positive | Negative | Total | |||
| Positive | 137 | 46 | 183 | 14.458 | <0.001 |
| Negative | 10 | 16 | 26 | ||
| Total | 147 | 62 | 209 | ||
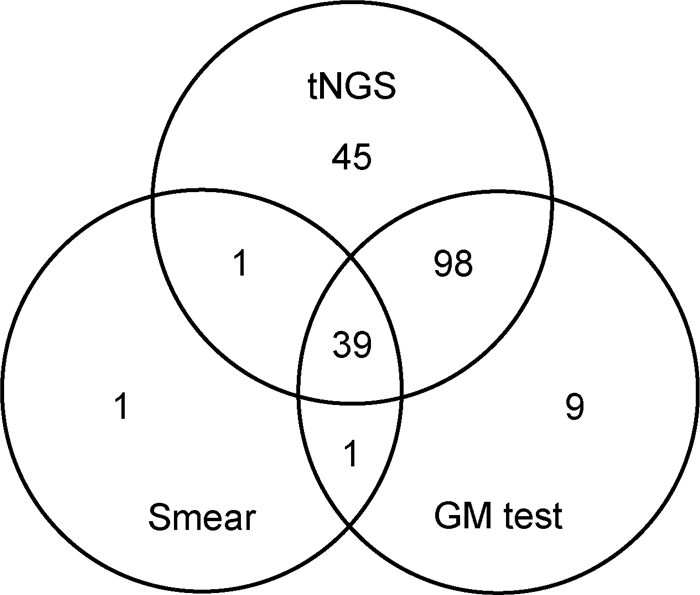
tNGS单独检出45例,GM试验单独检出9例,革兰氏染色涂片法单独检出1例,tNGS与GM试验同时检出98例,tNGS和革兰氏染色涂片同时检出1例,GM试验和革兰氏染色涂片同时检出1例,3种方法共同检出39例(见图 1)。在147例GM试验阳性的病例中,tNGS检出137例,阳性率为93.2%。
|
| 图 1 三种方法(tNGS、GM试验、革兰氏染色涂片法)对曲霉菌检出的一致性 Fig. 1 The consistency of three methods (tNGS, GM test, and Gram-stained smear method) in detecting Aspergillus |
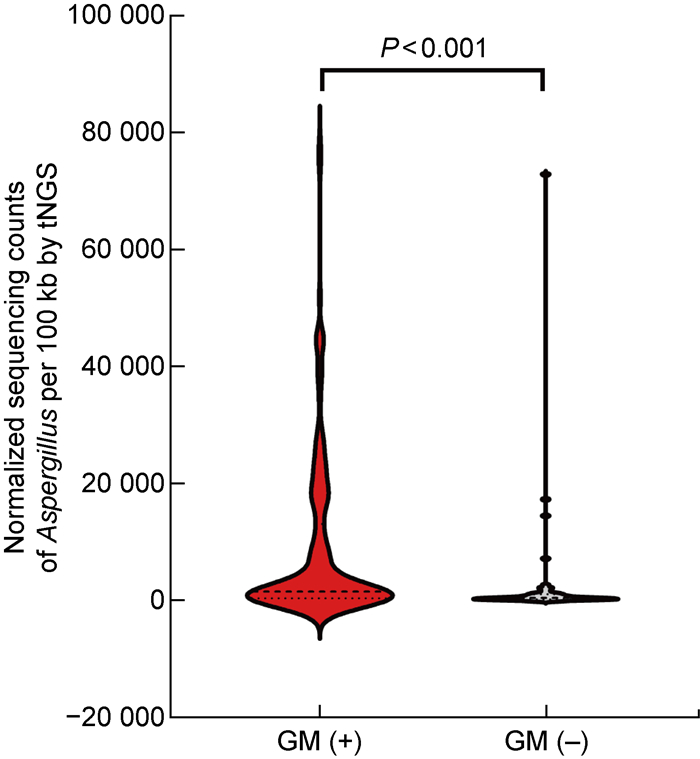
在183例tNGS检出曲霉菌阳性的患者中,GM试验阳性组曲霉菌均一化序列数的中位数为1 486,GM试验阴性组曲霉菌均一化序列数的中位数为427.5,GM试验阳性组曲霉菌均一化序列数显著高于GM试验阴性组(P<0.001,见图 2)。
|
| 图 2 183例tNGS检出曲霉菌的患者GM试验阳性组和阴性组曲霉菌的均一化序列数比较 Fig. 2 Comparison of the normalized sequence counts of Aspergillus between GM(+) and GM(-) group in 183 patients with Aspergillus detected by tNGS |
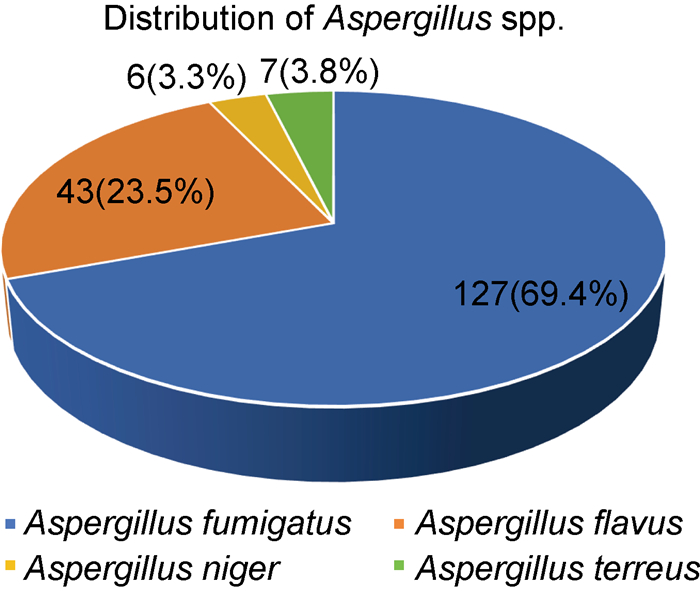
tNGS技术共检出曲霉菌183例,其中烟曲霉共检出127例,为检出率最高的曲霉菌种属,占比69.4%,其次为黄曲霉(43例,23.5%)、土曲霉(7例,3.8%)和黑曲霉(6例,3.3%)(见图 3)。
|
| 图 3 183例tNGS检出曲霉菌阳性的患者中曲霉菌种属构成 Fig. 3 Distribution of Aspergillus species in 183 patients with Aspergillus detected by tNGS |
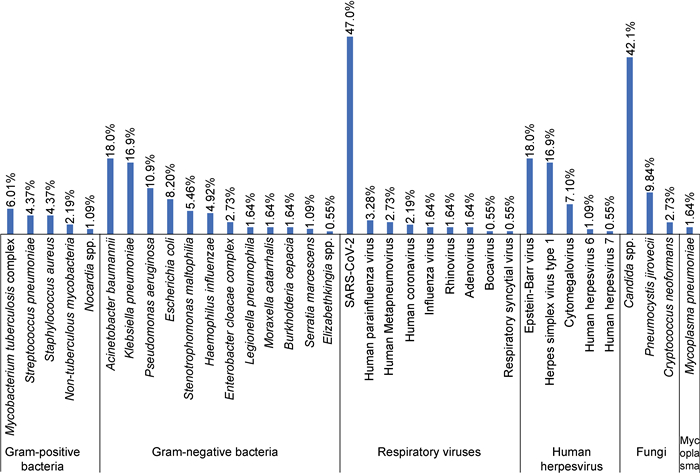
在183例tNGS检出曲霉菌阳性的BALF样本中,168例同时检出其他病原体,曲霉菌合并其他病原体检出占比91.8%(见图 4)。在革兰氏阳性菌中,与曲霉菌最常合并检出的是结核分枝杆菌复合群(6.01%)、肺炎链球菌(4.37%)、金黄色葡萄球菌(4.37%)。在革兰氏阴性菌中,与曲霉菌最常合并检出的是鲍曼不动杆菌(18.0%)、肺炎克雷伯菌(16.9%)、铜绿假单胞菌(10.9%)。在常见呼吸道病毒中,与曲霉菌最常合并检出的是新型冠状病毒(47.0%)。在疱疹病毒中,与曲霉菌最常合并检出的是EB病毒(18.0%)、单纯疱疹病毒1型(16.9%)和巨细胞病毒(7.1%)。在真菌中,最常合并检出的是念珠菌属(42.1%)和耶氏肺孢子菌(9.84%)。
|
| 图 4 183例曲霉菌阳性的肺泡灌洗液样本中其他病原体的合并检出率 Fig. 4 Co-detection rate of other pathogens in 183 case of Aspergillus-positive BALF samples |
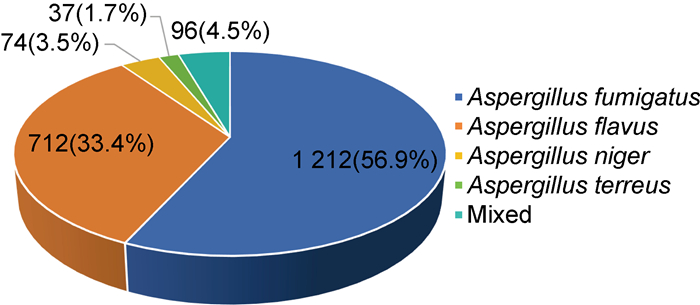
本文还统计了2023年本检验中心使用tNGS检测15 583份肺泡灌洗液样本的数据,共计2 131份样本检出曲霉菌,检出率为13.7%。其中,烟曲霉(56.9%)是最常检出的曲霉菌种属,其次为黄曲霉(33.4%)、黑曲霉(3.5%)和土曲霉(1.7%),2种以上曲霉种属混合检出占比为4.5%(见图 5)。
|
| 图 5 本检验中心2 131份肺泡灌洗液样本tNGS检出曲霉菌的种属构成比 Fig. 5 Distribution of Aspergillus species in 2 131 BALF samples detected by tNGS |
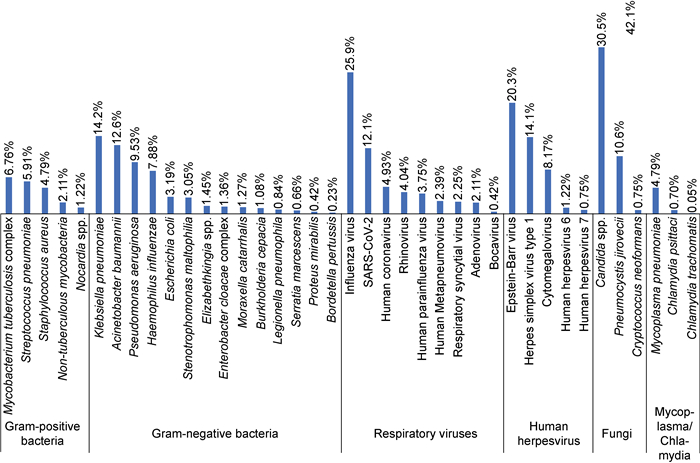
针对2 131份使用tNGS检出曲霉菌的样本,本文进一步评估了tNGS对于曲霉菌混合感染的检出能力。其中,1 942份检出其他病原体,曲霉菌合并其他病原体的检出占比为91.1%(见图 6)。在革兰氏阳性菌中,与曲霉菌最常合并检出的是结核分枝杆菌复合群(6.76%)、肺炎链球菌(5.91%)、金黄色葡萄球菌(4.79%)。在革兰氏阴性菌中,与曲霉菌最常合并检出的是肺炎克雷伯菌(14.2%)、鲍曼不动杆菌(12.6%)、铜绿假单胞菌(9.53%)。在常见呼吸道病毒中,与曲霉菌最常合并检出的是流感病毒(25.9%)和新型冠状病毒(12.1%)。在疱疹病毒中,与曲霉菌最常合并检出的是EB病毒(20.3%)、单纯疱疹病毒1型(14.1%)和巨细胞病毒(8.17%)。在真菌中,最常检出的是念珠菌属(30.5%)和耶氏肺孢子菌(10.6%)。
|
| 图 6 使用tNGS在2 131份曲霉菌阳性的肺泡灌洗液样本中检出的其他病原体 Fig. 6 Other pathogens detected in 2 131 Aspergillus-positive BALF samples using tNGS |
宏基因组二代测序技术具有广覆盖、无偏倚等特点,且无须进行微生物培养可直接对临床标本进行检测。越来越多的研究表明,mNGS在识别罕见的、新发的、难培养的微生物以及混合感染等方面具有显著优势。mNGS技术克服了传统微生物学检测的局限性,如出结果慢、灵敏度受抗菌药物影响大,以及在感染早期易出现假阴性血清学结果。侵袭性真菌感染在影像学上的表现往往不典型,且缺乏有效的诊断手段。近年来,已有多篇报道将mNGS技术用于侵袭性真菌感染的诊断。来自北京协和医院的一项回顾性研究表明,肺泡灌洗液mNGS诊断免疫缺陷患者IPA的敏感性、特异性和准确性分别为82.6%、97.7%和92.5%,其灵敏度优于任何单一的传统真菌检测方法,如革兰氏染色涂片法(8.7%)、真菌培养(39.1%)、血清GM试验(23.9%)和BALF GM试验(69.6%)[14]。mNGS能够准确区分曲霉属菌株,与真菌培养的一致性为77.8%。在39例临床诊断为IPA的非中性粒细胞减少患者中,使用BALF mNGS和传统检测方法均能检出病原体的病例占43.59%,48.72%的患者仅在BALF mNGS检测中呈阳性,5.13%的患者在BALF mNGS和传统检测方法中均为阴性,2.56%的患者仅在传统检测方法中呈阳性[15]。这些研究均表明mNGS技术不仅具有更高的敏感性,而且其能够准确区分曲霉菌种属,在诊断IPA方面优于真菌培养、GM试验等传统的检测方法。此外,mNGS技术在鉴别真菌混合感染方面具有独特的优势。Zhao等[16]利用mNGS技术,在119例确诊为肺部真菌感染的患者中发现40.3%的患者存在真菌与细菌混合感染。
与先前mNGS的研究结果相一致,本研究表明tNGS技术与传统的微生物检测方法(革兰氏涂片染色法、GM试验)相比,在诊断IPA方面也具有显著的优势,包括其具有更高的诊断敏感性、对曲霉菌的精准分型及混合感染的诊断能力。在本研究中,tNGS技术共检出曲霉菌183例,阳性率为87.6%,检出率优于GM试验(70.3%)。GM试验阳性组曲霉菌序列数远高于GM试验阴性组。在147例GM试验阳性的病例中,tNGS检出137例,阳性率为93.2%。采用tNGS技术对靶序列进行扩增,理论上提升了靶标病原体检测的灵敏度。以结核分枝杆菌复合群为例,在肺部感染的患者中,tNGS的检出率为9.30%,mNGS的检出率为4.76%,tNGS对结核分枝杆菌复合群的检出率较高[8]。一项来自美国约翰霍普金斯医院的研究,比较了mNGS和tNGS对201份肺泡灌洗液样本中病原体的检出性能,两者整体准确度相当(67.1% vs. 65.6%)[17]。在真菌感染诊断方面,tNGS技术的检测准确性也不弱于mNGS技术(92.6% vs. 90.2%)。不过该研究仅纳入了10余例真菌感染的患者,结果还有待大样本研究去证实。与mNGS技术相比,tNGS技术有效降低了检测成本,强化了耐药/毒力基因的检测性能,且不受人类基因组及背景菌的影响,通过引入内参实现了对病原体的定量检测。未来,这些优势可能使tNGS技术在IPA诊断中具有更广阔的应用前景。
免疫缺陷人群,如器官移植、中性粒细胞减少、艾滋病等患者,易发生各种机会性感染,也是IPA的高危人群。无论是IPA患者,还是免疫缺陷人群,均易出现混合感染。然而,传统的微生物检测方法局限于一种或若干种微生物,对混合感染的检出能力不足。根据本检验中心的数据,在15 583份肺泡灌洗液样本中,tNGS对曲霉菌的检出率为13.7%,其中烟曲霉(56.9%)是最常检出的曲霉菌种属,其次为黄曲霉(33.4%)、黑曲霉(3.5%)和土曲霉(1.7%),混合检出占比为4.5%,与先前基于真菌培养的报道基本一致[18]。这些研究表明了tNGS可以实现对曲霉菌的精准分型。此外,tNGS可鉴定出曲霉菌的混合感染。其中,流感病毒(25.9%)、肺炎克雷伯菌(14.2%)、鲍曼不动杆菌(12.6%)、新型冠状病毒(12.1%)、铜绿假单胞菌(9.53%)、结核分枝杆菌复合群(6.76%)是曲霉菌合并检出较高的病原体。
肺部曲霉菌感染多由外源性感染引起,呼吸道样本检出曲霉菌具有重要的临床价值,但其结果的解读仍须谨慎。Jiang等[19]的一项研究采集了376例怀疑肺部感染的患者的BALF样本并进行mNGS检测,其中68例检出曲霉菌阳性,36例确诊为曲霉菌感染,32例考虑为曲霉菌定植。在曲霉菌感染组,超过80%的患者均一化的序列数(每10 M数据量)>20条,而在曲霉菌定植组这一比例不到20%。烟曲霉和EB病毒的丰度在曲霉菌感染组显著增加,而定植组表现出更高的黑曲霉丰度。在另一项国内研究中[20],62例BALF mNGS检出曲霉菌的患者中,有41例临床诊断为曲霉菌感染。免疫缺陷患者的BALF mNGS结果与IPA诊断的符合率为82.1%,在免疫功能正常的患者中这一比例仅为52.9%。结合mNGS报告的解读经验,BALF样本tNGS检出曲霉菌时,感染、定植或污染均有可能,临床医生还须结合免疫状态、传统微生物检测结果(包括GM试验、培养、抗体检测)、临床表现及影像学特征等信息来综合判断。曲霉菌的细胞壁较厚,核酸释放效率低,可能导致mNGS检测出现假阴性结果。Qian等[21]的一项研究显示,约有40%的mNGS阴性结果最终被证实为假阴性,即其他样本类型的mNGS或其他方法检测结果为阳性。分枝杆菌、曲霉菌和支原体是mNGS最常漏检的3种病原体。因此,对于tNGS未检出曲霉菌的病例,若临床仍然怀疑,可以结合其他方法进行验证,多种方法联合,共同提高曲霉菌的检出率与诊断的准确性。
综上所述,tNGS技术在检测肺部真菌感染病原体方面展现出显著的优势。相较于传统的GM试验,tNGS在诊断IPA方面具有更高的敏感性,且能够鉴别曲霉菌的混合感染,有效提升IPA的诊断准确性。此外,tNGS技术还能精确识别曲霉菌的种类,为肺部真菌感染的病原学诊断提供了重要依据。tNGS技术的应用,不仅提高了诊断的效率和准确性,也为临床治疗提供了更为精确的指导。
| [1] |
Zhou LH, Jiang YK, Li RY, Huang LP, Yip CW, Denning DW, Zhu LP. Risk-based estimate of human fungal disease burden, China[J]. Emerg Infect Dis, 2020, 26(9): 2137-2147.
[DOI]
|
| [2] |
Chotirmall SH, Al-Alawi M, Mirkovic B, Lavelle G, Logan PM, Greene CM, McElvaney NG. Aspergillus-associated airway disease, inflammation, and the innate immune response[J]. Biomed Res Int, 2013, 2013: 723129.
[DOI]
|
| [3] |
Kanj A, Abdallah N, Soubani AO. The spectrum of pulmonary aspergillosis[J]. Respir Med, 2018, 141: 121-131.
[DOI]
|
| [4] |
Gong Y, Li C, Wang C, Li J, Ding M, Chen D, Lao M. Epidemiology and mortality-associated factors of invasive fungal disease in elderly patients: a 20-year retrospective study from Southern China[J]. Infect Drug Resist, 2020, 13: 711-723.
[DOI]
|
| [5] |
中国初级卫生保健基金会检验医学研究与转化专业委员会, 中国医院协会临床微生物实验室专业委员会, 全国真菌病监测网侵袭性霉菌感染监测项目组. 侵袭性霉菌感染实验室诊断临床应用专家共识[J]. 中华检验医学杂志, 2024, 47(6): 597-609. [DOI]
|
| [6] |
张传明, 曹泓宇, 孙继德, 牛司强, 王詝, 蒲姝丽, 徐绣宇. 血清和支气管肺泡灌洗液GM试验对非粒细胞缺乏患者不同类型肺曲霉病的诊断价值分析[J]. 中国真菌学杂志, 2022, 17(2): 146-151. [DOI]
|
| [7] |
Wang C, You Z, Fu J, Chen S, Bai D, Zhao H, Song P, Jia X, Yuan X, Xu W, Zhao Q, Pang F. Application of metagenomic next-generation sequencing in the diagnosis of pulmonary invasive fungal disease[J]. Front Cell Infect Microbiol, 2022, 12: 949505.
[DOI]
|
| [8] |
Li S, Tong J, Liu Y, Shen W, Hu P. Targeted next generation sequencing is comparable with metagenomic next generation sequencing in adults with pneumonia for pathogenic microorganism detection[J]. J Infect, 2022, 85(5): e127-e129.
[DOI]
|
| [9] |
Lin R, Xing Z, Liu X, Chai Q, Xin Z, Huang M, Zhu C, Luan C, Gao H, Du Y, Deng X, Zhang H, Ma D. Performance of targeted next-generation sequencing in the detection of respiratory pathogens and antimicrobial resistance genes for children[J]. J Med Microbiol, 2023, 72(11).
[DOI]
|
| [10] |
王辉, 马筱玲, 钱渊, 李若瑜, 曹建平, 史红. 临床微生物学手册[M]. 第11版. 北京: 中华医学电子音像出版社, 2017.
|
| [11] |
高通量测序共识专家组. 高通量测序技术在分枝杆菌病诊断中的应用专家共识[J]. 中华传染病杂志, 2023, 41(3): 175-182. [DOI]
|
| [12] |
中国医疗保健国际交流促进会临床微生物学分会. 靶向高通量测序在感染性疾病中应用与实践专家共识[J]. 中华医学杂志, 2024, 104(48): 4375-4383. [DOI]
|
| [13] |
Donnelly JP, Chen SC, Kauffman CA, Steinbach WJ, Baddley JW, Verweij PE, Clancy CJ, Wingard JR, Lockhart SR, Groll AH, Sorrell TC, Bassetti M, Akan H, Alexander BD, Andes D, Azoulay E, Bialek R, Bradsher RW, Bretagne S, Calandra T, Caliendo AM, Castagnola E, Cruciani M, Cuenca-Estrella M, Decker CF, Desai SR, Fisher B, Harrison T, Heussel CP, Jensen HE, Kibbler CC, Kontoyiannis DP, Kullberg BJ, Lagrou K, Lamoth F, Lehrnbecher T, Loeffler J, Lortholary O, Maertens J, Marchetti O, Marr KA, Masur H, Meis JF, Morrisey CO, Nucci M, Ostrosky-Zeichner L, Pagano L, Patterson TF, Perfect JR, Racil Z, Roilides E, Ruhnke M, Prokop CS, Shoham S, Slavin MA, Stevens DA, Thompson GR, Vazquez JA, Viscoli C, Walsh TJ, Warris A, Wheat LJ, White PL, Zaoutis TE, Pappas PG. Revision and update of the consensus definitions of invasive fungal disease from the European organization for research and treatment of cancer and the mycoses study group education and research consortium[J]. Clin Infect Dis, 2020, 71(6): 1367-1376.
[DOI]
|
| [14] |
Shi Y, Peng JM, Hu XY, Yang QW, Wang Y. Metagenomic next-generation sequencing for detecting Aspergillosis pneumonia in immunocompromised patients: a retrospective study[J]. Front Cell Infect Microbiol, 2023, 13: 1209724.
[DOI]
|
| [15] |
Zhu N, Zhou D, Xiong W, Zhang X, Li S. Performance of mNGS in bronchoalveolar lavage fluid for the diagnosis of invasive pulmonary aspergillosis in non-neutropenic patients[J]. Front Cell Infect Microbiol, 2023, 13: 1271853.
[DOI]
|
| [16] |
Zhao Z, Song J, Yang C, Yang L, Chen J, Li X, Wang Y, Feng J. Prevalence of fungal and bacterial co-infection in pulmonary fungal infections: a metagenomic next generation sequencing-based study[J]. Front Cell Infect Microbiol, 2021, 11: 749905.
[DOI]
|
| [17] |
Gaston DC, Miller HB, Fissel JA, Jacobs E, Gough E, Wu J, Klein EY, Carroll KC, Simner PJ. Evaluation of metagenomic and targeted next-generation sequencing workflows for detection of respiratory pathogens from bronchoalveolar lavage fluid specimens[J]. J Clin Microbiol, 2022, 60(7): e0052622.
[DOI]
|
| [18] |
Khan S, Bilal H, Shafiq M, Zhang D, Awais M, Chen C, Khan MN, Wang Q, Cai L, Islam R, Zeng Y. Distribution of Aspergillus species and risk factors for aspergillosis in mainland China: a systematic review[J]. Ther Adv Infect Dis, 2024, 11: 20499361241252537.
[DOI]
|
| [19] |
Jiang Z, Gai W, Zhang X, Zheng Y, Jin X, Han Z, Ao G, He J, Shu D, Liu X, Zhou Y, Hua Z. Clinical performance of metagenomic next-generation sequencing for diagnosis of pulmonary Aspergillus infection and colonization[J]. Front Cell Infect Microbiol, 2024, 14: 1345706.
[DOI]
|
| [20] |
Zhan W, Liu Q, Yang C, Zhao Z, Yang L, Wang Y, Feng J. Evaluation of metagenomic next-generation sequencing diagnosis for invasive pulmonary aspergillosis in immunocompromised and immunocompetent patients[J]. Mycoses, 2023, 66(4): 331-337.
[DOI]
|
| [21] |
Qian M, Zhu B, Zhan Y, Wang L, Shen Q, Zhang M, Yue L, Wu D, Chen H, Wang X, Cheng Y. Analysis of negative results of metagenomics next-generation sequencing in clinical practice[J]. Front Cell Infect Microbiol, 2022, 12: 892076.
[DOI]
|
 2025, Vol. 20
2025, Vol. 20


