2. 复旦大学上海医学院基础医学院教育部、卫健委、医科院医学分子病毒学重点实验室,上海 200032;
3. 教育部感染性疾病分子生物学重点实验室,重庆医科大学病毒性肝炎研究所,重庆医科大学附属第二医院感染科,重庆 400010
2. Key Laboratory of Medical Molecular Virology (MOE/NHC/CAMS), School of Basic Medical Sciences, Shanghai Medical College Fudan University, Shanghai 200032, China;
3. Key Laboratory of Molecular Biology for Infectious Diseases (Ministry of Education), Institute for Viral Hepatitis, Department of Infectious Diseases, The Second Affiliated Hospital, Chongqing Medical University, Chongqing 400010, China
准确、灵敏且具有重复性的核酸检测方法对准确获知并描述靶标核酸分子在生物学过程中的作用和动力学具有重要意义。自诞生以来,聚合酶链反应(polymerase chain reaction,PCR)及其衍生技术在推动现代分子生物学乃至整个生命科学的研究和应用发展中起到了基础性作用。特别是实时定量PCR(quantitative PCR,qPCR)技术,已成为核酸检测的金标准,广泛应用于科研和临床诊断各领域。
然而,qPCR应用的深入和拓展受其固有方法学局限的制约。作为核酸间接定量手段,qPCR难以稳定、可靠地检测高丰度同源序列背景下的极低丰度靶标序列[1],难以识别微小靶标核酸浓度变化[2],无法脱离标准曲线等外部参照进行定量[3],难以保证数据的精密度,特别是在靶标核酸含量较低时[4]。
1992年,Morley等提出了不同于qPCR的“有限稀释PCR”(limiting dilution PCR),用于定量检测IgH基因重排,可在160 000拷贝野生型背景中检测出2拷贝的突变序列[5]。随后,他们于The Lancet报道了使用有限稀释PCR高灵敏度定量检测白血病细胞,并监测治疗反应和分析急性白血病的预后[6]。1999年,Vogelstein和Kinzler[7]第1次使用“数字PCR”(digital PCR,dPCR)来命名基于有限稀释PCR的方法,并成功在结直肠癌患者粪便DNA样本中对KRAS突变进行了定量分析。
1 dPCR的原理和特性 1.1 dPCR的基本原理现代dPCR依然遵从“有限稀释PCR”的基本原理,通过特定的技术手段,将原本是一个整体的荧光PCR反应体系(例如20 μL)随机“分装/分配”(partitioning)至大量(通常是数万个)等体积微小独立PCR反应单元中(纳升级甚至更小的体积),使一部分微反应单元中不含有靶标核酸分子,而其他微反应单元中含有1拷贝至多个拷贝的靶标核酸分子[4, 8-9]。在随后的PCR步骤中,各微反应单元扩增至终点,未含靶标序列的微反应单元保持背景荧光强度,含有靶标序列的微反应单元的荧光信号显著提升,从而可将各微反应单元划分为阴性或阳性反应(图 1)。
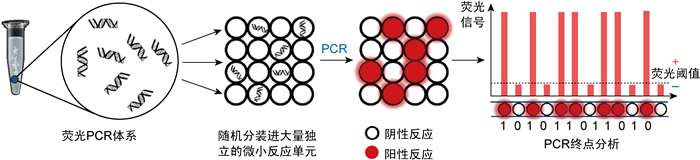
|
| 图 1 数字PCR的原理和工作流程 Fig. 1 The principle and workflow of digital PCR |
泊松分布概率函数的引入拓展了dPCR的动态范围。阳性微反应单元中可能含有多于1拷贝的靶标分子(特别是在样本浓度较高时),故阳性微反应单元的数量并不能直接代表靶标分子的数量,且阳性微反应单元或阴性微反应单元出现的概率遵从泊松分布概率函数[4, 10]。因此,只要获得阳性微反应单元或阴性微反应单元在总反应单元中的比例,即可根据泊松分布函数计算出样本的拷贝数浓度(拷贝/μL)。计算方式如下:
| $ C=\frac{-\ln (1-P)}{V} \times \frac{A}{B} $ |
其中C为靶标分子的拷贝数浓度;P为阳性微反应单元的比例;V为微反应单元的反应体积;A为总反应体积;B为样本载入体积。注意这个过程中没有使用任何外部参照,如标准曲线。dPCR将荧光PCR扩增过程中与PCR产物积累相关的呈指数增长的荧光模拟信号转变为线性数字化信号,基于统计学分析直接获取靶标分子的初始含量。
根据随机分装的方式及微反应单元性质的不同,目前dPCR可分为芯片式数字PCR(chip-based digital PCR,cdPCR)和微滴式数字PCR(droplet digital PCR,ddPCR)两大类。前者是在硅芯片上通过蚀刻技术制作出数万个彼此独立的反应孔组成的矩阵;后者是通过数万至千万个均匀的油包水(water in oil,W/O)微液滴作为微反应单元。由于技术手段不同,两种数字PCR系统各有特点。一般来说,ddPCR设计较为灵活,作为微反应单元的油包水微滴数的增加容易实现,也更易实现高通量和自动化,运行成本更低。由于微滴的可操控性,ddPCR的应用范围可进一步深入拓展。
1.2 dPCR的特性与优势dPCR作为一种“分子计数”的方法,有着独特的方法学特性。首先,dPCR的线性范围与微反应单元的数量有关,反应单元数越多,线性范围越宽。例如,2万个微液滴的ddPCR平台可覆盖5个数量级的线性范围,阳性微滴的比例在0.16%~99.6%的区间[9],对应1~100 000拷贝范围。样本浓度超过dPCR的线性动态范围会导致微反应单元呈饱和阳性状态,无法进行定量。其次,dPCR检测数据的精密度在平均每个微反应单元含有约1.59拷贝时最高,与微反应单元数量无关。此外,微反应单元体积的均一性对确保dPCR定量的准确性来说非常重要[10]。最后,基于泊松分布函数的特性,从单个反应的dPCR结果即可估计出泊松误差范围。
目前qPCR依然是包括感染性疾病在内的疾病相关核酸标记检测的金标准,但其dPCR与qPCR相比在诸多方面有明显优势(表 1)。
| qPCR | dPCR | |
| 定量方式 | 相对定量 | 绝对定量 |
| 标准曲线 | 依赖 | 无须 |
| 检测方式 | 实时荧光信号检测,有Cq值 | PCR终点荧光信号检测,无Cq值 |
| 受PCR扩增效率变化的影响 | 大 | 小 |
| 动态范围 | 9~10个数量级 | 5~6个数量级 |
| 灵敏度 | ** | *** |
| 精确度 | ** | *** |
| 运行成本 | * | ** |
与qPCR依赖已知标准品构建的标准曲线来实现定量分析不同,dPCR的绝对定量并不依赖任何外部参照,也不会受标准品的来源、储运、降解等影响而降低准确性;dPCR提供的拷贝数浓度相比质量浓度更能反映样本中完整的可扩增序列的有效含量。此外,定量检测结果能为样本阴阳性判断提供量化而客观的、唯一固定的阈值。检验人员可更加严谨地在给定的置信度内,根据量化的指标而无须依赖qPCR上无法唯一确定的Cq值来判断靶标分子的有无和确定样本的状态。
1.2.2 受PCR扩增效率变化的影响较小由于采用PCR终点荧光分析的方式,dPCR并不关注扩增过程中的荧光信号增长速度,仅需测量各微反应单元在PCR终点时的荧光信号,进而分辨阳性和阴性反应并统计其数量。PCR扩增效率的降低可能会影响各阳性微反应单元在PCR终点的荧光强度,表现为荧光信号强度降低,但只要还能清晰地区分阳性反应单元簇与阴性反应单元簇,即确定靶标分子存在(1)或无(0)的状态,仍可维持准确定量。因此,dPCR受PCR扩增效率变化的影响较qPCR小。
1.2.3 更高的灵敏度dPCR的高灵敏度尤其体现在高丰度野生型序列背景下对低丰度单碱基突变的检测能力上。使用与qPCR相同的引物/探针时,dPCR能将对低丰度突变的检测灵敏度提高几个数量级。样本的随机分装使得靶标核酸和干扰检测的背景序列都随机进入微反应单元,在含有靶标分子的反应单元中减少了背景序列的丰度,相对提高了靶标分子的含量,使靶标分子的扩增信号能更容易地被识别,进而提高检出率。例如,在肿瘤液体活检中,ddPCR对各种酪氨酸激酶抑制剂(tyrosine kinase inhibitor,TKI)敏感或耐药的突变位点的检测限可达到 < 0.01%的水平[1]。在感染性疾病方面,ddPCR对病毒耐药突变的检测限能达到0.1%,比临床常规使用的qPCR提高50倍以上[11]。
此外,对于低浓度靶标序列的定量,qPCR通常会由于扩增不明显或Cq值偏大(如>35)而不能有效检测;但对于dPCR,靶标序列在微小反应单元中的扩增会使该反应单元荧光信号陡增,明显区别于未扩增的单元,能更容易地被检测器识别出来。Persaud等报道,ddPCR能从106个人体免疫细胞中检出 < 3拷贝的人类免疫缺陷病毒(human immunodeficiency virus,HIV)[12]。
1.2.4 更高的精密度得益于“分子计数”的直接定量方式和终点分析,dPCR定量数据的精密度有很大提升,特别是对低浓度样本的检测,dPCR往往有更小的标准偏差(standard derivation,SD)和变异系数(coefficient of variation,CV)。有报道称,对于微小RNA(micro RNA,miRNA)的定量检测,相对于qPCR,ddPCR能将CV降低37%~86%[4]。理论上,dPCR要实现在95%置信度下区分1.1倍的拷贝数变异(copy number variation,CNV)差异或基因表达差异,仅需8 000个左右的微反应单元[2],比qPCR更容易分辨靶标分子的微小浓度变化,可用于CNV分析[2, 13]、等位基因差异表达[14]及单细胞分析。
1.2.5 更好的重复性和再现性凭借不依赖外部参照可独立获得定量结果及更强的抗干扰能力,dPCR的检查结果可在不同实验室间、不同批次间得到很好的重复和再现。2017年的一项针对ddPCR重复性和可操作性的室间评比研究表明,ddPCR在21个独立的国际实验室之间针对低丰度(~0.17%)的KRAS G12D突变具有高度的检测重复性[15]。
2 dPCR的发展历程尽管技术原理简单,但早期实现dPCR并不容易。在早期应用中,Vogelstein和Kinzler等不得不使用384孔PCR反应板,分析一个样本需使用超过2 mL的反应试剂[7];且移液加样操作烦琐,实验效率低下。Perkel曾表示,通常很难说服一名研究生使用dPCR开展实验[16]。2006年,Fluidigm公司推出了第1台商业化的dPCR设备,这是基于集成流体回路(芯片)的dPCR系统,通过集成的腔室和阀门系统来实现样本的分隔。2010年,Life Technologies公司收购BioTrove公司,并推出了基于OpenArray系统的dPCR平台。2011年,Bio-Rad公司收购QuantaLife公司,推出了成熟的ddPCR系统,并获得广泛应用,发表了大量应用文献和数据。
3 ddPCR在感染性疾病中的应用作为新一代核酸定量分析工具,dPCR的应用涉及生命科学各方面,特别是在医学基础研究及临床检验领域中的应用中不断深入和拓展。在感染性疾病方面,最近几年持续增长的文献报道指出,ddPCR可用于多种病原微生物的临床检验和相关研究。基于其技术特点,ddPCR不仅能对病原微生物的数量进行定量检测,还能检测分析病原微生物耐药突变、基因组多态性及用于单细胞捕获分析等。
3.1 病原微生物的绝对定量 3.1.1 标准参考物质的标定ddPCR不依赖外部参照即可精确定量的能力可用于标准参考物(standard reference material,SRM)的标定。目前,dPCR潜在的计量学价值已在核酸SRM标定方面获得应用[17-18]。美国国家标准与技术研究院(National Institute of Standards and Technology,NIST)推出使用dPCR标定的巨细胞病毒(cytomegalovirus,CMV)SRM 2366[19]。这样的SRM有助于建立qPCR方法质量控制、实验室间的量值溯源、方法学验证及不同检测方法的比较。
3.1.2 病毒载量的监测病毒载量的动态变化具有显著的临床意义。ddPCR高灵敏度和高数据精密度的特性,有助于识别病毒载量的动态变化,加深对一系列感染性疾病病原体的动态和治疗反应的理解,如HIV感染、病毒性肝炎及结核病,进而实现对疾病进展和疗效的监控。Strain等分别使用ddPCR和qPCR对150例HIV感染者样本的总HIV DNA(pol基因拷贝/百万细胞)和染色体外2-LTR circle DNA进行了分析,相比qPCR,ddPCR对这两种靶标检测结果的CV分别下降了5倍和20倍[20]。Taylor等[21]对1例流感病毒H1N1感染者在接受奥司他韦和扎那米韦(zanamivir)治疗期间的鼻咽分泌物(nasopharyngeal aspirate,NPA)样本分别用ddPCR和qPCR进行检测分析,发现qPCR数据的CV范围为45%~109.7%,而ddPCR仅为0.9%~11.4%。更重要的是,qPCR的定量结果没有显示出药物治疗与病毒载量的相关性,各检测点之间差异无统计学意义(P>0.05),而ddPCR定量结果反映药物治疗过程中病毒载量显著下降(P < 0.05)。
3.1.3 克服抑制物的影响dPCR受PCR扩增效率变化的影响较小,在应用中对PCR抑制物的耐受程度更高,能在一定程度上克服临床样本中常见抑制物的不利影响[22],从而有助于提高数据准确性和阳性检出率。有报道证明,ddPCR可从各种复杂样本中稳定检测靶标核酸分子。Sedlak等使用多重ddPCR检测粪便样本中的CMV[23],比qPCR有更好的表现。Cao等[24]使用双重ddPCR定量检测肠球菌属和人类粪便相关的HF183标记以评估水质,通过对标注物质和131个粪便和水质样品的分析,认为ddPCR对抑制物的耐受程度比qPCR高1~2个数量级,具有更好的精确度和可重复性。Pavšič等[25]使用ddPCR和qPCR对未经过DNA提取纯化的细胞裂解液中的CMV进行检测,并与经过提取的样本结果进行比较,发现直接定量的结果具有重复性,考虑到核酸提取的效率及损耗,这可能与样本中的实际病毒含量更接近。
此外,dPCR适用于准确定量序列高度变异的靶标。如针对同一种病毒,不同的引物/探针靶向不同的基因序列。在qPCR平台上,不同引物/探针的扩增效率差异及靶标序列本身变异会导致定量结果有很大的差异[26]。而dPCR平台不易受引物/探针结合位点的序列变化,能减少对定量结果的影响,提高检测结果的一致性和可比性。
3.2 耐药突变检测ddPCR可早期检测痕量的病原微生物耐药突变,不仅可减少无效药物治疗,还可在早期提示更换其他有效药物,及时调整治疗方案,进而实现感染性疾病的“精准医疗”。这可能对感染性疾病的临床研究和感染病患者的全程管理产生重要影响。Whale等[11]使用ddPCR检测对奥司他韦耐药的甲型流感病毒,其检测限低至0.1%突变丰度,比临床常用的qPCR高50倍。Taylor等[21]使用2009年新型甲型H1N1流感病毒野生株和对奥司他韦耐药的H275Y点突变(神经氨酸酶)H1N1毒株提取的RNA,比较qPCR与ddPCR在高丰度野生型H1N1背景下检出H275Y的能力,结果发现ddPCR显著提高了对突变病毒的检测灵敏度(>30倍)。Mukaide等[27]对多态性病毒基因组中突变进行定量检测时,建立了对丙型肝炎病毒(hepatitis C virus,HCV)核心氨基酸第70位点突变的ddPCR检测方法,LoD达0.005%,超过qPCR方法200倍。
3.3 基因编辑事件检测在慢性病毒病治疗中,根除或控制病毒潜伏库对患者停止抗反转录病毒治疗并实现治愈至关重要。基于转录激活因子样效应核酸酶(transcription activator-like effector nuclease,TALEN),特别是成簇的规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeat,CRISPR)/CRISPR相关蛋白9(CRISPR-associated protein 9,Cas9)基因编辑技术删除病毒基因[28-29],条件性敲除病毒感染的宿主辅助受体(如CCR5[30]、内源T细胞受体[31-32]),是颇有前途的病毒病基因治疗方法。ddPCR可应用于基于基因编辑的基因治疗方法的开发,能分析病毒颗粒水平[33],还有助于基因编辑技术的优化及质量控制。ddPCR可有效地从高丰度野生型细胞中定量检出特异性DNA双链断裂(double strand break,DSB)介导的同源重组修复(homology-directed repair,HDR),通过独特的“Drop-off”引物探针设计策略,定量非同源末端连接(non-homologous end-joining,NHEJ)事件[34-36],从而加快筛选富集低丰度的基因编辑细胞[37],评估向导RNA(guide RNA,gRNA)效率,优化HDR:HNEJ比例,还能评估基因编辑的脱靶率[33]。
3.4 基因连锁分析产志贺毒素型大肠埃希菌(shiga toxin-producing Escherichia coli,STEC)可能引起人类消化道疾病,其中肠出血性大肠埃希菌(enterohemorrhagic Escherichia coli,EHEC)感染严重时可致命。EHEC与其他可共生的大肠埃希菌或低毒的STEC很难通过生化特征区分,但EHEC同时含有志贺毒力基因stx1或stx2及编码紧密黏附素的基因eae。经典的选择培养PCR检测法会导致50%的假阳性。McMahon等[38]利用ddPCR的纳升级微滴直接包裹完整的细菌细胞,分析stx和eae基因同时出现的连锁性(linkage):如果stx1/stx2基因和eae基因存在于同一个细胞即EHEC细胞,则微滴呈双阳性;如果stx1/stx2和eae基因分别处于不同的细胞,则微滴呈单阳性。双阳性微滴的数量只与样本细菌密度有关。通过ddPCR的QuantaSoft软件自动计算连锁率,可稳定可靠地区分真假阳性结果。
3.5 单细胞捕获和分析要实现对HIV-1感染的治愈,主要挑战是清除病毒潜伏库。许多HIV-1根除策略基于“休克和杀灭”(shock and kill)的思路,需重新激活病毒潜伏库,这也是HIV-1根除研究的前沿热点。但人们对HIV-1转录活性与重新激活的感染细胞数量之间的关联知之甚少,现有的以细胞团或组织块为样本的检测手段无法将转录数量与细胞个体联系起来,潜伏逆转剂(latency reversing agent,LRA)可增加已激活的细胞中病毒转录水平,非活化的潜伏态感染的细胞被激活并重新表达HIV-1 RNA。Yucha等[39]通过ddPCR油包水微滴包裹单个CD4+ T细胞、巨噬细胞和脑源性神经胶质细胞,在细胞水平直接对体外经LRA处理后产生未剪切RNA和多重剪接RNA的CD4+ T细胞进行定量,检测数十万个CD4+ T细胞中的HIV-1转录活性,并分析抗反转录病毒治疗中的HIV-1感染者的CD4+ T细胞。他们分离了包含单个CD4+ T细胞或8E5细胞和感染HIV的外周血单个核细胞(peripheral blood mononuclear cell,PBMC)的微滴,对基因组DNA和mRNA进行定量。结果表明,经HIV-1再激活的单个细胞数量与通过传统测量方法获得的总HIV-1 RNA水平无关,而LRA介导的usRNA和msRNA表达可能在临床样品间乃至单个细胞间存在异质性。该研究突出了直接分析单个细胞的重要性,可更好地理解病原微生物与宿主细胞之间的相互作用。
4 结语dPCR的主要技术特点是可实现复杂样本中小概率事件的差异性检测。dPCR的出现,特别是对ddPCR的应用,将核酸分子的定量分析带入无须标准物质的绝对定量时代,有助于检验结果的追溯和比较。dPCR对疾病相关核酸分子标记的定量检测,特别是对痕量靶标分子的定量检测,为疾病早期诊断、疗效评估和预后提供了更具重复性和再现性的数据支持。在感染性疾病领域,未来对病原微生物的检测很可能从静态到动态、从定性到定量发展,治疗也可秉持“精准医疗”的理念,根据病原微生物的动态变化适时调整治疗方案,优化对感染患者的全程管理。dPCR特别是ddPCR技术将为这个理念的实现提供重要的支撑。未来dPCR技术特别是更加灵活的ddPCR应朝着高通量、自动化、多通道、多功能的方向发展。
| [1] |
Diaz LJ Jr, Bardelli A. Liquid biopsies:genotyping circulating tumor DNA[J]. J Clin Oncol, 2014, 32(6): 579-586.
[DOI]
|
| [2] |
Weaver S, Dube S, Mir A, Qin J, Sun G, Ramakrishnan R, Jones RC, Livak KJ. Taking qPCR to a higher level:analysis of CNV reveals the power of high throughput qPCR to enhance quantitative resolution[J]. Methods, 2010, 50(4): 271-276.
[DOI]
|
| [3] |
Higuchi R, Fockler C, Dollinger G, Watson R. Kinetic PCR analysis:real-time monitoring of DNA amplification reactions[J]. Biotechnology (NY), 1993, 11(9): 1026-1030.
[URI]
|
| [4] |
Hindson CM, Chevillet JR, Briggs HA, Gallichotte EN, Ruf IK, Hindson BJ, Vessella RL, Tewari M. Absolute quantification by droplet digital PCR versus analog real-time PCR[J]. Nat Methods, 2013, 10(10): 1003-1005.
[DOI]
|
| [5] |
Sykes PJ, Neoh SH, Brisco MJ, Hughes E, Condon J, Morley AA. Quantitation of targets for PCR by use of limiting dilution[J]. Biotechniques, 1992, 13(3): 444-449.
[URI]
|
| [6] |
Brisco MJ, Codon J, Hughes E, Neoh SH, Sykes PJ, Seshadri R, Morley AA, Toogood I, Waters K, Tauro G, Ekert H. Outcome prediction in childhood acute lymphoblastic leukaemia by molecular quantification of residual disease at the end of induction[J]. Lancet, 1994, 343(8891): 196-200.
[DOI]
|
| [7] |
Vogelstein B, Kinzler KW. Digital PCR[J]. Proc Natl Acad Sci USA, 1999, 96(16): 9236-9241.
[DOI]
|
| [8] |
Nakano M, Komatsu J, Matsuura S, Takashima K, Katsura S, Mizuno A. Single-molecule PCR using water-in-oil emulsion[J]. J Biotechnol, 2003, 102(2): 117-124.
[URI]
|
| [9] |
Pinheiro LB, Coleman VA, Hindson CM, Herrmann J, Hindson BJ, Bhat S, Emslie KR. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification[J]. Anal Chem, 2012, 84(2): 1003-1011.
[DOI]
|
| [10] |
Hindson BJ, Ness KD, Masquelier DA, Belgrader P, Heredia NJ, Makarewicz AJ, Bright IJ, Lucero MY, Hiddessen AL, Legler TC, Kitano TK, Hodel MR, Petersen JF, Wyatt PW, Steenblock ER, Shah PH, Bousse LJ, Troup CB, Mellen JC, Wittmann DK, Erndt NG, Cauley TH, Koehler RT, So AP, Dube S, Rose KA, Montesclaros L, Wang S, Stumbo DP, Hodges SP, Romine S, Milanovich FP, White HE, Regan JF, Karlin-Neumann GA, Hindson CM, Saxonov S, Colston BW. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number[J]. Anal Chem, 2011, 83(22): 8604-8610.
[DOI]
|
| [11] |
Whale AS, Bushell CA, Grant PR, Cowen S, Gutierrez-Aguirre I, O'Sullivan DM, Žel J, Milavec M, Foy CA, Nastouli E, Garson JA, Huggett JF. Detection of rare drug resistance mutations by digital PCR in a human influenza A virus model system and clinical samples[J]. J Clin Microbiol, 2016, 54(2): 392-400.
[URI]
|
| [12] |
Persuad D, Gay H, Ziemniak C, Chen YH, Piatak M Jr, Chun TW, Strain M, Richman D, Luzuriaga K. Absence of detectable HIV-1 viremia after treatment cessation in an infant[J]. N Engl J Med, 2013, 369(19): 1828-1835.
[DOI]
|
| [13] |
Bharuthram A, Paximadis M, Picton AC, Tiemessen CT. Comparison of a quantitative real-time PCR assay and droplet digital PCR for copy number analysis of the CCL4L genes[J]. Infect Genet Evol, 2014, 25: 28-35.
[DOI]
|
| [14] |
de Smith AJ, Walsh KM, Hansen HM, Endicott AA, Wiencke JK, Metayer C, Wiemels JL. Somatic mutation allelic ratio test using ddPCR (SMART-ddPCR):an accurate method for assessment of preferential allelic imbalance in tumor DNA[J]. PLoS One, 2015, 10(11): e0143343.
[DOI]
|
| [15] |
Whale AS, Devonshire AS, Karlin-Neumann G, Regan J, Javier L, Cowen S, Fernandez-Gonzalez A, Jones GM, Redshaw N, Beck J, Berger AW, Combaret V, Dahl Kjersgaard N, Davis L, Fina F, Forshew T, Fredslund Andersen R, Galbiati S, González Hernández Á, Haynes CA, Janku F, Lacave R, Lee J, Mistry V, Pender A, Pradines A, Proudhon C, Saal LH, Stieglitz E, Ulrich B, Foy CA, Parkes H, Tzonev S, Huggett JF. International interlaboratory digital PCR study demonstrating high reproducibility for the measurement of a rare sequence variant[J]. Anal Chem, 2017, 89(3): 1724-1733.
[DOI]
|
| [16] |
Perkel MJ. Life science technologies:The digital PCR revolution[J]. Science, 2014, 344(6180).
[DOI]
|
| [17] |
Dong L, Meng Y, Sui Z, Wang J, Wu L, Fu B. Comparison of four digital PCR platforms for accurate quantification of DNA copy number of a certified plasmid DNA reference material[J]. Sci Rep, 2015, 5: 13174.
[DOI]
|
| [18] |
Dobnik D, Demšar T, Huber I, Gerdes L, Broeders S, Roosens N, Debode F, Berben G, Žel J. Inter-laboratory analysis of selected genetically modified plant reference materials with digital PCR[J]. Anal Bioanal Chem, 2018, 410(1): 211-221.
[URI]
|
| [19] |
Haynes RJ, Kline MC, Toman B, Scott C, Wallace P, Butler JM, Holden MJ. Standard reference material 2366 for measurement of human cytomegalovirus DNA[J]. J Mol Diagn, 2013, 15(2): 177-185.
[DOI]
|
| [20] |
Strain MC, Lada SM, Luong T, Rought SE, Gianella S, Terry VH, Spina CA, Woelk CH, Richman DD. Highly precise measurement of HIV DNA by droplet digital PCR[J]. PLoS One, 2013, 8(4): e55943.
[DOI]
|
| [21] |
Taylor SC, Carbonneau J, Shelton DN, Boivin G. Optimization of droplet digital PCR from RNA and DNA extracts with direct comparison to RT-qPCR:Clinical implications for quantification of Oseltamivir-resistant subpopulations[J]. J Virol Methods, 2015, 224: 58-66.
[DOI]
|
| [22] |
Dingle TC, Sedlak RH, Cook L, Jerome KR. Tolerance of droplet-digital PCR vs real-time quantitative PCR to inhibitory substances[J]. Clin Chem, 2013, 59(11): 1670-1672.
[DOI]
|
| [23] |
Sedlak RH, Kuypers J, Jerome KR. A multiplexed droplet digital PCR assay performs better than qPCR on inhibition prone samples[J]. Diagn Microbiol Infect Dis, 2014, 80(4): 285-286.
[DOI]
|
| [24] |
Cao Y, Raith MR, Griffith JF. Droplet digital PCR for simultaneous quantification of general and human-associated fecal indicators for water quality assessment[J]. Water Res, 2015, 70: 337-349.
[DOI]
|
| [25] |
Pavšič J, Žel J, Milavec M. Digital PCR for direct quantification of viruses without DNA extraction[J]. Anal Bioanal Chem, 2016, 408(1): 67-75.
[URI]
|
| [26] |
Hoffman NG, Cook L, Atienza EE, Limaye AP, Jerome KR. Marked variability of BK virus load measurement using quantitative real-time PCR among commonly used assays[J]. J Clin Microbiol, 2008, 46(8): 2671-2680.
[DOI]
|
| [27] |
Mukaide M, Sugiyama M, Korenaga M, Murata K, Kanto T, Masaki N, Mizokami M. High-throughput and sensitive next-generation droplet digital PCR assay for the quantitation of the hepatitis C virus mutation at core amino acid 70[J]. J Virol Methods, 2014, 207: 169-177.
[DOI]
|
| [28] |
Bloom K, Ely A, Mussolino C, Cathomen T, Arbuthnot P. Inactivation of hepatitis B virus replication in cultured cells and in vivo with engineered transcription activator-like effector nucleases[J]. Mol Ther, 2013, 21(10): 1889-1897.
[DOI]
|
| [29] |
Sedlak RH, Liang S, Niyonzima N, De Silva Feelixge HS, Roychoudhury P, Greninger AL, Weber ND, Boissel S, Scharenberg AM, Cheng A, Magaret A, Bumgarner R, Stone D, Jerome KR. Digital detection of endonuclease mediated gene disruption in the HIV provirus[J]. Sci Rep, 2016, 6: 20064.
[DOI]
|
| [30] |
Mock U, Machowicz R, Hauber I, Horn S, Abramowski P, Berdien B, Hauber J, Fehse B. mRNA transfection of a novel TAL effector nuclease (TALEN) facilitates efficient knockout of HIV co-receptor CCR5[J]. Nucleic Acids Res, 2015, 43(11): 5560-5571.
[DOI]
|
| [31] |
Provasi E, Genovese P, Lombardo A, Magnani Z, Liu PQ, Reik A, Chu V, Paschon DE, Zhang L, Kuball J, Camisa B, Bondanza A, Casorati G, Ponzoni M, Ciceri F, Bordignon C, Greenberg PD, Holmes MC, Gregory PD, Naldini L, Bonini C. Editing T cell specificity towards leukemia by zinc finger nucleases and lentiviral gene transfer[J]. Nat Med, 2012, 18(5): 807-815.
[DOI]
|
| [32] |
Berdien B, Mock U, Atanackovic D, Fehse B. TALEN-mediated editing of endogenous T-cell receptors facilitates efficient reprogramming of T lymphocytes by lentiviral gene transfer[J]. Gene Ther, 2014, 21(6): 539-548.
[DOI]
|
| [33] |
Weber ND, Stone D, Sedlak RH, De Silva Feelixge HS, Roychoudhury P, Schiffer JT, Aubert M, Jerome KR. AAV-mediated delivery of zinc finger nucleases targeting hepatitis B virus inhibits active replication[J]. PLoS One, 2014, 9(5): e97579.
[DOI]
|
| [34] |
Findlay SD, Vincent KM, Berman JR, Postovit LM. A digital PCR-based method for efficient and highly specific screening of genome edited cells[J]. PLoS One, 2016, 11(4): e0153901.
[DOI]
|
| [35] |
Miyaoka Y, Chan AH, Conklin BR. Detecting single-nucleotide substitutions induced by genome editing[J]. Cold Spring Harb Protoc, 2016.
[PubMed]
|
| [36] |
Mock U, Hauber I, Fehse B. Digital PCR to assess gene-editing frequencies (GEF-dPCR) mediated by designer nucleases[J]. Nat Protoc, 2016, 11(3): 598-615.
[DOI]
|
| [37] |
Miyaoka Y, Chan AH, Judge LM, Yoo J, Huang M, Nguyen TD, Lizarraga PP, So PL, Conklin BR. Isolation of single-base genome-edited human iPS cells without antibiotic selection[J]. Nat Methods, 2014, 11(3): 291-293.
[DOI]
|
| [38] |
McMahon TC, Blais BW, Wong A, Carrillo CD. Multiplexed single intact cell droplet digital PCR (MuSIC ddPCR) method for specific detection of enterohemorrhagic E. coli (EHEC) in food enrichment cultures[J]. Front Microbiol, 2017, 8: 332.
[DOI]
|
| [39] |
Yucha RW, Hobbs KS, Hanhauser E, Hogan LE, Nieves W, Ozen MO, Inci F, York V, Gibson EA, Thanh C, Shafiee H, El Assal R, Kiselinova M, Robles YP, Bae H, Leadabrand KS, Wang S, Deeks SG, Kuritzkes DR, Demirci U, Henrich TJ. High-throughput characterization of HIV-1 reservoir reactivation using a single-cell-in-droplet PCR assay[J]. EBioMedicine, 2017, 20: 217-229.
[DOI]
|
 2019, Vol. 14
2019, Vol. 14


