B组柯萨奇病毒(coxsackievirus B,CVB)属小RNA病毒科肠道病毒属,是人类病毒性心肌炎的主要病原[1]。根据美国疾病预防控制中心统计数据,CVB每年可导致大约500万人患肠道系统疾病,其中20%患有CVB3感染引起的急性心肌炎[2],在我国也有相应疾病的流行报道[3]。但目前尚无特异性有效抑制CVB感染的药物[4]。
CVB为无包膜单正链RNA病毒,全长7.4 kb。基因组只有1个开放读码框架和2个非编码区。开放读码框架可编码4个结构蛋白(VP1~VP4)和7个非结构蛋白(2A~2C和3A~3D)[5]。其中3D基因编码RNA依赖的RNA聚合酶,负责病毒基因组RNA的转录,合成子代病毒基因组,在CVB感染和复制过程中发挥关键作用[6]。黄病毒属的寨卡病毒[7]、丙型肝炎病毒[8]均为单正链RNA病毒。研究表明[7-8],以病毒RNA聚合酶为靶点的抑制剂抗病毒效果显著。
RNA干扰(RNA interference,RNAi)是指在进化过程中高度保守的双链RNA(double stranded RNA,dsRNA)诱发的基因沉默现象[9],能特异性剔除或关闭靶基因的表达[10],已广泛用于探索基因功能和基因治疗领域[11]。慢病毒载体能稳定介导基因沉默,不仅转染效率高,还可持续稳定地表达[12-13]。本研究旨在筛选作用于CVB 3D基因的短发夹RNA(short hairpin RNA, shRNA),并构建慢病毒Lenti-sh3D,为后续抗CVB致病机制的研究奠定基础。
1 材料与方法 1.1 材料HeLa细胞、293T细胞、CVB3 Woodruff株由本教研室保存,抗CVB3 3D抗体由本教研室制备。pLVTHM、pMD2.G、psPAX2载体(Addgene #12247、#12259、#12260)和StbI3感受态购自上海唯地生物技术有限公司。小干扰RNA(small interfering RNA, siRNA)由苏州吉玛基因股份有限公司合成,单链shRNA由苏州金维智生物科技有限公司合成。Annealing buffer for DNA oligos(5×)购自Beyotime公司,限制性内切酶ClaⅠ、限制性内切酶MluⅠ购自纽英伦生物技术有限公司,质粒DNA小量提取试剂盒、DNA凝胶回收试剂盒购自康宁生命科学有限公司,T4连接酶、DL15000 DNA Marker、6×Loading Buffer购自大连TaKaRa公司,聚丙烯酰胺凝胶电泳(polyacrylamide gel electrophoresis,PAGE)凝胶快速制备试剂盒购自EpiZyme公司,PageRuler预染蛋白Marker购自Thermo Fisher公司,6×SDS-PAGE蛋白上样缓冲液购自上海碧云天生物技术有限公司,辣根过氧化物酶(horseradish peroxidase,HRP)标记山羊抗兔IgG购自万类生物科技有限公司,GAPDH兔多克隆抗体购自美国Proteintech公司,超敏增强化学发光(enhanced chemiluminescence,ECL)即用型底物购自武汉博士德生物工程有限公司。1周龄BALB/c小鼠购自哈尔滨医科大学附属第二医院动物实验中心。
1.2 方法 1.2.1 细胞培养HeLa、293T细胞均于含10%胎牛血清的高糖DMEM培养基中培养,培养条件为37 ℃、5% CO2。
1.2.2 CVB3 3D siRNA靶向序列的设计根据美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)中CVB3 3D基因(2157045)的核酸序列,利用BLOCK-iTTM RNAi Designer公用设计软件(http://rnaidesigner.thermofisher.com/rnaiexpress/insert.do)设计并合成3对siRNA靶向序列及1对阴性对照序列(表 1)。
| siRNA | Target sequence | Site |
| siRNA-1043 | GCAAGGACTATGGATTAATCA | 1 043-1 063 |
| siRNA-1218 | GGAACCAAAGAATACCCAAGA | 1 218-1 238 |
| siRNA-1281 | GCACGAATATGAGGAGTTTAT | 1 281-1 301 |
| siRNA-NC | TCAGGAGGTCTCGCTAAGTCA | — |
分别将合成的3条siRNA经Lipofectamine 2000转染至已培养24 h的HeLa细胞(24孔板:5×104个),同时设置阴性对照组。转染48 h后,收集细胞沉淀并提取RNA和蛋白,通过定量反转录-聚合酶链反应(quantitative reverse transcription-polymerase chain reaction,RT-qPCR)和蛋白免疫印迹法筛选抑制效率最佳的siRNA序列用于设计并合成shRNA。合成的shRNA中,loop茎环结构为TTCAAGAGA,3 ′端加入TTTTT终止信号,5 ′端及3 ′端分别引入ClaⅠ和MluⅠ酶切位点(表 2)。
| shRNA | Primer |
| shRNA-1043 | CGCGTCCCCGCAAGGACTATGGATTAATCATTCAAGAGATGATTAATCCATAGT CCTTGCTTTTTGGAAAT |
| shRNA-1218 | CGCGTCCCCGGATCCAAAGAATACCCAAGATTCAAGAGATCTTGGGTATTCTTTGGAT CCTTTTTGGAAAT |
| Scrambled shRNA | CGCGTCCCCTCAGGAGGTCTCGCTAAGTCATTCAAGAGATGACTTAGCGAGACCTCCT- GATTTTTGGAAAT |
将合成的单链shRNA配对退火,并将退火产物进行200倍稀释后备用。pLVTHM载体质粒经ClaⅠ/MluⅠ双酶切并凝胶电泳,回收酶切产物。将退火产物与获得的线性化pLVTHM经T4连接酶于16 ℃连接过夜(不超过12 h),转化大肠埃希菌StbI3感受态,然后涂布于氨苄西林(ampicillin,Amp)抗性LB固体培养基平板,37 ℃过夜培养;随机挑取单克隆菌落,接种于Amp抗性LB液体培养基中,37 ℃过夜培养;提取质粒,酶切,行DNA测序鉴定(测序引物序列:TCGATATGTGTTCT-GGGAAA)。
将酶切及序列比对正确的pLVTHM-3DshRNA重组质粒经Lipofectamine 2000转染于已培养24 h的HeLa细胞(细胞数:5×104),转染48 h后观察荧光。收集细胞沉淀并提取RNA和蛋白,通过RT-qPCR和蛋白免疫印迹法筛选抑制效率最佳的pLVTHM-3DshRNA进行慢病毒包装。
1.2.5 慢病毒Lenti-sh3D表达载体的包装及干扰效果鉴定将1×106个293T细胞接种于6孔板,于37 ℃、5% CO2培养箱中常规培养24 h。细胞生长融合度达70%~80%时,将pLVTHM-3DshRNA质粒与慢病毒包装质粒(psPAX2和pMD2.G)利用Lipofectamine 2000共转染293T细胞进行病毒包装。慢病毒包装所需的3个质粒pLVTHM-3DshRNA、psPAX2和pMD2.G的比例为4:2:1。转染6 h后换新鲜完全培养液,继续培养48 h,并观察荧光。转染后48 h收取病毒上清液,用0.45 μm微孔滤器过滤,收集过滤液,即得病毒原液Lenti-sh3D。
将慢病毒Lenti-sh3D感染已培养24 h的HeLa细胞(24孔板:5×104个),48 h后用CVB3感染(TCID50=10-4.75/mL)24 h,同时设对照组。收集细胞沉淀并提取蛋白,用蛋白免疫印迹法检测抑制效果。
慢病毒Lenti-sh3D经腹腔注射感染1周龄BALB/c小鼠,再用CVB3感染,同时设对照组。5 d后取小鼠心脏组织,用蛋白免疫印迹法检测抑制效果。
1.2.6 慢病毒Lenti-sh3D滴度测定将1×104个293T细胞接种于96孔板,感染时细胞融合度为70%。将病毒原液用opti-MEM进行梯度稀释(1:10、1:100、1:1 000、1:10 000、1:100 000),吸去96孔板中的培养液,每孔加入100 μL各梯度稀释液,培养过夜,24 h后更换为完全培养基,培养3 d。荧光显微镜下观察,根据公式计算病毒滴度(滴度=荧光细胞个数×相应的稀释倍数)。
1.3 统计学方法实验数据均以x±s表示,每个实验至少重复3次,采用t检验,P < 0.05为差异有统计学意义。
2 结果 2.1 CVB 3D基因siRNA有效靶点的筛选根据NCBI中3D序列和在线公共设计软件,筛选出3条评分较高的靶向序列,合成siRNA后转染293T细胞,采用蛋白免疫印迹和RT-qPCR检测3D聚合酶的表达和病毒复制情况。蛋白免疫印迹结果显示,与对照组相比,siRNA-1043的干扰效果显著(P<0.001),3D聚合酶表达的抑制率约为50%(图 1A、1B)。RT-qPCR结果表明,与对照组相比,3条靶向序列均可有效降低CVB3 RNA,其中siRNA-1043抑制效果较好(P<0.001),抑制率为43.7%(图 1C)。综合以上结果,siRNA-1043抑制效果显著,筛选为shRNA候选序列,合成后用于制备慢病毒。
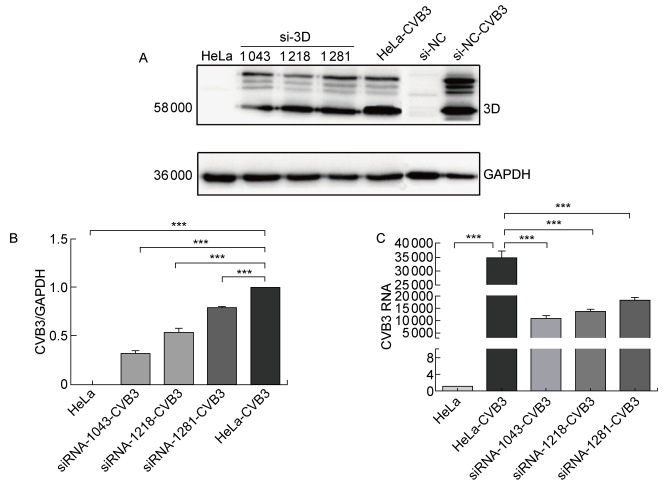
|
| A: Expression of 3D in CVB3-induced cells after siRNA interference by Western blotting. B: Quantitative analysis of protein expression. n=3, * * *P < 0.001. C: Expression of CVB3 RNA in CVB3-induced cells after siRNA interference by RT-qPCR. 图 1 CVB 3D基因siRNA有效靶点的筛选 Fig. 1 Screening of effective targets of CVB 3D siRNA |
将干扰效率较高的siRNA-1043和siRNA-1218的寡核苷酸序列合成shRNA,退火后与经ClaⅠ和MluⅠ双酶切的慢病毒骨架质粒pLVTHM连接,构建pLVTHM-3DshRNA重组质粒。ClaⅠ单酶切鉴定结果显示,酶切产物大小为11 085 bp,与预期结果相符。DNA测序结果显示,基因序列正确,无突变与缺失(图 2)。由此可见,pLVTHM-3DshRNA重组质粒构建成功。
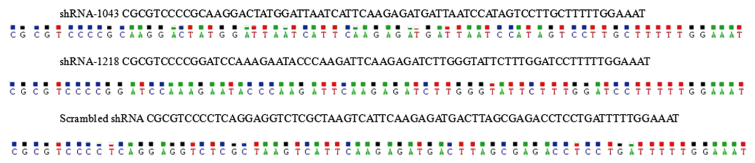
|
| Sequence of recombinant plasmid. 图 2 pLVTHM-3DshRNA重组质粒的鉴定 Fig. 2 Identification of pLVTHM-3DshRNA expression vector |
将所得正确的重组质粒转染HeLa细胞,48 h后观察荧光表达。结果显示,转染48 h后,镜下可见绿色荧光,表达量约为60%。
2.3 慢病毒Lenti-sh3D包装及滴度测试将pLVTHM-3DshRNA重组质粒与慢病毒包装质粒共转染293T细胞,转染48 h后,镜下可观察到绿色荧光,收集细胞上清液并过滤,即为含有慢病毒颗粒Lenti-sh3D的病毒液。取1×104个293T细胞接种于96孔板,感染时细胞融合度为70%~80%,梯度稀释病毒原液,以感染单位TU/mL表示病毒滴度。3 d后观测荧光,在10-4稀释度下可观测到5个细胞(图 3),经计算,制备的慢病毒原液滴度为5×107 TU。
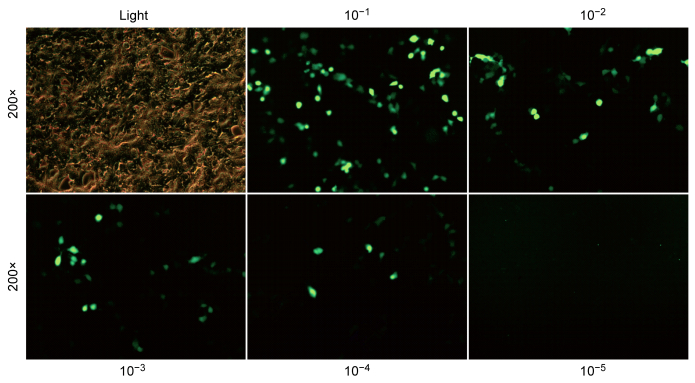
|
| Green fluorescence expression at each dilution. 图 3 慢病毒Lenti-sh3D滴度的测定 Fig. 3 Determination of Lenti-sh3D titer |
将病毒液加入HeLa细胞中,48 h后观察荧光(图 4C)。再用CVB3感染24 h,收集各组细胞沉淀,提取蛋白。蛋白免疫印迹结果显示(图 4A、4B),与对照组Lenti-mock相比,Lenti-sh3D感染组3D聚合酶表达水平明显降低(P<0.001),抑制率约为52%,表明慢病毒包装成功,且能显著抑制CVB3 3D聚合酶的表达水平。
将病毒液经腹腔注入小鼠体内,5 d后收集心脏组织,提取蛋白。蛋白免疫印迹结果显示(图 4D、4E),与对照组Lenti-mock相比,Lenti-sh3D感染组3D聚合酶表达水平降低(P<0.001),抑制率约为44%。

|
| A: Expression of 3D in CVB3-induced HeLa cells after lentiviral infection by Western blotting. B: Quantitative analysis of protein expression. n=3, * * *P < 0.001. C: Expression of green fluorescent protein was observed under fluorescence microscope (200×). D: Expression of 3D in CVB3-induced BALB/c mice after lentiviral infection by Western blotting. E: Quantitative analysis of protein expression. n=3, * * *P < 0.001. 图 4 慢病毒Lenti-sh3D干扰效果鉴定 Fig. 4 Interference effect of Lenti-sh3D |
CVB是病毒性心肌炎的主要病原,但致病机制尚未明确。3D基因编码RNA依赖的RNA聚合酶,负责病毒基因组RNA的转录,合成子代病毒基因组,在CVB感染和复制中发挥重要作用,选择3D作为靶基因可为CVB基因治疗奠定基础。目前,干扰靶点设计的网站有GeneLink、IDT、siRNA Wizard等。本研究利用BLOCK-iTTM RNAi Designer公用软件设计出3条siRNA,评分均较高。因此,首先进行靶点筛选。依据实验结果,虽然评分较高的几条siRNA均可有效抑制病毒3D聚合酶的表达及病毒的复制,但siRNA-1043抑制效果最显著,可抑制约50%的3D聚合酶表达和43.7%的病毒复制,被确定为后续shRNA的候选序列。由此提示,靶点序列的筛选很重要[14-15]。此外,相对shRNA,siRNA具有易筛选、价格低的优势,且两者在靶序列的选择上具有一致性。因此,本研究针对设计软件提供的多条靶序列首先采用siRNA进行筛选,确定最终干扰效率最佳的序列合成shRNA,然后用于慢病毒的制备,既降低了shRNA合成的消耗,又保障了慢病毒的高效性。
慢病毒载体是源于反转录病毒的载体,其转染效率高,可感染分裂期和非分裂期的细胞,还可容纳较大的基因片段,有良好的科研应用价值。近20年来,RNAi技术发展迅速,被广泛用于一些疾病治疗[16-17]。将慢病毒载体用于RNAi技术,成为基因功能研究和基因治疗领域的一种有力手段[18]。
本研究成功构建了pLVTHM-3DshRNA重组质粒,采用第二代慢病毒包装系统包装,成功获得15 mL慢病毒Lenti-sh3D,收获的病毒可显著抑制3D基因的表达,抑制率约为52%,病毒滴度为5×107TU。
通过相关实验,发现慢病毒载体法还存在很多缺点:①实验对细胞状态有很严格的要求。转染慢病毒重组质粒时,需进行48~72 h的长期培养,这对细胞密度和状态要求很高。②病毒包装表达率不是很高。采用三质粒包装系统进行慢病毒包装,细胞状态、转染效率等因素可能导致病毒滴度不是很高,在10-5稀释度就观测不到荧光,需对病毒进行纯化和浓缩以提高滴度。③保存的病毒在反复冻融后滴度下降。经-80 ℃反复冻融时病毒滴度降低很快,建议分装收集到的上清液。④实验操作繁琐,时间较长。
| [1] |
Zhai X, Wu S, Lin X, Wang T, Zhong X, Chen Y, Xu W, Tong L, Wang Y, Zhao W, Zhong Z. Stress granule formation is one of the early antiviral mechanisms for host cells against coxsackievirus B infection[J]. Virol Sin, 2018, 33: 314-322.
[DOI]
|
| [2] |
Kim DS, Nam JH. Characterization of attenuated coxsackievirus B3 strains and prospects of their application as live-attenuated vaccines[J]. Expert Opin Biol Ther, 2010, 10(2): 179-190.
[DOI]
|
| [3] |
汤晶晶, 张杰, 李凯, 樊帆, 李琼芬, 丁峥嵘. 云南省2011—2014年急性弛缓性麻痹病例非脊髓灰质炎肠道病毒分子流行病学特征[J]. 中国疫苗和免疫, 2017, 23(6): 608-612, 646. [CNKI]
|
| [4] |
Andréoletti L, Hober D, Becquart P, Belaich S, Copin MC, Lambert V, Wattré P. Experimental CVB3-induced chronic myocarditis in two murine strains: evidence of interrelationships between virus replication and myocardial damage in persistent cardiac infection[J]. J Med Virol, 1997, 52(2): 206-214.
[DOI]
|
| [5] |
Lindberg AM, Stålhandske PO, Pettersson U. Genome of coxsackievirus B3[J]. Virology, 1987, 156(1): 50-63.
[URI]
|
| [6] |
Kempf BJ, Barton DJ. Picornavirus RNA polyadenylation by 3Dpol, the viral RNA-dependent RNA polymerase[J]. Virus Res, 2015, 206: 3-11.
[DOI]
|
| [7] |
Xu HT, Hassounah SA, Colby-Germinario SP, Oliveira M, Fogarty C, Quan Y, Han Y, Golubkov O, Ibanescu I, Brenner B, Stranix BR, Wainberg MA. Purification of Zika virus RNA-dependent RNA polymerase and its use to identify small-molecule Zika inhibitors[J]. J Antimicrob Chemother, 2017, 72(3): 727-734.
[URI]
|
| [8] |
张秀杰, 韩晓峰, 欧阳红生. HCV治疗的新方法:RNA依赖的RNA聚合酶抑制剂[J]. 生命的化学, 2007, 27(3): 251-253. [DOI]
|
| [9] |
Mondal M, Klimov P, Flynt AS. Rewired RNAi-mediated genome surveillance in house dust mites[J]. PLoS Genet, 2018, 14(1): e1007183.
[DOI]
|
| [10] |
王永佳, 陈永胜, 李国瑞. RNAi在植物中的作用机理及其应用研究进展[J]. 内蒙古民族大学学报(自然汉文版), 2010, 25(1): 54-58. [URI]
|
| [11] |
MacKeigan JP, Murphy LO, Blenis J. Sensitized RNAi screen of human kinases and phosphatases identifies new regulators of apoptosis and chemoresistance[J]. Nat Cell Biol, 2005, 7(6): 591-600.
[DOI]
|
| [12] |
Frank SB, Schulz VV, Miranti CK. A streamlined method for the design and cloning of shRNAs into an optimized Dox-inducible lentiviral vector[J]. BMC Biotechnol, 2017, 17(1): 24.
[DOI]
|
| [13] |
Poling BC, Tsai K, Kang D, Ren L, Kennedy EM, Cullen BR. A lentiviral vector bearing a reverse intron demonstrates superior expression of both proteins and microRNAs[J]. RNA Biol, 2017, 14(11): 1570-1579.
[DOI]
|
| [14] |
Rao DD, Vorhies JS, Senzer N, Nemunaitis J. siRNA vs. shRNA: similarities and differences[J]. Adv Drug Deliv Rev, 2009, 61(9): 746-759.
[DOI]
|
| [15] |
Li M, Rossi JJ. Lentiviral vector delivery of siRNA and shRNA encoding genes into cultured and primary hematopoietic cells[M/OL]. In: Carmichael GG. ed. Methods in Molecular Biology, vol 309: RNA Silencing. New Jersey, USA: Humana Press, 2005. https://doi.org/10.1385/1-59259-935-4:261.
|
| [16] |
李跃萍, 宋丽萍, 邱曙东. 慢病毒载体在肿瘤基因治疗中的应用[J]. 现代肿瘤医学, 2006, 14(12): 1614-1617. [DOI]
|
| [17] |
陈志达, 丁丽华, 刘婕, 张亚楠, 朱杰, 罗晓丽, 刘国晓, 叶棋浓, 卫勃. 慢病毒载体介导E6AP基因敲低乳腺癌细胞株的构建及其功能检测[J]. 军事医学, 2014, 38(11): 863-866, 870. [URI]
|
| [18] |
Swamy MN, Wu H, Shankar P. Recent advances in RNAi-based strategies for therapy and prevention of HIV-1/AIDS[J]. Adv Drug Deliv Rev, 2016, 103: 174-186.
[DOI]
|
 2019, Vol. 14
2019, Vol. 14


