分泌霍乱毒素(cholera toxin,CT)的霍乱弧菌产毒株可导致感染者发生剧烈的水样腹泻[1],引起霍乱暴发和流行。霍乱弧菌包括一大类基因组明显差异的菌群,目前已发现超过200个血清群。引起霍乱暴发和流行的霍乱弧菌主要有O1和O139两个血清群。霍乱毒素由ctxAB基因编码,该基因位于霍乱弧菌溶源性丝状噬菌体CTXΦ上[2]。CTXΦ的gⅢ基因编码产物PⅢ能够与霍乱弧菌的毒素共调菌毛(toxin co-regulated pilus, TCP)中的主要亚单位TcpA发生相互作用,因此,CTXΦ能以TCP为受体感染霍乱弧菌,并以霍乱弧菌染色体上的dif序列为特异整合位点,将CTXΦ基因组整合到霍乱弧菌染色体中,导致ctxAB发生水平转移,形成新的产毒霍乱弧菌[3]。
CTXΦ的基因组由重复序列2(repeat sequence 2,RS2)和核心区组成。RS2中含有与噬菌体复制和整合相关的rstA和rstB基因,另外还有抑制这2个基因表达的rstR基因。rstR编码产生的RstR蛋白,能抑制CTXΦ超感染作用。核心区则由psh、cep、gⅢ、ace、zot和ctxAB基因组成,其结构相对保守[2, 4]。此外,在CTXΦ的基因组结构两侧,常见1个RS1结构。RS1由rstR、rstA、rstB和rstC组成,作为CTXΦ的辅助噬菌体,发挥促进CTXΦ的复制和整合作用[5]。rstA、rstB和rstC基因相对保守,而rstR基因的序列变异则非常明显,因此rstR基因是区分和命名不同CTXΦ亚型的主要依据。O1血清群古典型菌株和El Tor型菌株的rstR序列分别称为rstRclass和rstRET,两者之间的序列差异很大,且相互之间没有由rstR序列型介导的对相同CTXΦ超感染的免疫能力[6-7]。O139群菌株中的rstR大部分是rstRET[8], 后来,相继发现了新的rstR序列型,如rstRcalc和rstR6[9-10]。除了携带ctxAB的CTXΦ外,在霍乱弧菌中还发现了不携带ctxAB的CTXΦ,命名为nct-CTXΦ[11]或pre-CTXΦ[12]。rstR也是pre-CTXΦ中变异最为明显的基因[13-14]。除此之外,CTXΦ/pre-CTXΦ基因组在霍乱弧菌基因组中还具有多拷贝串联的复杂形式,可整合至霍乱弧菌大、小染色体上[13-14]。
目前对O1群和O139群产毒霍乱弧菌中CTXΦ/pre-CTXΦ家族成员基因组的结构和分布研究较多,但自环境中分离的不携带ctxAB的O1、O139群霍乱弧菌非产毒株同样具有丰富的pre-CTXΦ家族及其基因组排列的多样化[6-7, 13, 15-16]。后者基因组多态性也非常明显[17],其是否携带pre-CTXΦ的多样性也值得关注。
对于霍乱弧菌中CTXΦ和pre-CTXΦ基因组的分析,不但可丰富这类溶源性的丝状噬菌体分类及对霍乱弧菌中CTXΦ和pre-CTXΦ家族成员多样性的认识,也有助于研究产毒株的产生和新流行克隆群的形成。本研究中,我们筛选了4株携带pre-CTXΦ的O1和O139群霍乱弧菌,对其中的pre-CTXΦ进行了结构和整合位点的分析。结果发现了O139群霍乱弧菌非产毒株中的pre-CTXΦ基因组明显的序列型和排列多样化,显示出霍乱弧菌中该噬菌体家族的复杂进化及对宿主感染和整合的复杂性。
1 材料与方法 1.1 菌株和染色体DNA提取O139群霍乱弧菌菌株VC3741(序列号:SAMN11287978)、VC3193(序列号:SAMN11287980)、VC702(序列号:SAMN11287979)和O1群霍乱弧菌菌株VC1813(序列号:SAMN11287981)为本室保存。其中VC3741和VC3193分离于自然水体,VC702和VC1813分离于养殖牛蛙。4株霍乱弧菌均不携带霍乱毒素基因。菌株经LB培养基增菌培养,用Genomic Wizard试剂盒(Promega公司)按照说明书提取菌株DNA。
1.2 基因组框架图测定使用Hyper Prep Kit(Kapa Biosystems公司, kk8504)构建长度为350 bp插入片段的DNA测序文库。采用Illumina公司的Illumina HiSeqX Ten测序,双端读长为150 bp。
1.3 基因组完成图测定构建片段长度>10 kb的测序文库,使用PacBio RSII平台进行完成图测定。
1.4 pre-CTXΦ基因组序列的获取从GenBank中下载所有霍乱弧菌的基因组数据,筛选没有ctxAB但含有pre-CTXΦ的非产毒株。具体流程如下:通过Trimmomatic-0.38对原始reads数据进行质量控制;使用SPAdes-3.11.1对质控后的reads数据重新进行组合;之后使用本地blast+比对查找ctxAB、tcpA和gⅢ序列;对于完成图和只有组装序列的基因组数据,直接使用本地blast+比对查找ctxAB、tcpA和gⅢ序列。如果某一个基因组中tcpA和gⅢ均为阳性,且ctxAB基因为阴性,则认为是携带pre-CTXΦ的基因组。共筛选到6株菌株的基因组符合条件,分别是TUC_VC849_07(O1群)、TUC_VC182_14(O1群)、TUC_Me3(非O1/非O139群)、2710(O1群)、2174(O1群)和2012EL-1759(非O1/非O139群)。
1.5 tcpA和gⅢ进化树的构建及比较使用muscle分别对tcpA和gⅢ的核苷酸和氨基酸序列进行多序列比对,使用RAxML构建进化树;再通过R语言treespace包比较两者之间的进化关系。
2 结果 2.1 基因组测序及与产毒流行株的进化关系选择4株具有pre-CTXΦ基因簇的非产毒株进行了基因组测序,包括O1血清群中的3株(VC3193、VC3741和VC702)和O139血清群中的1株(VC1813)。在菌株VC1813和VC3193第2代测序的组装结果中,pre-CTXΦ位于完整的contig中(表 1)。而在菌株VC702和菌株VC3741第2代测序的组装结果中,pre-CTXΦ位于多个contig中,未得到完整的pre-CTXΦ结构。随后我们利用PacBio RSⅡ系统,对VC702和VC3741的基因组进行了第3代测序(长读长),使用该测序结果进行基因组序列组装,并使用第3代测序(短读长)的结果进行校正,最终获得这2株菌的基因组完成图(表 2)。
| Strains (Serogroups) | G+C% | N50 (bp) | Maximum contig (bp) | Contig amounts | Length (bp) |
| VC1813(O139) | 47.45 | 192 060 | 417 242 | 380 | 4 046 532 |
| VC3193(O1) | 47.5 | 246 422 | 401 242 | 388 | 3 944 082 |
| Strains (Serogroups) | G+C% | Chromosome 1 (bp) | Chromosome 2 (bp) | Plasmid (bp) | Length (bp) | Amount of plasmid |
| VC702(O1) | 47.57 | 3 145 023 | 1 097 465 | 33 998 | 4 276 486 | 1 |
| VC3741(O1) | 47.52 | 3 007 795 | 1 049 053 | - | 4 056 848 | 0 |
为确定这些菌株与霍乱弧菌O1/O139群流行株及与其他血清群的遗传学关系,我们选取了霍乱弧菌O1群(古典型和El Tor型)、O139群,以及非O1/非O139群共计44株基因组序列,其中O1群和O139群产毒株为1961年以来我国和其他国家流行的产毒株33株(图 1)。以O1群El Tor型霍乱弧菌产毒株N16961的基因组为参照,去除基因组中的重复片段区域以及重组片段区域, 获得核心基因组(core-genome)。核心基因组中单核苷酸多态性(single nucleotide polymorphism,SNPs)共18 500个,利用这些SNPs并通过Neighbor Joining方法构建了这些菌株基因的系统发生树(图 1),显示O1群El Tor型和O139群产毒株聚集形成了明显的产毒菌株簇,其SNPs差异在80~280个;O1群古典型产毒株O395及其他群的非产毒株表现出明显分化差异,其中O139群非产毒株VC1813和O1群非产毒株VC702、VC3193与O1群El Tor型产毒株SNPs差异数目达到3 300~18 500个。但菌株VC3741与O1群El Tor型产毒菌株簇聚集在一起,说明该菌株与该流行菌株簇的基因组高度相似。数据表明在O1群和O139群的非产毒菌株中,尽管具有广泛的基因组多样化,但个别菌株也具有与大流行菌株相似的基因组背景。

|
| The serogroups are marked in the brackets in the strain names. Non-toxigenic strain VC3741 was included within the cluster of the toxigenic O1 El Tor strains, which was shown in the box of dotted line. Strain O395 and the strains in this box, except for VC3741, are ctxAB+, the others are all ctxAB-. 图 1 4株非产毒株与霍乱弧菌O1/O139群流行株及与其他血清群的基因组进化关系 Fig. 1 The phylogenetic profile of the of the four V. cholerae strains compared with other toxigenic and non-toxigenic strains |
利用组装的4株研究菌株基因组序列,进行pre-CTXΦ基因组识别,发现在VC3741、VC3193和VC1813大染色体上有pre-CTXΦ基因组,而在菌株VC702的大、小染色体上均有pre-CTXΦ基因组。鉴定各基因组的基因结构、拷贝数及侧翼的菌株染色体基因,得到以上菌株4个pre-CTXΦ基因组结构(图 2)。

|
| 图 2 4株非产毒株pre-CTXΦ及RS1基因组的染色体整合排列 Fig. 2 Integrated genome arrangement of pre-CTXΦ and RS1 in the chromosomes of the strains used in this study |
在菌株VC3741的基因组中,pre-CTXΦ整合在大染色体邻近基因rtxA的dif1位点上,该位点是霍乱弧菌染色体I的DNA进行复制时双拷贝二聚体最后解离的位点, 而CTXΦ基因组利用该位点整合到霍乱弧菌染色体。它含有1个完整pre-CTXΦ基因组及前后各1个RS1结构,该区域为“RS1-RS2-core region-RS1”形式。进一步比对其rstR序列,发现pre-CTXΦ3741的rstR及下游RS1均为rstRET型,而其上游的RS1为rstR1型(相同序列的GenBank序列号:JX969003)。
菌株VC3193的pre-CTXΦ整合在基因rtxA旁侧的dif1位点上,含有2个pre-CTXΦ基因组拷贝,无RS1结构,此区域结构为(RS2-core region)×2形式,2个拷贝的rstR序列相同,为rstRclass。该种前体以往出现在产毒株中,这是首次在非产毒株基因组中发现的。
菌株VC1813中pre-CTXΦ整合在基因rtxA旁侧的dif2位点上。该前噬菌体中的rstR均为rstR232(GenBank序列号:DQ288668),前噬菌体为pre-CTX232Φ,也是重复的双拷贝,为(RS2-core region)×2形式。
在菌株VC702的大染色体中,pre-CTXΦ整合在基因rtxA旁侧的dif1位点上,有串联的2个相同拷贝,rstR序列型为rstZJ(相同序列的GenBank序列号:HM590227),因此前噬菌体为pre-CTXZJΦ。其下游还含有1个RS1,rstR为rstR-5型(相同序列的GenBank序列号:AF133308),该区域结构为(RS2-core region)×2+RS1形式。在该菌的小染色体上,有RS1在VCA0569和VCA05702基因之间dif1位点上的整合,没有pre-CTXΦ基因组。该区域实际有2个串联的RS1,rstR序列型均为rstR-4**(相同序列的GenBank序列号:AF133307)。这种pre-CTXΦ及RS1(GenBank序列号:SRIL00000000)的染色体整合形式系首次报道。另外,在第2个RS1的rstA基因中插入一个转座子结构,包含4个基因,分别编码重组酶、甲硫氨酸抑制子样蛋白、PIN结构域核酶和Tn3家族转座酶。经蛋白质库比对,发现这些基因与来自肺炎克雷白菌(Klebsiella pneumoniae)DA48896的质粒p48896_2(GenBank序列号:CP024431.1)上4个编码基因的序列一致性达99%。
2.3 非产毒株pre-CTXΦ的受体识别蛋白PⅢ及所在菌株的感染受体TcpA序列的多样性CTXΦ中PⅢ的吸收结构域(D2,134~252位氨基酸)能与TcpA的结构和相关结构域(SD_ID,121~191位氨基酸)相互作用,从而介导CTXΦ整合到细菌的染色体上[18]。但TcpA和PⅢ在不同菌株中序列可有所不同。进一步分析4株菌株中TcpA及其pre-CTXΦ携带编码的PⅢ的核苷酸和氨基酸序列。比较中还另外包括了9株霍乱弧菌:O1群El Tor型参考株N16961和O1群古典型参考株O395,非O1/非O139群菌株TUC_Me3和2012EL-1759,O1群菌株TUC_VC182_14、TUC_VC849_07、2710、2174[19]及O139群菌株ICDC-VC661。相应菌株的基因序列从GenBank中获取,要求这些菌株同时具有CTXΦ/pre-CTXΦ以及完整tcpA基因序列,并尽可能包括较多血清群。对这些菌株的tcpA核苷酸序列进行比对,进化树显示(图 3)本研究中的VC3741和VC3193与第7次大流行的El Tor型菌株N16961序列相同,VC1813也与已测序发现的两个菌株序列相同,但VC702显示了与所有菌株较大的差别,是一个新序列型。所有13株的TcpA氨基酸序列比较显示,氨基酸序列差异主要出现在αβloop区以及D区[20],在TcpA的结构和相互作用结构域(SD_ID)存在大量的变异位点(图 3)。在PⅢ的基因序列比对中(图 4),发现菌株VC3741和VC1813的pre-CTXΦ分别为pre-CTXETΦ和pre-CTX232Φ,均与第7次大流行的O1群El Tor型菌株N16961中带ctxAB毒素基因的CTXETΦ非常相似;菌株VC702的pre-CTXZJΦ中PⅢ的基因序列则比较独特,为新的序列亚型;菌株VC3193中携带pre-CTXclassΦ,其PⅢ的基因序列更接近于O1群古典型菌株O395中携带的CTXclassΦ。氨基酸序列比较(图 4)显示,氨基酸位点变异主要出现在D2区,该区是与TcpA相互作用的区域[18]。本研究中菌株VC3741、VC1813和VC702携带的3类pre-CTXΦ与El Tor型菌株N16961的基本相同,而VC3193与第6次大流行中的古典型菌株O395序列更为接近,但存在3个氨基酸残基的差异,属类古典型序列。
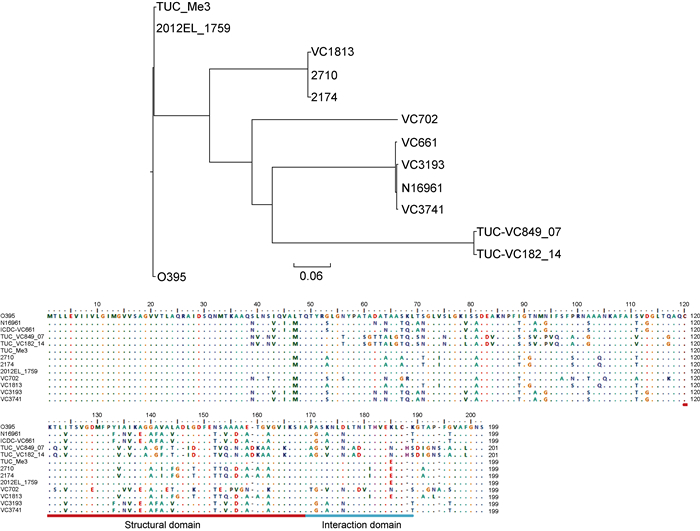
|
| 图 3 菌株tcpA基因序列进化树及蛋白氨基酸序列比对 Fig. 3 Phylogenetic analysis (up) of the tcpA genes and their amino acid sequence alignment (down) from the strains used in this study |
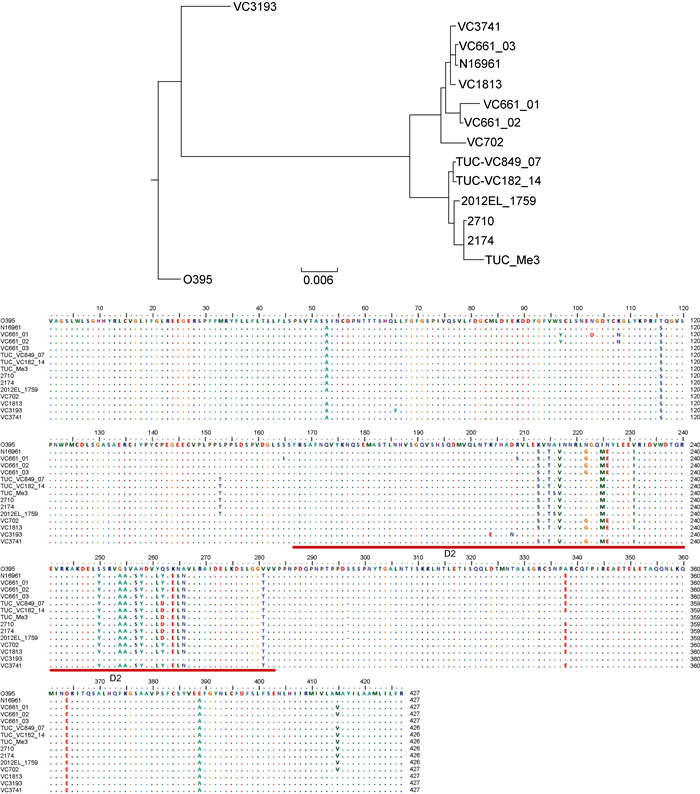
|
| 图 4 菌株CTXΦ/pre-CTXΦ的gⅢ基因序列进化树及蛋白氨基酸序列比对 Fig. 4 Phylogenetic analysis (up) of the CTXΦ/pre-CTXΦ gⅢ genes and their amino acid sequence alignment (down) from the strains used in this study |
早先对霍乱弧菌大流行菌株携带CTXΦ的研究发现O1群El Tor型和O139群产毒株主要携带El Tor型的CTXΦ基因组。但目前在非产毒株中,发现了更多具有不同序列型的pre-CTXΦ以及基因组上复杂的整合排列方式。因此,CTXΦ/pre-CTXΦ实际上是一个具有多种序列型的复杂家族。本研究报道了pre-CTXΦ新的序列型以及在菌株染色体上的排列方式。
典型的CTXΦ基因组长约7.2 kb,pre-CTXΦ长约6 kb,另外RS1长度约2.7 kb,这些序列还具有多拷贝串联的复杂形式。因此,应用第2代基因组测序难以组装出结构复杂的CTXΦ/pre-CTXΦ在染色体上的基因组排列。使用PCR确定基因间相互位置关系并通过Sanger测序验证构建其基因组物理图谱,将会涉及复杂的设计分析过程,且需要对多种可能组合进行验证和排除。本研究则采用第3代基因组测序方法,基于PacBio平台的长读长测序以及用短读长测序对序列进行矫正和补充,得到了完整的pre-CTXΦ在菌株中整合位置和基因组排列信息,这显示了基因组长读长测序以及整合高序列准确性的短读长测序分析长片段重复序列的优势。
产毒O1群El Tor型霍乱弧菌可引起全球流行,这类流行菌株携带的CTXΦ中,只检测到3种rstR序列型,而rstRET是占绝对优势的型别,极少出现2种或以上rstR序列型的CTXΦ同时存在于1个菌株的情况[15, 19, 21]。多数O139群产毒株中CTXΦ的结构也类似于El Tor型产毒株,但本研究也发现一些菌株中有较为复杂的CTXΦ/pre-CTXΦ整合形式,有的菌株CTXΦ/pre-CTXΦ共存且具有不同的rstR序列型[22]。4株不携带ctxAB的O1和O139群霍乱弧菌中,检测到5种rstR序列型别,3株具有串联双拷贝的pre-CTXΦ基因组,另2株具有复杂多样的RS1整合形式。这些RS1还具有多样的rstR序列型,与O1群El Tor型产毒流行株明显不同。非产毒株与产毒株基因组存在明显差异,从目前分析看,其携带的pre-CTXΦ也具有多样的序列型,即rstR序列多样;而产毒的O1和O139群流行菌株相对单一,主要为El Tor型CTXΦ,pre-CTXΦ很少见。在菌株VC3193中,存在古典型CTXΦ的前体pre-CTXclassΦ。霍乱弧菌中携带毒素基因ctxAB的CTXΦ有CTXclassΦ、CTXETΦ和CTXcalcΦ,后两者均被报道携带ctxAB的其前体形式,但CTXclassΦ的前体pre-CTXclassΦ以往一直未见报道。本研究发现的该基因组为第6次大流行的古典型菌株中CTXclassΦ的进化来源找到了前体。另外,菌株VC3741属于O1群非产毒株,在其基因组系统发生树上与O1群El Tor型产毒株聚集在一起,而其携带的pre-CTXΦ属于El Tor型,这可能提示具有与大流行菌株相似基因组的非产毒株也倾向于获得El Tor型的CTXΦ/pre-CTXΦ。
多种rstR序列型出现在1个菌株中,说明这些pre-CTXΦ和RS1具有相容性,即不同的rstR基因型产生的RstR蛋白不会对后来的噬菌体产生超感染的抑制,从而呈现了复杂的pre-CTXΦ/RS1基因组结构形式。有意思的是,在单个菌株小染色体上发现与肺炎克雷白菌质粒上序列一致的转座成分也插入到其中1个RS1序列中,这说明O139群环境菌株在自然环境中也发生跨种属的基因水平转移。
通过PⅢ的D2区与TcpA中的SD_ID区相互识别和结合,是CTXΦ/pre-CTXΦ感染霍乱弧菌的第一步。且不论在CTXΦ/pre-CTXΦ的PⅢ,还是在霍乱弧菌菌株TcpA及其相互作用结构域,均发现了多种多样的序列型。本研究中,我们通过对环境分离的O139群非产毒株基因组的分析,发现了还未曾报道的新的TcpA和PⅢ序列型,继续丰富了霍乱弧菌中这类丝状噬菌体及其受体的序列型。序列多样性往往意味着对不同环境的适应性改变,可能是为适应生存压力而形成的。而CTXΦ/pre-CTXΦ中PⅢ与TcpA相互作用的D2区也有序列多样化的现象,是否这些具有相互识别和相互作用的区域序列高变化有可能的对应关系,即PⅢ序列改变,能与不同TcpA序列型有不同的亲和力,还需要分析精细的蛋白结构域空间结构及其相互作用能力才能明确。
CTXΦ的感染和基因组整合是产生霍乱弧菌新产毒株的重要机制。目前发现CTXΦ/pre-CTXΦ家族具有复杂多样的成员。对这些具有不同rstR序列型成员及其霍乱弧菌基因组整合的研究,可认识这类噬菌体基因组的多样性和家族内基因组重组与进化等机制。更重要的是这些成员之间是否存在超感染的交叉免疫,尤其是当pre-CTXΦ先感染一个菌株时,其是否能抑制携带霍乱毒素基因的同型和其他rstR序列型的CTXΦ再感染,即可能成为产毒株。因此,pre-CTXΦ在菌株中的整合,能够在霍乱弧菌新产毒株产生上发挥影响作用。另外,不同PⅢ序列型的CTXΦ/pre-CTXΦ对不同TcpA序列型菌株的感染力是否有差异?两者间受体-配体的识别和相互作用特异性与能力决定了CTXΦ/pre-CTXΦ对不同遗传背景菌株的感染力,这有待于进一步的蛋白相互作用强度与特异性的实验来研究。
总之,本研究利用第3代基因组测序(长读长)的优势,获得了4株O1和O139群霍乱弧菌非产毒株中具有多拷贝重复长片段的pre-CTXΦ基因组结构,得到了前噬菌体pre-CTXΦ基因组及RS1基因组的排列图谱,发现了新的pre-CTXΦ基因组及与RS1的排列组合形式。另外,结合对菌株与噬菌体的TcpA/PⅢ序列分析,展示了不同基因组进化分支中霍乱弧菌中CTXΦ/pre-CTXΦ的感染及整合到宿主菌基因组的结构多样化,为研究这种具有自私特征的溶源性噬菌体的生存及携带毒素基因的CTXΦ在有和无pre-CTXΦ溶源化的霍乱弧菌中的传播能力提供了更多的论据。
| [1] |
Kaper JB, Morris JG Jr, Levine MM. Cholera[J]. Clin Microbiol Rev, 1995, 8(1): 48-86.
[DOI]
|
| [2] |
Waldor MK, Mekalanos JJ. Lysogenic conversion by a filamentous phage encoding cholera toxin[J]. Science, 1996, 272(5270): 1910-1914.
[DOI]
|
| [3] |
Waldor MK, Rubin EJ, Pearson GD, Kimsey H, Mekalanos JJ. Regulation, replication, and integration functions of the Vibrio cholerae CTXphi are encoded by region RS2[J]. Mol Microbiol, 1997, 24(5): 917-926.
[DOI]
|
| [4] |
Goldberg I, Mekalanos JJ. Cloning of the Vibrio cholerae recA gene and construction of a Vibrio cholerae recA mutant[J]. J Bacteriol, 1986, 165(3): 715-722.
[DOI]
|
| [5] |
Faruque SM, Kamruzzaman M, Asadulghani, Sack DA, Mekalanos JJ, Nair GB. CTXphi-independent production of the RS1 satellite phage by Vibrio cholerae[J]. Proc Natl Acad Sci USA, 2003, 100(3): 1280-1285.
[DOI]
|
| [6] |
Kimsey HH, Waldor MK. CTXphi immunity: application in the development of cholera vaccines[J]. Proc Natl Acad Sci USA, 1998, 95(12): 7035-7039.
[DOI]
|
| [7] |
Davis BM, Kimsey HH, Chang W, Waldor MK. The Vibrio cholerae O139 Calcutta bacteriophage CTXphi is infectious and encodes a novel repressor[J]. J Bacteriol, 1999, 181(21): 6779-6787.
[PubMed]
|
| [8] |
Zhang P, Zhou H, Diao B, Li F, Du P, Li J, Kan B, Morris JG Jr, Wang D. A molecular surveillance reveals the prevalence of Vibrio cholerae O139 isolates in China from 1993 to 2012[J]. J Clin Microbiol, 2014, 52(4): 1146-1152.
[DOI]
|
| [9] |
Li M, Kotetishvili M, Chen Y, Sozhamannan S. Comparative genomic analyses of the vibrio pathogenicity island and cholera toxin prophage regions in nonepidemic serogroup strains of Vibrio cholerae[J]. Appl Environ Microbiol, 2003, 69(3): 1728-1738.
[DOI]
|
| [10] |
芮勇宇, 阚飙, 高守一, 刘延清, 祁国明. 霍乱弧菌JS9803株CTX.EI TorΦ和nct-CTX.newO139Φ基因组克隆、测序及序列分析[J]. 热带医学杂志, 2006, 6(8): 861-864. [DOI]
|
| [11] |
阚飙, 祁国明, 刘延清, 刘彩莲, 高守一. 霍乱弧菌中存在不含霍乱毒素基因的噬菌体CTXΦ基因组[J]. 中华微生物学和免疫学杂志, 1999, 9(3): 175-179. [DOI]
|
| [12] |
Davis BM, Moyer KE, Boyd EF, Waldor MK. CTX prophages in classical biotype Vibrio cholerae: functional phage genes but dysfunctional phage genomes[J]. J Bacteriol, 2000, 182(24): 6992-6998.
[DOI]
|
| [13] |
Wang D, Wang X, Li B, Deng X, Tan H, Diao B, Chen J, Ke B, Zhong H, Zhou H, Ke C, Kan B. High prevalence and diversity of pre-CTXPhi alleles in the environmental Vibrio cholerae O1 and O139 strains in the Zhujiang River estuary[J]. Environ Microbiol Rep, 2014, 6(3): 251-258.
[DOI]
|
| [14] |
Wang H, Pang B, Xiong L, Wang D, Wang X, Zhang L, Kan B. The hybrid pre-CTXΦ-RS1 prophage genome and its regulatory function in environmental Vibrio cholerae O1 strains[J]. Appl Environ Microbiol, 2015, 81(20): 7171-7177.
[DOI]
|
| [15] |
Mutreja A, Kim DW, Thomson NR, Connor TR, Lee JH, Kariuki S, Croucher NJ, Choi SY, Harris SR, Lebens M, Niyogi SK, Kim EJ, Ramamurthy T, Chun J, Wood JL, Clemens JD, Czerkinsky C, Nair GB, Holmgren J, Parkhill J, Dougan G. Evidence for several waves of global transmission in the seventh cholera pandemic[J]. Nature, 2011, 477(7365): 462-465.
[DOI]
|
| [16] |
Maiti D, Das B, Saha A, Nandy RK, Nair GB, Bhadra RK. Genetic organization of pre-CTX and CTX prophages in the genome of an environmental Vibrio cholerae non-O1, non-O139 strain[J]. Microbiology, 2006, 152(Pt 12): 3633-3641.
[DOI]
|
| [17] |
Pang B, Yan M, Cui Z, Ye X, Diao B, Ren Y, Gao S, Zhang L, Kan B. Genetic diversity of toxigenic and nontoxigenic Vibrio cholerae serogroups O1 and O139 revealed by array-based comparative genomic hybridization[J]. J Bacteriol, 2007, 189(13): 4837-4849.
[DOI]
|
| [18] |
Kirn TJ, Lafferty MJ, Sandoe CM, Taylor RK. Delineation of pilin domains required for bacterial association into microcolonies and intestinal colonization by Vibrio cholerae[J]. Mol Microbiol, 2000, 35(4): 896-910.
[DOI]
|
| [19] |
Domman D, Quilici ML, Dorman MJ, Njamkepo E, Mutreja A, Mather AE, Delgado G, Morales-Espinosa R, Grimont PAD, Lizárraga-Partida ML, Bouchier C, Aanensen DM, Kuri-Morales P, Tarr CL, Dougan G, Parkhill J, Campos J, Cravioto A, Weill FX, Thomson NR. Integrated view of Vibrio cholerae in the Americas[J]. Science, 2017, 358(6364): 789-793.
[DOI]
|
| [20] |
Lim MS, Ng D, Zong Z, Arvai AS, Taylor RK, Tainer JA, Craig L. Vibrio cholerae El Tor TcpA crystal structure and mechanism for pilus-mediated microcolony formation[J]. Mol Microbiol, 2010, 77(3): 755-770.
[DOI]
|
| [21] |
Zhang P, Zhou H, Kan B, Wang D. Novel ctxB variants of Vibrio cholerae O1 isolates, China[J]. Infect Genet Evol, 2013, 20: 48-53.
[DOI]
|
| [22] |
陈爱平, 阚飙, 陈拱立, 陈亢川, 刘延清. O139群霍乱弧菌染色体中整合两种CTXΦ前噬菌体基因组的多样性[J]. 中华微生物学和免疫学杂志, 2002, 22(3): 239-242. [DOI]
|
 2019, Vol. 14
2019, Vol. 14


