细菌中组成核糖体的蛋白超过50个,但绝大多数核糖体蛋白的功能还没有完全被了解[1]。为探究它们的生物学功能,Fujio Kawamura课题组试图一一敲除枯草芽孢杆菌(Bacillus subtilis,B. subtilis)中57个核糖体蛋白编码基因,并成功得到16个敲除株,继而测定它们在32 ℃和37 ℃时的生长情况,发现L1、L22、L23、L34、L36、S6和S21敲除株的生长较野生株慢,而在45 ℃中除L1敲除株外,其余的均可恢复至野生株水平[2],提示核糖体蛋白组分缺陷可能导致细菌对温度更敏感[2]。此外,研究发现L1影响枯草芽孢杆菌芽孢形成,S21影响枯草芽孢杆菌的运动性能[2]。以上研究说明核糖体结构存在动态调控,其变化与细菌发育、环境适应等过程密切相关,但相关分子机制仍不明确。Eric J. Rubin课题组利用结核分枝杆菌(Mycobacterium tuberculosis,M. tuberculosis)Tn转座子突变库侵染小鼠,根据小鼠生存率筛选其毒力相关基因,其中发现核糖体蛋白S9(RpsI)、L35(RpmI)和L36(RpmJ)插入突变株在感染小鼠后第8周的存活率显著降低[3],提示核糖体蛋白参与调节细菌毒力,但相关分子机制仍需进一步研究。
本研究通过比较结核分枝杆菌(致病菌)与耻垢分枝杆菌(Mycobacterium smegmatis,M. smegmatis)(非致病菌)中核糖体蛋白RpsI、RpmI和RpmJ的氨基酸序列同源性,发现RpmI与RpmJ的氨基酸序列相似度极高,而RpsI N端20多个氨基酸存在较大差异。为进一步研究RpsI N端差异序列对核糖体结构与功能的影响,构建了以结核分枝杆菌rpsI替换耻垢分枝杆菌同源基因的重组菌株,观察RpsI N端序列的差异对分枝杆菌核糖体结构和功能的影响,期望对核糖体蛋白参与调节细菌毒力的相关分子机制研究提供数据。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒结核分枝杆菌H37Rv购自美国ATCC; 耻垢分枝杆菌mc2155菌株、大肠埃希菌(Escherchia coli,E. coli)DH5α菌株、大肠埃希菌BL21菌株、质粒pMV306、质粒pET22b(+)和质粒pYUB854均为本实验室储存。
1.1.2 主要试剂Middlebrook 7H9、7H10培养基购自美国BD公司; 胶回收试剂盒、聚合酶链反应(polymerase chain reaction,PCR)产物回收试剂盒、质粒提取试剂盒、RNA纯化试剂盒、Maxima H Minus First Strand cDNA合成试剂盒、Ni柱购自美国Thermo Fisher Scientific公司; 0.1 mm(直径)陶瓷珠购自美国Nalgene公司; 限制性核酸内切酶、DNaseⅠ(M0303L)、10×DNaseⅠBuffer(M0303S)购自美国NEB公司; 抗结核分枝杆菌Hsp65的鼠单克隆抗体(ab20519)购自英国Abcam公司; 辣根过氧化物酶(horseradish peroxidase,HRP)标记的山羊抗小鼠IgG(A9044)及山羊抗兔IgG(A0545)、蛋白定量试剂Bradford(B6916)购自美国Sigma公司; RNA提取试剂TRIzol(9109)购自日本Takara公司。
1.2 方法 1.2.1 细菌培养大肠埃希菌采用LB液体培养基(37 ℃、250 r/min)和LB固体培养基(37 ℃静置)分别培养16~24 h。耻垢分枝杆菌mc2155采用含0.2%甘油、0.05%吐温80的Middlebrook 7H9液体培养基(37 ℃、150 r/min)和含0.2%甘油的Middlebrook 7H10固体培养基(37 ℃静置)分别培养2~3 d。
1.2.2 重组菌株构建利用同源重组的方法进行基因敲除。以结核分枝杆菌H37Rv基因组DNA为模板,PCR扩增rpsI启动子区和编码区,并通过重叠PCR(overlapping PCR)将其连接。启动子区引物为,3442promoter-F: ATGCGAATTCGGG-ACCACACAGTCATTTTGA,3442promoter-R: GCTGGGGTGGTTTCGGTCATAGCGCTTACC-TTCTTTTCTC; 编码区引物为,3442-F: GAGA-AAAGAAGGTAAGCGCTATGACCGAAACCA-CCCCAGC,3442-R:ATGCGTTAACTCAGCG-CTTGCTGTACTGGG。PCR产物经酶切后连至pMV306质粒,电转至耻垢分枝杆菌mc2155,获得含有结核分枝杆菌rpsI(rpsI_Rv)的重组耻垢分枝杆菌,即mc2155∷pMV306_rpsI_Rv。同时,电转空载体pMV306质粒至耻垢分枝杆菌mc2155,得到Ms_rpsI_Ms菌株(mc2155∷pMV306,空载对照菌株)。以耻垢分枝杆菌mc2155基因组DNA为模板,通过引物Ms3442KO-F1:ATGCCTTAAGGG-AAACCCGAAACGAGAAGG,Ms3442KO-R1:ATCGACCGGTGGGAAGTAGTTCTCCAGCGT和Ms3442KO-F2:ATGCGCTAGCCCCTGATCC-TGGTGCAGC,Ms3442KO-R2:ATCGCTCGA-GGTGTTGTTGCAGACGTTGGT扩增上下臂,得到的PCR产物经酶切后连至pYUB854质粒,并通过噬菌体转导的方法[4]将重组质粒导入mc2155∷pMV306_rpsI_Rv中,敲除耻垢分枝杆菌rpsI(rpsI_Ms),得到Ms_rpsI_Rv菌株(mc2155ΔrpsI_Ms∷pMV306_rpsI_Rv)。利用引物(P1: CGT-AGACATTGCGGAACACC,P2: GGTCGATTC-CTGGGTGCC)验证rpsI_Ms是否敲除成功,同时利用306质粒引物(P3: TTACCGCCTTTGAG-TGAGCT,P4: GATGCTGACAAACGAATAG-AG)验证rpsI_Rv是否整合成功。PCR验证正确后,进一步对PCR产物进行测序验证。挑取3个验证正确的克隆用于后续实验。
1.2.3 RpsI_Rv抗体制备以结核分枝杆菌H37Rv基因组DNA为模板,以Pet-3442F: ATG-CCATATGACCGAAACCACCCCAGCCCC,Pet-3442R: ATGCCTCGAGGCGCTTGCTGTACTG-GGGCG为引物,PCR扩增rpsI编码序列; 经NdeⅠ和XhoⅠ双酶切后,连至pET22b(+)质粒,得到重组质粒(pET22b_rpsI_Rv),转化至大肠埃希菌感受态细胞DH5α,并对PCR阳性克隆进行测序。得到的重组质粒热激转化大肠埃希菌感受态细胞BL21。将重组BL21转接至含有100 μg/mL氨苄西林(Amp)的LB培养基中,37 ℃、250 r/min培养至OD600至0.6左右,加入0.5 mmol/L异丙基-β-D-硫代半乳糖苷(isopropyl β-D-1-thiogalactopyranoside,IPTG),置16 ℃、250 r/min诱导过夜。3 220×g离心收集菌体,并用Buffer A〔50 mmol/L Tris-HCl,500 mmol/L NaCl,10%甘油,5 mmol/L咪唑,2.5 mmol/L二硫苏糖醇(dithiothreitol,DTT)〕重悬至25 mL,利用6 mm的超声探头4 ℃超声破碎。破碎结束后,收集上清液全菌蛋白,并利用Ni柱进行纯化。目的蛋白进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate polyacrylamidegel electrophoresis,SDS-PAGE; 5%浓缩胶,12%分离胶),考马斯亮蓝染色,检测蛋白纯化情况。纯化好的目的蛋白移至截留分子量为10 000的超滤管,4 ℃、4 000×g离心40 min以浓缩目的蛋白。加入30 mL Buffer B〔50 mmol/L Tris-HCl,150 mmol/L NaCl,2.5 mmol/L DTT,0.25 mmol/L乙二胺四乙酸(ethylene diamine tetraacetic acid,EDTA)〕,4 ℃、4 000×g离心40 min平衡目的蛋白。最后加入1 mL Buffer B,重悬后加入25%甘油,于-80 ℃保存。将纯化后的RpsI_Rv送至上海艾比玛特(ABmart)公司,制备兔抗RpsI_Rv多克隆抗体。
1.2.4 蛋白质印迹法(Western blot)检测蛋白表达分别取对数生长期(OD600约1.0)的重组耻垢杆菌菌株Ms_rpsI_Rv与空载对照菌株Ms_rpsI_Ms 50 mL,3 220×g室温离心5 min,收集菌体,用10 mL磷酸盐缓冲液(phosphate buffered saline,PBS)重悬,3 220×g离心5 min,加入500 μL PBS悬浮菌体,吹打混匀,加入0.25 mL 0.1 mm(直径)陶瓷珠。用均质破菌仪剧烈振荡30 s,冰上静置1 min,连续破菌5次。4 ℃、16 000×g离心10 min,上清液即为可溶性蛋白组分。利用Bradford法[5]测定蛋白浓度,取5 μg蛋白进行SDS-PAGE。20 V电转至聚偏氟乙烯(polyvinylidene fluoride,PVDF)膜。3%牛血清蛋白(bovine serum albumin,BSA)室温封闭1 h,分别加入anti-RpsI_Rv兔源一抗(1:10 000)和anti-Hsp65鼠源一抗(1:10 000)(内参蛋白),室温孵育2 h。Tris-HCl缓冲液(Tris-buffered saline with Tween 20,TBST)室温洗5遍,每次5 min。加入HRP标记的山羊抗兔IgG(1:10 000)二抗和山羊抗鼠IgG(1:10 000)二抗,室温孵育1 h,TBST在室温洗2遍,每次5 min。按1:1的比例避光配制显色液(Millipore),并加至PVDF膜上,于Tanon全自动化学发光仪(上海天能科技有限公司,Tannon 4600)进行显色,并分析图像。
1.2.5 RNA的提取将-80 ℃保存的Ms_rpsI_Rv与Ms_rpsI_Ms接种至7H9液体培养基,37 ℃、150 r/min培养至稳定期(OD600约2.0),按1%的比例转接至10 mL 7H9液体培养基,37 ℃、150 r/min培养至对数期(OD600约0.8)。3 220×g离心5 min收集菌体,并用1 mL TRIzol试剂[6]重悬,重悬的菌体用均质破菌仪进行破碎,共破碎5次,每次30 s,间隔1 min。破碎完成后,16 000×g、4 ℃离心5 min,收集上清液至含Phase Lock Gel的管中,并加入0.3 mL氯仿,剧烈震荡10 s,室温放置10 min使其分层。16 000×g、4 ℃离心10 min,收集上层水相至新的EP管,并加入等体积异丙醇,于-20 ℃静置30 min。16 000×g、4 ℃离心20 min,沉淀物用75%乙醇洗1次,室温干燥后,加入85 μL RNase-free H2O、5 μL DNaseⅠ和10 μL 10×DNaseⅠbuffer〔10 mmol/L Tris-HCl(pH 7.6),2.5 mmol/L MgCl2,0.5 mmol/L CaCl2〕,37 ℃处理30 min。最后利用RNA纯化试剂盒进行纯化,得到的RNA保存于-80 ℃。
1.2.6 反转录PCR利用Maxima H Minus First Strand cDNA合成试剂盒反转录合成cDNA。分别以rpsI_Rv和rpsI_Ms的cDNA为模板,以相应的RNA为对照检测DNA污染情况。分别利用引物RpsI_Rv_F:CGCAGCTTGGAGGACTA-CTT,RpsI_Rv_R:GGCGATACCAGAATC-AATGC和RpsI_Ms_F: TCAGTTCGACAT-CTACGCCC,RpsI_Ms_R:TGTACTGAGG-AGCCTTACGG进行25个循环的PCR,最后用1%琼脂糖凝胶电泳观察条带相对亮度。
1.2.7 生长曲线的测定将-80 ℃保存的Ms_rpsI_Rv与Ms_rpsI_Ms分别接种至7H9液体培养基,37 ℃、150 r/min培养至稳定期(OD600约2.0),转接至20 mL 7H9液体培养基,并将OD600调至0.02,分别置于30 ℃、37 ℃和45 ℃,于150 r/min培养,每隔3 h测定菌液的OD600,记录实验数据,绘制生长曲线。利用不同的克隆重复3次实验。
1.2.8 重组菌株MIC90的测定采用通用的液体倍比稀释法测定药物MIC90[7]。用7H9液体培养基将Ms_rpsI_Rv与Ms_rpsI_Ms于37 ℃、150 r/min培养至OD600约1.0,并用7H9液体培养基将其稀释至OD600约0.01;同时将待测定的抗生素用7H9液体培养基进行倍比稀释,分别加入待测菌液中,于37 ℃、150 r/min培养1~2 d。以未加抗生素的菌液为对照,培养相同天数,测定OD600。若最后测得OD600值低于对照OD600值的10%,则该浓度梯度为药物的MIC90。利用不同的克隆重复测定3次。
1.2.9 统计学分析实验至少重复3次,实验数据进行t检验,P < 0.05为有统计学差异。采用Graphpad Prism 6.0软件进行统计并作图。
2 结果 2.1 分枝杆菌核糖体蛋白RpsI、RpmI和RpmJ氨基酸序列保守性分析利用美国国家生物信息中心生物大分子序列比对搜索工具(NCBI BLAST)比较结核分枝杆菌H37Rv与耻垢分枝杆菌mc2155核糖体蛋白RpsI、RpmI和RpmJ氨基酸序列的相似性,结果RpsI、RpmI和RpmJ氨基酸序列一致性分别达83.85%、84.38%和94.59%(图 1A),其中RpmI和RpmJ的氨基酸覆盖率为100%,而RpsI的氨基酸覆盖率仅86%(图 1A)。利用Bioedit软件进一步比对这两种菌株的RpsI、RpmI和RpmJ氨基酸序列相似性,结果显示RpmJ的相似性为100%,RpmI和RpsI相似性均为89%(图 1A),且RpsI氨基酸序列差异主要集中在蛋白序列的N端(图 1B)。提示结核分枝杆菌H37Rv与耻垢分枝杆菌mc2155核糖体蛋白RpsI的氨基酸序列差异较大,且主要集中在序列的N端。
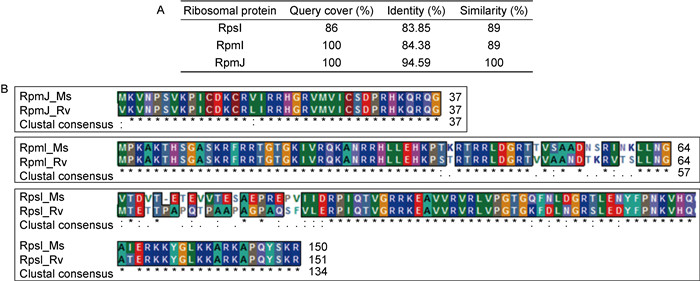
|
| A: Systematic comparison of RpsI, RpmI and RpmJ amino acid sequence between M. tuberculosis and M. smegmatis. B: Sequence similarity of RpsI, RpmI and RpmJ between M. tuberculosis and M. smegmatis. 图 1 RpsI、RpmI及RpmJ序列比对分析 Fig. 1 Sequence analyses of mycobacterial RpsI, RpmI and RpmJ |
为证明分枝杆菌RpsI N端差异序列可能影响核糖体的结构和功能,利用整合质粒pMV306将rpsI_Rv整合至耻垢分枝杆菌mc2155基因组中,并敲除其内源性基因rpsI_Ms,获得重组耻垢菌株mc2155 ΔrpsI∷pMV306_rpsI_Rv(Ms_rpsI_Rv)(图 2A)。利用PCR对该重组菌株进行验证,结果显示rpsI_Ms敲除成功,且rpsI_Rv成功整合至耻垢分枝杆菌基因组中(图 2B)。通过对PCR产物测序,进一步证实该菌株构建成功。
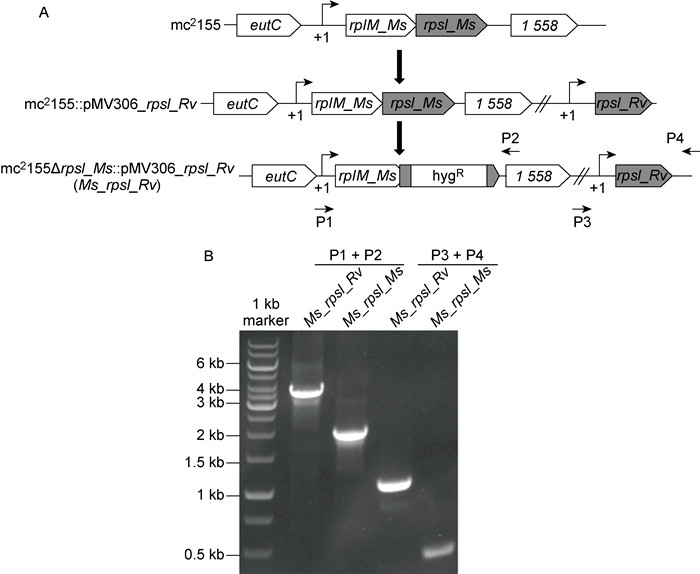
|
| A: Schematic diagram of strain genotypes and the process of construction. B: Verification of recombinant strain by PCR. P1 and P2 primers were used to genotype the rpsI_Ms locus. P3 and P4 primers were used to verify the insertion of plasmid containing rpsI_Rv. 图 2 重组菌株构建与验证 Fig. 2 Strain construction and verification |
为在蛋白水平上检测RpsI_Rv是否在Ms_rpsI_Rv中成功表达,本研究需制备该蛋白的特异性抗体。利用蛋白表达质粒pET22b(+)表达并纯化RpsI_Rv蛋白; SDS-PAGE检测RpsI_Rv蛋白的诱导和纯化情况,结果显示0.5 mmol/L IPTG于16 ℃可成功诱导表达RpsI_Rv。在纯化过程中,30 mmol/L咪唑能将绝大部分杂蛋白洗脱,用100 mmol/L咪唑洗脱的目的蛋白纯度较高(图 3)。最后用该纯化蛋白制备了兔源抗RpsI_Rv多克隆抗体,酶联免疫吸附试验(ELISA)测定该抗体的效价为1 600 000。
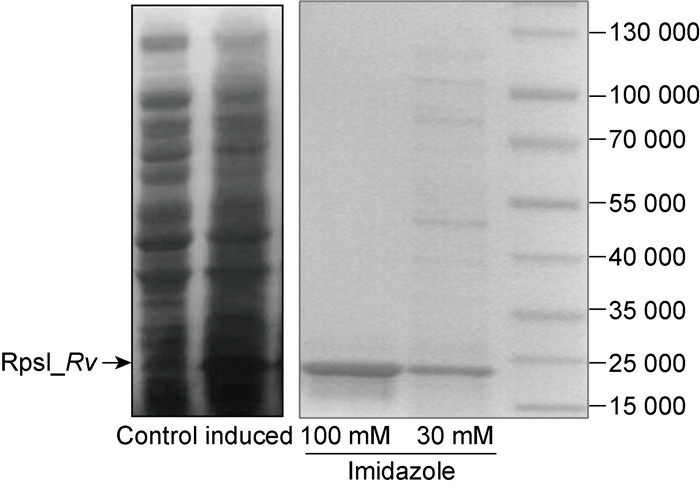
|
| Purification of M. tuberculosis RpsI expressed in E.coli strain BL21. RpsI_Rv was induced by 0.5 mM IPTG (left) and affinity-purified by NTA-agarose (right). 图 3 RpsI_Rv蛋白表达纯化 Fig. 3 Expression and purification of RpsI_Rv |
该重组耻垢菌株rpsI被结核分枝杆菌相应基因替换,并整合至基因组另一位点。为了验证rpsI_Rv易位插入是否影响rpsI_Rv表达,利用TRIzol法提取Ms_rpsI_Rv和对照菌株Ms_rpsI_Ms对数生长期的总RNA,经反转录成cDNA后,以RNA为对照,分别用相应的引物进行25轮的PCR,扩增rpsI_Rv和rpsI_Ms。电泳结果显示该重组菌株rpsI_Rv RNA的表达量与对照菌株rpsI_Ms一致(图 4A),表明rpsI_Rv的异位整合不影响rpsI_Rv的表达。同时利用RpsI_Rv多克隆抗体对重组菌株中的RpsI_Rv进行蛋白免疫印迹检测,结果显示RpsI_Rv在Ms_rpsI_Rv中成功表达,但是空载对照菌株Ms_rpsI_Ms的信号较弱(图 4B)。
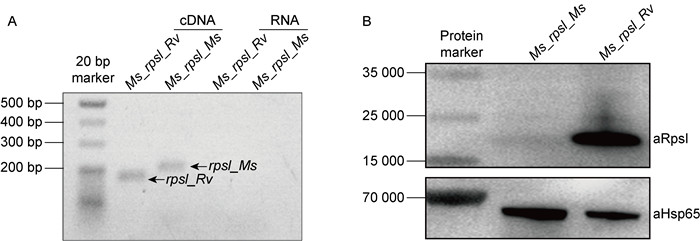
|
| A. The relative RNA expression of rpsI_Rv and rpsI_Ms was measured by reverse transcript PCR using corresponding primers, purified RNA was used as template to exclude DNA contamination. B. The expression of RpsI_Rv was assessed by Western blot using antibody against RpsI_Rv. Hsp65 was used as the internal reference. 图 4 rpsI_Rv和rpsI_Ms表达情况的检测 Fig. 4 Detecting the relative expression of rpsI_Rv and rpsI_Ms |
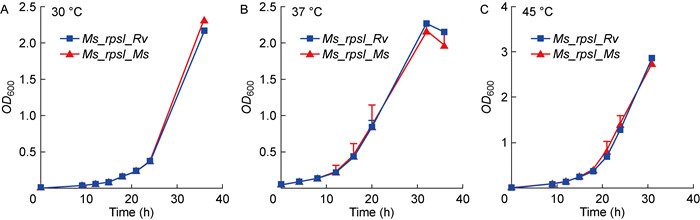
通过绘制不同温度下重组菌株和空载对照菌株的生长曲线,研究RpsI的改变是否影响Ms_rpsI_Rv在不同温度下的生长速率。结果如图 5所示,Ms_rpsI_Rv不同克隆之间的生长特性在30 ℃、37 ℃(正常生长温度)和45 ℃下没有显著差异(P>0.05),且与Ms_rpsI_Ms相比也未见显著差异(P>0.05)。以上结果说明RpsI N端序列的差异并不影响重组耻垢菌株的正常生长,且对温度的敏感性也未改变。
|
| 图 5 重组菌株Ms_rpsI_Rv与空载对照菌株Ms_rpsI_Ms的生长速率测定 Fig. 5 Growth rate of Ms_rpsI_Rv and Ms_rpsI_Ms |
为了检测RpsI N端的差异是否影响核糖体的结构,测定了作用于核糖体不同位点抗生素的MIC90,包括作用于30S亚基的链霉素(streptomycin)、阿米卡星(amikacin)和庆大霉素(gentamicin),以及作用于50S亚基的氯霉素(chlorampheniol)和红霉素(erythromycin)。结果如表 1所示,当用rpsI_Rv取代rpsI_Ms后,该重组菌株对链霉素、庆大霉素、氯霉素和红霉素的MIC90未发生改变,但对阿米卡星的敏感性升高了1倍。以上结果提示RpsI的替换可能影响了A位点的结构,使Ms_rpsI_Rv对阿米卡星更敏感。
| Antibiotics | MIC90 (μg/mL) | Binding site | Resistance mechanism | |
| Ms_rpsI_Rv | Ms_rpsI_Ms | |||
| Streptomycin | 0.10 | 0.10 | 30S decoding center | Missense mutations in rpsL [15] |
| Amikacin | 0.25 | 0.50 | A site of 16S rRNA | A1400G in 16S rRNA[16] |
| Erythromycin | 0.50 | 0.50 | 23S rRNA | Methylation of A2058 orA2058G of 23S rRNA[17] |
| Chloramphenicol | 4.00 | 4.00 | Peptide transferase center | Mutation of L4[18] |
| Gentamicin | 2.00 | 2.00 | H44 of 16S rRNA | Alteration in L6[19] |
核糖体一直被认为由固定的成分(核糖体蛋白和rRNA)组成,机械性地参与翻译过程。但目前认为核糖体存在异质性(ribosome heterogeneity)[8],包括核糖体组成成分和核糖体的数量,它们处于不断变化的过程中,并参与调节细胞适应环境的变化。Michael Hecke课题组发现枯草芽孢杆菌在遇到不同的环境压力如氧化压力、饥饿或热应激等条件时,细菌会上调不同的应激蛋白(stress proteins),其中包括Ctc蛋白[9]。Jorg Stulke课题组证明Ctc蛋白为核糖体蛋白,敲除ctc使枯草芽孢杆菌在热应激条件下无法形成芽孢[10]。大肠埃希菌核糖体蛋白bL31和bL36分别有两个同源蛋白,即bL31A、bL31B和bL36A、bL36B。Jaanus Remme课题组发现当大肠埃希菌从对数期过渡到稳定期时,核糖体的bL31A和bL36A被bL31B和bL36B替代[11]。以上研究说明细菌可通过调整核糖体的结构或组成使其适应环境,但相关作用机制仍不明确。
Eric J. Rubin课题组发现致病性结核分枝杆菌核糖体蛋白编码基因rpsI、rpmI和rpmJ插入突变后,该菌在小鼠体内的存活率显著降低(P < 0.05)[3],提示核糖体蛋白在结核分枝杆菌宿主适应过程中发挥着重要作用。耻垢分枝杆菌属于环境分枝杆菌,为非致病菌。本研究通过比较结核分枝杆菌和耻垢分枝杆菌RpsI、RpmI和RpmJ氨基酸序列的相似性,发现RpsI N端序列差异较大。为了研究RpsI N端序列的差异对分枝杆菌核糖体结构和功能的影响,用rpsI_Rv替换了rpsI_Ms,得到重组耻垢菌株。PCR结果显示该重组菌株构建成功,反转录PCR发现重组菌株和对照菌株rpsI RNA表达量一致,利用RpsI_Rv的特异性抗体检测该重组菌株RpsI_Rv表达成功,但对照菌株的RpsI_Ms信号较弱。由于rpsI_Rv和rpsI_Ms的RNA表达量一致,推测RpsI_Rv多克隆抗体识别的抗原表位主要集中在差异较大的N端,导致该多克隆抗体对RpsI_Ms的蛋白免疫印迹检测信号较弱。结核分枝杆菌与耻垢分枝杆菌核糖体蛋白RpsI氨基酸N端序列存在较大差异,序列的不同可能影响核糖体的结构,从而影响核糖体的功能,而核糖体与细菌的生长密切相关。同时,核糖体蛋白组分的缺陷可能使细菌对温度更加敏感[2],但通过测定该重组菌株不同克隆在不同温度下的生长曲线,发现rpsI_Rv异位插入不影响该重组菌株的正常生长,且与对照菌株相比,其生长特性也没有显著差异。通过测定作用于核糖体不同位点的5种抗生素的MIC90,发现rpsI_Rv异位插入后,该重组菌株不同克隆对这5种抗生素的敏感性并没有发生改变,但是与对照菌株相比,该重组耻垢菌株对阿米卡星的敏感性升高了1倍,由0.5 μg/mL变为0.25 μg/mL。阿米卡星主要与核糖体16S rRNA的H44结合[12],从而抑制翻译转位。通过分析已解析的耻垢分枝杆菌核糖体结构,发现RpsI的C端与阿米卡星作用位点接近[13],说明RpsI N端差异可能通过影响周围其他核糖体蛋白或rRNA来改变耻垢分枝杆菌核糖体的结构,从而使阿米卡星更容易结合至作用位点。测定发现卡介苗(BCG)对阿米卡星的MIC90也为0.5 μg/mL,但由于结核分枝杆菌与耻垢分枝杆菌细胞壁通透性及生长速度存在较大差异,因此无法通过比较两个物种的MIC90来推测药靶差异。
结核分枝杆菌与耻垢分枝杆菌的核糖体结构大体相似,但是在核糖体的外围区域存在菌种特异性的结构,这使两者核糖体结构存在一定的差异性[14]。比如在耻垢分枝杆菌核糖体中,23S rRNA的H15和H16a可形成吻合环(kissing loop),而这个吻合环可与bL9核糖体蛋白相互作用,导致bL9核糖体蛋白构象发生改变[13]。但是这种现象在结核分枝杆菌中并不存在,结核分枝杆菌bL9主要与23S rRNA的H54a相互作用,吻合环的存在并不影响bL9的构象[14]。本研究利用NCBI BLAST比较结核分枝杆菌与耻垢分枝杆菌bL9的氨基酸序列,发现相似性达75.5%,只是某些区域存在差异,这些部位的差异可能与周围其他核糖体成分相互作用有关。以上结果进一步说明,核糖体蛋白序列的细微差异可能会对核糖体结构甚至功能产生影响。
综上所述,结核分枝杆菌核糖体小亚基蛋白RpsI N端序列的差异可能微调核糖体的结构,但该结构变化是否会影响核糖体在特定环境下的功能还有待进一步研究。许多作用于核糖体的抗生素在感染性疾病的治疗中占据重要地位。例如,链霉素是第1个用于治疗结核病的抗生素,其主要作用于核糖体30S亚基解码中心,导致核糖体错译[12]。由于抗生素耐药情况越来越严重,临床上急需新的治疗药物,了解核糖体的结构和功能对于设计和筛选药物具有重要临床意义。
| [1] |
Shoji S, Dambacher CM, Shajani Z, Williamson JR, Schultz PG. Systematic chromosomal deletion of bacterial ribosomal protein genes[J]. J Mol Biol, 2011, 413(4): 751-761.
[DOI]
|
| [2] |
Akanuma G, Nanamiya H, Natori Y, Yano K, Suzuki S, Omata S, Ishizuka M, Sekine Y, Kawamura F. Inactivation of ribosomal protein genes in Bacillus subtilis reveals importance of each ribosomal protein for cell proliferation and cell differentiation[J]. J Bacteriol, 2012, 194(22): 6282-6291.
[DOI]
|
| [3] |
Sassetti CM, Rubin EJ. Genetic requirements for mycobacterial survival during infection[J]. Proc Natl Acad Sci U S A, 2003, 100(22): 12989-12994.
[DOI]
|
| [4] |
Bardarov S, Bardarov S, Pavelka MS, Sambandamurthy V, Larsen M, Tufariello J, Chan J, Hatfull G, Jacobs WR. Specialized transduction: an efficient method for generating marked and unmarked targeted gene disruptions in Mycobacterium tuberculosis, M. bovis BCG and M. smegmatis[J]. Microbiology (Reading), 2002, 148(Pt 10): 3007-3017.
[URI]
|
| [5] |
Cheng Y, Wei H, Sun R, Tian Z, Zheng X. Rapid method for protein quantitation by Bradford assay after elimination of the interference of polysorbate 80[J]. Anal Biochem, 2016, 494: 37-39.
[DOI]
|
| [6] |
Villa-Rodríguez E, Ibarra-Gámez C, de Los Santos-Villalobos S. Extraction of high-quality RNA from bacillus subtilis with a lysozyme pre-treatment followed by the Trizol method[J]. J Microbiol Methods, 2018, 147: 14-16.
[DOI]
|
| [7] |
Fan XY, Tang BK, Xu YY, Han AX, Shi KX, Wu YK, Ye Y, Wei ML, Niu C, Wong KW, Zhao GP, Lyu LD. Oxidation of dCTP contributes to antibiotic lethality in stationary-phase mycobacteria[J]. Proc Natl Acad Sci USA, 2018, 115(9): 2210-2215.
[DOI]
|
| [8] |
Xue S, Barna M. Specialized ribosomes: a new frontier in gene regulation and organismal biology[J]. Nat Rev Mol Cell Biol, 2012, 13(6): 355-369.
[DOI]
|
| [9] |
Völker U, Engelmann S, Maul B, Riethdorf S, Völker A, Schmid R, Mach H, Hecker M. Analysis of the induction of general stress proteins of Bacillus subtilis[J]. Microbiology (Reading), 1994, 140(Pt 4): 741-752.
[URI]
|
| [10] |
Schmalisch M, Langbein I, Stülke J. The general stress protein Ctc of Bacillus subtilis is a ribosomal protein[J]. J Mol Microbiol Biotechnol, 2002, 4(5): 495-501.
[PubMed]
|
| [11] |
Lilleorg S, Reier K, Pulk A, Liiv A, Tammsalu T, Peil L, Cate JHD, Remme J. Bacterial ribosome heterogeneity: Changes in ribosomal protein composition during transition into stationary growth phase[J]. Biochimie, 2019, 156: 169-180.
[DOI]
|
| [12] |
Wilson DN. The A-Z of bacterial translation inhibitors[J]. Crit Rev Biochem Mol Biol, 2009, 44(6): 393-433.
[DOI]
|
| [13] |
Hentschel J, Burnside C, Mignot I, Leibundgut M, Boehringer D, Ban N. The complete structure of the Mycobacterium smegmatis 70S ribosome[J]. Cell Rep, 2017, 20(1): 149-160.
[URI]
|
| [14] |
Yang K, Chang JY, Cui Z, Li X, Meng R, Duan L, Thongchol J, Jakana J, Huwe CM, Sacchettini JC, Zhang J. Structural insights into species-specific features of the ribosome from the human pathogen Mycobacterium tuberculosis[J]. Nucleic Acids Res, 2017, 45(18): 10884-10894.
[DOI]
|
| [15] |
Honoré N, Cole ST. Streptomycin resistance in mycobacteria[J]. Antimicrob Agents Chemother, 1994, 38(2): 238-242.
[DOI]
|
| [16] |
Alangaden GJ, Kreiswirth BN, Aouad A, Khetarpal M, Igno FR, Moghazeh SL, Manavathu EK, Lerner SA. Mechanism of resistance to amikacin and kanamycin in Mycobacterium tuberculosis[J]. Antimicrob Agents Chemother, 1998, 42(5): 1295-1297.
[DOI]
|
| [17] |
Vester B, Douthwaite S. Macrolide resistance conferred by base substitutions in 23S rRNA[J]. Antimicrob Agents Chemother, 2001, 45(1): 1-12.
[DOI]
|
| [18] |
Wolter N, Smith AM, Farrell DJ, Schaffner W, Moore M, Whitney CG, Jorgensen JH, Klugman KP. Novel mechanism of resistance to oxazolidinones, macrolides, and chloramphenicol in ribosomal protein L4 of the pneumococcus[J]. Antimicrob Agents Chemother, 2005, 49(8): 3554-3557.
[DOI]
|
| [19] |
Buckel P, Buchberger A, Böck A, Wittmann HG. Alteration of ribosomal protein L6 in mutants of Escherichia coli resistant to gentamicin[J]. Mol Gen Genet, 1977, 158(1): 47-54.
[DOI]
|
 2020, Vol. 15
2020, Vol. 15


