2. 复旦大学附属上海市公共卫生临床中心上海噬菌体与耐药研究所,上海 200540
2. Shanghai Institute of Phage, Shanghai Public Health Clinical Center Affiliated to Fudan University, Shanghai 201508, China
肺炎克雷伯菌是临床上常见的机会致病菌,可引起广泛感染,导致肺炎、尿路感染、菌血症和肝脓肿等疾病[1]。其毒力因子主要包括具有抗吞噬作用的荚膜多糖[2]、可引发宿主败血症和败血性休克的脂多糖(lipopolysaccharide,LPS)[3]、能获取宿主体内铁元素的铁载体[4]以及通过黏附于人类黏膜或上皮表面而致病的菌毛[5]。近年来,耐药肺炎克雷伯菌的感染率逐年升高,而可供选择的抗菌药物越来越少,导致耐多药(multidrug-resistant,MDR)肺炎克雷伯菌感染造成的死亡率高达40%~50%[6]。
肺炎克雷伯菌基因组大小为5~6 Mbp,G+C含量一般在50%~60%,编码5 000~6 000个基因。其中约1 700个基因是保守的,称为核心基因;其余基因则各有不同,称为辅助基因,包括染色体编码基因和可移动遗传元件(mobile genetic element,MGE)基因组。基于1 000~2 000个核心基因的系统进化树分析表明,肺炎克雷伯菌有数百个遗传型,彼此之间核苷酸差异在0.5%左右[7]。肺炎克雷伯菌基因组具有高度可塑性,其中通过水平而频繁转移的可移动基因组是导致其基因组多样性的主要原因。可移动基因组对肺炎克雷伯菌的耐药性和致病性有重要作用,因为其毒力基因和耐药基因通常位于MGE,并通过细菌之间的水平转移而获得。
细菌的可移动基因组是指一个细菌基因组内的全部MGE,包括质粒(plasmid)、插入序列(insertion sequence,IS)、前噬菌体(prophage)、整合性接合元件(integrative and conjugative element,ICE)等,其作用是介导细菌基因组内或细菌细胞之间DNA移动,且通过自身携带的功能基因促进宿主生长及提高对环境的适应性[8] (见图 1)。本文就肺炎克雷伯菌基因组中的可移动遗传成分及其在基因组进化中的作用进行综述。
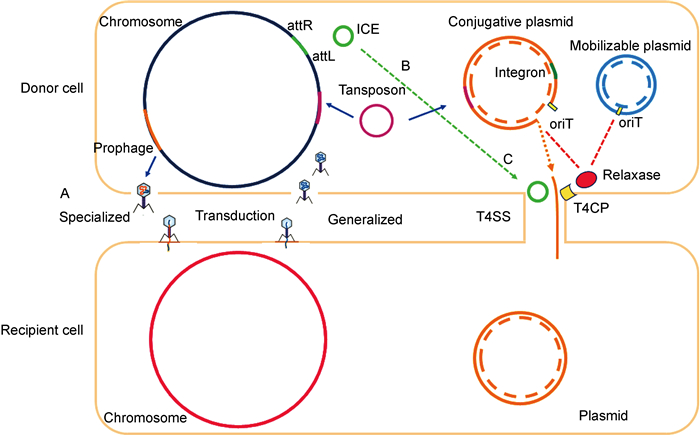
|
| A: Temperate phage DNA genome (yellow) is integrated into the chromosome (dark blue) as a prophage, then replicates, and assembles the host chromosome fragments (generalized transduction) or directly assembles its own DNA (specialized transduction) to lyse the cell and infect the recipient cell so that the DNA can be integrated into the chromosome (red) of the recipient host cell. B: ICEs (light green) are integrated into the chromosome of host cell through the encoded recombinase. Transposons (purple) are integrated into new sites on chromosomes or plasmids by non-homologous recombination, depending on their own transposases. The integron (dark green) uses a similar mechanism to move. C: Conjugative plasmid (orange) carries oriT, relaxase and T4CP, which are the necessary components for transmitting to other host cells by T4SS. 图 1 细菌细胞中可转移的遗传成分及其转移方式[8] Fig. 1 Mobile genetic components and transfer methods in bacterial cells |
质粒是最常见的MGE,通常指在宿主细胞中能够独立于染色体自主复制的线性或环状DNA分子。质粒不仅携带可促进宿主生存的优势基因,还携带其他MGE,如IS、整合子和转座子等。根据可否转移,质粒分为接合型、可移动型和不可移动型[9]。接合是DNA通过细胞间直接物理连接从供体菌转移到受体菌,一般由接合型质粒介导,是耐药基因转移的重要机制。接合转移是一个复杂过程,主要包括转移起始位点缺刻、DNA转移起始、双链分离、单链转移、供体和受体互补链的合成及再环化等[10]。对于接合型质粒和可转移质粒而言,接合机制中唯一的蛋白质成分是松弛酶,其为接合中的关键蛋白,是一种位点特异性核酸内切酶,能识别转移起点oriT(短DNA序列,是质粒接合转移中的唯一序列)。松弛酶在接合的初始和最终阶段发挥作用,即作用于供体中的oriT位点,使双链DNA切割成可转移的单链DNA,并在一些辅助蛋白(如T4CP)的帮助下将可移动的DNA单链通过Ⅳ型分泌系统(type Ⅳ secretion system,T4SS)或性菌毛等介质接合转移到受体细胞中[11]。F质粒是在细菌中发现的第1个接合质粒,也是最早发现的与抗生素抗性转移相关的质粒,是目前肠杆菌科中发现的最丰富的质粒类型[12]。
目前,对肺炎克雷伯菌质粒的分析主要基于基因组测序数据,使用replicon基因和relaxase(mob)基因进行质粒分析及分型。对已测序的肺炎克雷伯菌基因组进行分析,发现其基因组中质粒数量为0~12个。目前在肺炎克雷伯菌基因组中发现的质粒类型多达19种,主要包括IncFII、IncN、IncR和IncX3几类不相容类型,这些质粒通常比较大且为接合型质粒。在肺炎克雷伯菌基因组中,大多数水平获得的抗性基因主要通过这些不相容的大的接合型质粒传播,其中FIBk复制子为主要的抗性及毒力质粒,FⅡk和R复制子亦为主要的大的接合型复制子。研究者在肺炎克雷伯菌基因组中还发现了小型复制子,如Col复制子及F质粒的变种FⅡ、FⅠA和FⅠB也携带和转移耐药基因。还发现了其他类型的复制子如IncX3 N、HI1B和AC/2型复制子[13]。随着肺炎克雷伯菌全基因组测序数据的增加,许多不同长度和不相容性类型的质粒不断被发现[14]。某些肺炎克雷伯菌能允许质粒摄取和维持,导致质粒载量通常大于大肠埃希菌和其他革兰阴性菌[15]。例如,肺炎克雷伯菌分离株常携带4~6种不同质粒,多者能携带10种[16]。
除了耐药基因,肺炎克雷伯菌的毒力基因也通过质粒携带,这些质粒被称作毒力质粒。最具特征的毒力质粒是来自血清型K1的ST23型菌株NTUH-K2044中的质粒pK2044、来自血清型K2的ST86型菌株CG43中的质粒pLVPK,以及来自血清型K2的ST66型菌株Kp52.145中的质粒Kp52.145pⅡ(菌株也称为52145或B5055;质粒也称为pKP100)。普通肺炎克雷伯菌在获得这些毒力质粒后,毒力显著提升,主要是因为这些毒力质粒携带一些毒力相关基因,可使肺炎克雷伯菌具有毒力表型,如通过上调荚膜含量来赋予菌株黏液表型的rmpA基因、与铁载体合成相关的iuc基因以及编码重金属抗性的基因座[17]。有研究对从不同肝脓肿患者体内分离到的40株高毒力肺炎克雷伯菌进行分析,均检测到rmpA和iuc基因,且大多数菌株具有至少1个类似pK2044的质粒[18]。最近在一株K1 ST15型肺炎克雷伯菌基因中发现质粒pKp104014_1既具有IncFIBk型(与KpVP-1质粒同源)毒力质粒特征,又具有耐药质粒IncFIIK特征,既携带碳青霉烯类耐药基因又携带毒力基因[19]。这一兼具耐药性及毒力的接合型质粒的出现,无疑将加剧肺炎克雷伯菌的临床危害性。
2 前噬菌体噬菌体基因组是大小为2~300 kb的单链或双链DNA/RNA,通常编码噬菌体成分和特定复制酶基因,在某些情况下还编码细菌基因。由于基因组和形态特征不同,噬菌体之间存在广泛差异。根据其生命周期,可以分为裂解性噬菌体或温和性噬菌体。在裂解性周期中,噬菌体利用宿主系统复制并产生新的后代,然后从感染的细胞中释放出来。温和性噬菌体既可以通过裂解性周期繁殖,也可以通过将其基因组整合到宿主染色体中而进入溶原性周期[20]。在大多数溶原情况下,噬菌体将基因组整合到细菌染色体中并作为“前噬菌体”复制。但在少数情况下,噬菌体基因组可作为环状或线性质粒自主复制[7]。噬菌体DNA作为可移动DNA元件,是细菌之间横向基因转移的载体[21]。噬菌体可以通过普遍性或局限性转导进行基因交换,从而使噬菌体颗粒将遗传特性从供体细菌细胞传递到受体细胞。实际上,细菌的许多毒力因子是由噬菌体编码的[22]。噬菌体的这种作用不仅限于致病菌,非致病菌对环境的某些适应性也可能由噬菌体基因组介导[23]。
根据目前已完成测序的肺炎克雷伯菌基因组数据,发现92%的菌株中有前噬菌体基因组存在,但数量有较大差别,多者达20几个,平均为5.4个。不同肺炎克雷伯菌克隆群甚至同一克隆群的不同菌株中前噬菌体数目也有很大差异。高毒力肺炎克雷伯菌与其他类型肺炎克雷伯菌株相比前噬菌体的数量无显著差异,而耐药肺炎克雷伯菌基因组中噬菌体的数量明显高于其他类型,表明肺炎克雷伯菌特别是MDR肺炎克雷伯菌经常受到多种类型噬菌体的攻击[24]。此外,肺炎克雷伯菌染色体的基因组大小与前噬菌体的总基因组大小正相关,表明前噬菌体是肺炎克雷伯菌的主要辅助基因之一,对其基因组的可塑性具有重大贡献[25]。虽然肺炎克雷伯菌基因组中含有数量众多的前噬菌体,但对前噬菌体功能的研究相对较少,推测其在肺炎克雷伯菌基因组进化及功能适应中发挥了一定的作用。目前发现,前噬菌体溶原性转换可以使肺炎克雷伯菌获得blaSHV、fosA和oqxAB等耐药基因,同时前噬菌体FR2和AP3的溶原性转换可以对肺炎克雷伯菌荚膜进行修饰,从而发生表型改变。
3 ISIS是最小和最多的自主转座元件(transposable element,TE),位于转座子两端,在塑造宿主基因组中起重要作用。IS通常指一段短的DNA片段,仅编码转座有关的酶,可以整合到基因组导致细菌基因突变。IS可独立存在,也可成为转座子的一部分。典型的IS长度为0.7~2.5 kb,具有一个或两个开放阅读框(open reading frame,ORF),用于编码转座必需的转座酶。这些ORF遗传结构紧凑,占据IS整个长度,并终止于IS两端的反向重复序列(inverted repeat,IR)[26]。IR有两个功能区,Ⅰ区位于IS末端处,一般包括末端2~3个碱基,与转座起始时的链切割和链转移相关;Ⅱ区在IR内部,是转座酶结合区域。根据转座酶催化活性中心氨基酸序列的差异,可将转座酶分为DDE转座酶、DEDD转座酶、HUH转座酶和丝氨酸转座酶,这是IS分类的主要依据。大多数IS的转座酶为DDE转座酶[27]。IS两侧可能有因插入而生成的短正向重复序列(direct repeat,DR)。IS的DR长度不定,一般为2~14 bp,相同IS可能会有不同DR,有的IS也可能没有DR。IS转座时,可通过双链切割和单链切割两种机制完成。双链切割后IS以线性方式游离,然后插入新位点;而单链切割后,有些IS以环形方式游离,在原位点可能留下一个拷贝,也可能因二次切割整体环出。有些IS还可能通过其他更复杂的方式复制转座[28]。IS对细菌宿主的一个重要影响是其调节基因表达的能力,除了作为载体具有以复合转座子和转运蛋白的形式从一个复制子传递到另一个复制子以及中断基因的能力外,还可激活基因表达。目前,由于细菌对各种抗生素的耐药性增强,公共健康安全受到严重威胁,IS的这种能力受到了广泛关注[29]。
对耐药肺炎克雷伯菌ST258的基因组进行分析,发现其基因组中耐药基因blaKPC通常包含Tn4401。Tn4401长10 kb,两端有39 bp的IR,编码blaKPC基因,具有Tn3转座酶基因tnpA、Tn3解聚酶基因tnpR、两个重复序列及ISKpn6和ISKpn7序列,被广泛认为与blaKPC的获得及转移有关。在肺炎克雷伯菌质粒pKPN3、pKPN4和pKPN5中也发现了Tn4401序列,更进一步证实其与肺炎克雷伯菌基因组中遗传成分特别是耐药基因blaKPC的转移相关[30-31]。Tn4401a-blaKPC-2位于肺炎克雷伯菌基因组中前噬菌体下游,但是否与前噬菌体整合有关尚未知。Anita等发现了一个新亚型Tn4401h,其在ISKpn7与blaKPC之间上游非编码区有188 bp的缺失,与其他常见Tn4401亚型相比,这种缺失对碳青霉烯类抗生素的最低抑菌浓度(minimum inhibitory concentration,MIC)有影响,表明转座子亚型对肺炎克雷伯菌的耐药性有影响[32]。Tn1331也具有tnpA和tnpR基因,在IncN、IncI2和IncFIA型质粒基因组中均被发现。Tn1331常携带耐药基因aac(6')-Ib和aadA1,以及blaOXA-9和blaTEM-1基因[33]。ISEcp1亦常在耐药质粒基因中被发现,与blaCTX-M-15基因的插入及转移有关。随着研究不断深入,还发现肺炎克雷伯菌基因组中很多耐药基因的插入及获得均与IS序列如IS1T、IS1R、IS26、ISKpn26、ISKpn28和In27等有关,而Tn6556常与前噬菌体的插入相关。
4 结语肺炎克雷伯菌基因组具有高度变异性,可移动元件特别是质粒和噬菌体的获得或丢失是导致基因组多样性的主要原因。耐药基因和毒力基因会与不同特征性的可移动元件一起,通过可移动元件进行转移,这可能是导致肺炎克雷伯菌高耐药性和高毒力不断增强并流行的主要原因。近年来不断发现同时携带高毒力及高耐药性的接合型质粒,可在短时间内快速播散转移,使肺炎克雷伯菌快速获得高毒力和高耐药性,给临床治疗带来了巨大挑战。因此,研究及关注不同可移动元件与耐药基因及毒力基因的关系,并揭示其转移特征及机制,对防控高毒力及高耐药性肺炎克雷伯菌的出现及传播有重要意义。
| [1] |
Paczosa MK, Mecsas J. Klebsiella pneumoniae: going on the offense with a strong defense[J]. Microbiol Mol Biol Rev, 2016, 80(3): 629-661.
[DOI]
|
| [2] |
Domenico P, Salo RJ, Cross AS, Cunha BA. Polysaccharide capsule-mediated resistance to opsonophagocytosis in Klebsiella pneumoniae[J]. Infect Immun, 1994, 62(10): 4495-4499.
[DOI]
|
| [3] |
Papo N, Shai Y. A molecular mechanism for lipopolysaccharide protection of Gram-negative bacteria from antimicrobial peptides[J]. J Biol Chem, 2005, 280(11): 10378-10387.
[DOI]
|
| [4] |
Martin RM, Bachman MA. Colonization, infection, and the accessory genome of Klebsiella pneumoniae[J]. Front Cell Infect Microbiol, 2018, 8: 4.
[DOI]
|
| [5] |
Schroll C, Barken KB, Krogfelt KA, Struve C. Role of type 1 and type 3 fimbriae in Klebsiella pneumoniae biofilm formation[J]. BMC Microbiol, 2010, 10: 179.
[DOI]
|
| [6] |
Bassetti M, Righi E, Carnelutti A, Graziano E, Russo A. Multidrug-resistant Klebsiella pneumoniae: challenges for treatment, prevention and infection control[J]. Expert Rev Anti Infect Ther, 2018, 16(10): 749-761.
[DOI]
|
| [7] |
Holt KE, Wertheim H, Zadoks RN, Baker S, Whitehouse CA, Dance D, Jenney A, Connor TR, Hsu LY, Severin J, Brisse S, Cao H, Wilksch J, Gorrie C, Schultz MB, Edwards DJ, Nguyen KV, Nguyen TV, Dao TT, Mensink M, Minh VL, Nhu NT, Schultsz C, Kuntaman K, Newton PN, Moore CE, Strugnell RA, Thomson NR. Genomic analysis of diversity, population structure, virulence, and antimicrobial resistance in Klebsiella pneumoniae, an urgent threat to public health[J]. Proc Natl Acad Sci U S A, 2015, 112(27): E3574-E3581.
[DOI]
|
| [8] |
Frost LS, Leplae R, Summers AO, Toussaint A. Mobile genetic elements: the agents of open source evolution[J]. Nat Rev Microbiol, 2005, 3(9): 722-732.
[DOI]
|
| [9] |
Smillie C, Garcillán-Barcia MP, Francia MV, Rocha EP, de la Cruz F. Mobility of plasmids[J]. Microbiol Mol Biol Rev, 2010, 74(3): 434-452.
[DOI]
|
| [10] |
翁幸鐾, 糜祖煌. 可移动遗传元件: 耐药基因的载体[J]. 中国人兽共患病学报, 2013, 29(4): 389-397. [DOI]
|
| [11] |
De La Cruz F, Frost LS, Meyer RJ, Zechner EL. Conjugative DNA metabolism in Gram-negative bacteria[J]. FEMS Microbiol Rev, 2010, 34(1): 18-40.
[DOI]
|
| [12] |
De Toro M, Garcilláon-Barcia MP, De La Cruz F. Plasmid diversity and adaptation analyzed by massive sequencing of escherichia coli plasmids[J]. Microbiol Spectr, 2014, 2(6).
[DOI]
|
| [13] |
Wyres KL, Lam MMC, Holt KE. Population genomics of Klebsiella pneumoniae[J]. Nat Rev Microbiol, 2020, 18(6): 344-359.
[DOI]
|
| [14] |
Navon-Venezia S, Kondratyeva K, Carattoli A. Klebsiella pneumoniae: a major worldwide source and shuttle for antibiotic resistance[J]. FEMS Microbiol Rev, 2017, 41(3): 252-275.
[DOI]
|
| [15] |
Wyres KL, Holt KE. Klebsiella pneumoniae as a key trafficker of drug resistance genes from environmental to clinically important bacteria[J]. Curr Opin Microbiol, 2018, 45: 131-139.
[DOI]
|
| [16] |
Conlan S, Park M, Deming C, Thomas PJ, Young AC, Coleman H, Sison C, NISC Comparative Sequencing Program, Weingarten RA, Lau AF, Dekker JP, Palmore TN, Frank KM, Segre JA. Plasmid dynamics in KPC-positive Klebsiella pneumoniae during long-term patient colonization[J]. mBio, 2016, 7(3): e00742-16.
[DOI]
|
| [17] |
Lam MMC, Wyres KL, Judd LM, Wick RR, Jenney A, Brisse S, Holt KE. Tracking key virulence loci encoding aerobactin and salmochelin siderophore synthesis in Klebsiella pneumoniae[J]. Genome Med, 2018, 10(1): 77.
[DOI]
|
| [18] |
Russo TA, Marr CM. Hypervirulent Klebsiella pneumoniae[J]. Clin Microbiol Rev, 2019, 32(3): e00001-19.
[DOI]
|
| [19] |
Yang X, Wai-Chi Chan E, Zhang R, Chen S. A conjugative plasmid that augments virulence in Klebsiella pneumoniae[J]. Nat Microbiol, 2019, 4(12): 2039-2043.
[DOI]
|
| [20] |
Salmond GP, Fineran PC. A century of the phage: past, present and future[J]. Nat Rev Microbiol, 2015, 13(12): 777-786.
[DOI]
|
| [21] |
Canchaya C, Proux C, Fournous G, Bruttin A, Brüssow H. Prophage genomics[J]. Microbiol Mol Biol Rev, 2003, 67(2): 238-276.
[DOI]
|
| [22] |
Boyd EF, Brussow H. Common themes among bacteriophage-encoded virulence factors and diversity among the bacteriophages involved[J]. Trends Microbiol, 2002, 10(11): 521-529.
[DOI]
|
| [23] |
Brüssow H, Hendrix RW. Phage genomics: small is beautiful[J]. Cell, 2002, 108(1): 13-16.
[DOI]
|
| [24] |
Wyres KL, Wick RR, Judd LM, Froumine R, Tokolyi A, Gorrie CL, Lam MMC, Duchêne S, Jenney A, Holt KE. Distinct evolutionary dynamics of horizontal gene transfer in drug resistant and virulent clones of Klebsiella pneumoniae[J]. PLoS Genet, 2019, 15(4): e1008114.
[DOI]
|
| [25] |
Shen J, Zhou J, Xu Y, Xiu Z. Prophages contribute to genome plasticity of Klebsiella pneumoniae and may involve the chromosomal integration of ARGs in CG258[J]. Genomics, 2020, 112(1): 998-1010.
[DOI]
|
| [26] |
Siguier P, Gourbeyre E, Chandler M. Bacterial insertion sequences: their genomic impact and diversity[J]. FEMS Microbiol Rev, 2014, 38(5): 865-891.
[DOI]
|
| [27] |
张孟思, 朱德康, 汪铭书. 细菌插入序列中的转座酶和转座机制[J]. 中国生物化学与分子生物学报, 2018, 34(10): 1057-1064. [CNKI]
|
| [28] |
Krüger NJ, Stingl K. Two steps away from novelty--principles of bacterial DNA uptake[J]. Mol Microbiol, 2011, 80(4): 860-867.
[DOI]
|
| [29] |
Siguier P, Gourbeyre E, Varani A, Ton-Hoang B, Chandler M. Everyman's guide to bacterial insertion sequences[J]. Microbiol Spectr, 2015, 3(2): MDNA3-0030-2014.
[DOI]
|
| [30] |
Naas T, Cuzon G, Villegas MV, Lartigue MF, Quinn JP, Nordmann P. Genetic structures at the origin of acquisition of the beta-lactamase bla KPC gene[J]. Antimicrob Agents and Chemother, 2008, 52(4): 1257-1263.
[DOI]
|
| [31] |
Chen L, Mathema B, Chavda KD, Deleo FR, Bonomo RA, Kreiswirth BN. Carbapenemase-producing Klebsiella pneumoniae: molecular and genetic decoding[J]. Trends Microbiol, 2014, 22(12): 686-696.
[DOI]
|
| [32] |
Cheruvanky A, Stoesser N, Sheppard AE, Crook DW, Hoffman PS, Weddle E, Carroll J, Sifri CD, Chai W, Barry K, Ramakrishnan G, Mathers AJ. Enhanced Klebsiella pneumoniae carbapenemase expression from a novel Tn4401 deletion[J]. Antimicrob Agents Chemother, 2017, 61(6): e00025-17.
[DOI]
|
| [33] |
Sarno R, Mcgillivary G, Sherratt DJ, Actis LA, Tolmasky ME. Complete nucleotide sequence of Klebsiella pneumoniae multiresistance plasmid pJHCMW1[J]. Antimicrob Agents Chemother, 2002, 46(11): 3422-3427.
[DOI]
|
 2021, Vol. 16
2021, Vol. 16


