2. 中国科学院苏州生物医学工程技术研究所,江苏 苏州 215163
2. Suzhou Institute of Biomedical Engineering and Technology, Chinese Academy of Sciences, Suzhou 215163, Jiangsu Province, China
目前,随着抗生素的广泛和大量使用,耐药菌不断出现,人类面临着可选择的抗生素种类越来越少而治疗成本和难度日益增加的困局。因此,快速准确的病原体检测和诊断至关重要,既能降低耐药菌感染的死亡率,也能有效抑制耐药菌的传播。
常规的临床病原微生物实验室检测主要通过生物化学反应或飞行时间质谱对分离纯化后的纯培养病原菌进行鉴定,采用抗菌药物敏感性试验(antimicrobial susceptibility test,AST)在鉴定的基础上观察药物作用后病原菌的生长变化[1]。但培养、纯化和飞行时间质谱鉴定通常需2 d以上,AST通常需1~3 d,“从样本到报告”往往需3~7 d,极大地降低了检测效率[2]。同时,还存在无法区分生物化学反应相似的病原菌、数据库中的已知病原菌种类有限以及难培养微生物难以鉴定等局限,为克服以上问题,近年来多种免培养的微生物快速检测技术得到了发展。
目前,免培养的微生物检测方法主要为分子遗传学鉴定法,如聚合酶链反应(polymerase chain reaction,PCR)、基因测序、核酸质谱法、生物芯片等,主要针对核酸等遗传物质。虽然这些方法各有优势,但存在易受外界条件干扰、检测步骤复杂等弊端,操作过程中还需操作技能娴熟、工作经验丰富且理论知识扎实的操作人员以及昂贵且精密的仪器[3]。以PCR为例,其主要原理为扩增待测微生物的DNA片段,然而随着DNA的扩增,其在获得极大检测灵敏度的同时也对检测中的错误有放大作用,因此操作稍有不当或实验条件微扰或核酸提取中的随机误差,均可能造成假阳性或假阴性结果[4]。PCR针对不同的病原菌须设计不同的引物,也增加了检测的难度和成本。此外,耐药基因的分子诊断只能针对已知的耐药机制及对应基因,且阳性耐药基因不能与耐药表型完全对应。因此,亟须建立可普遍用于临床的基于表型的病原菌诊断方法。
近年来,拉曼光谱技术不断发展,开始在病原微生物检测领域崭露头角。拉曼光谱技术作为一种典型的分析化学方法,具有快速、高灵敏度、高特异度、无破坏性、不受水样干扰等特点[5]。与常规检测相比,拉曼光谱技术无须培养病原菌,从鉴定菌种到获得AST结果仅需几小时,在医学检验和诊断方面有着巨大的潜力。
1 单细胞拉曼光谱技术原理1928年,印度科学家Raman发现了拉曼散射效应,其对应产生的光谱称为拉曼光谱[6]。当物质分子被入射光照射时,入射光子与分子发生相互作用,散射出不同频率的光。大部分散射光频率与入射光频率相同,称为“瑞利散射”,极少数散射光频率与入射光频率不同,称为“拉曼散射”[7]。拉曼光谱是基于光与物质内化学键的相互作用而产生,表明了分子中特定键的振动和转动,因此可根据散射光的特点对物质进行定性和定量分析。
将拉曼光谱与显微技术结合,能提供0.5~1 μm空间分辨率的单个微生物细胞内化学结构信息,其中包含核酸、蛋白质、糖类、脂质和色素(如类胡萝卜素)等信息[8-10]。每个细胞都有其特定的化学组成,因此这些信息被称为可鉴定细胞的独特的“拉曼指纹图谱”[11](见图 1)。由于拉曼光谱信号较弱,研究人员发明了多种拉曼增强技术,如共振拉曼散射(resonance Raman scattering,RRS)、相干反斯托克斯拉曼散射(coherent anti-Stokes Raman scattering,CARS)、受激拉曼散射(stimulated Raman scattering,SRS)和表面增强拉曼散射(surface-enhanced Raman scattering,SERS)。这些技术的出现使得单细胞拉曼光谱技术更加成熟,在感染检测领域具有更大的潜力。
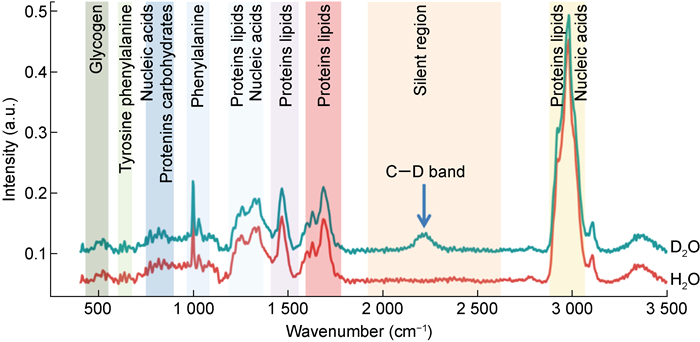
|
| 图 1 典型大肠埃希菌的拉曼图谱 Fig. 1 Raman spectra of Escherichia coli strains |
单细胞拉曼光谱技术可根据特异性“指纹图谱”,对不同病原菌菌种甚至亚种进行区分和鉴定,且具有非破坏性,细菌样本还可用于其他研究分析(见表 1)。与利用细菌“蛋白质指纹”进行鉴定的飞行时间质谱技术相比,单细胞拉曼光谱技术对细菌的数量要求更低,且不要求样品是纯菌,因此不需要耗时的细菌培养步骤,可直接进行细菌鉴定。
| Spectral type |
Type of bacteria tested | Detection levels | Accuracy | Detection time |
Data processing method |
Reference |
| Raman | Salmonella typhimurium (S. typhimurium), Escherichia coli (E. coli), Staphylococcus aureus (S. aureus), Listeria innocua | Species | 99% | — | PCA | [15] |
| Raman | E. coli, Enterococcus faecalis (E. faecalis) E. coli and E. faecalis in urine samples |
Species | 35 min | PCA LDA |
[21] | |
| Raman | E. coli, Bacillus subtilis, Staphylococcus epidermidis (S. epidermidis), Bacillus thuringiensis, Micrococcus luteus, Serratia marcescens, etc. | Species | 87% | — | SVM | [24] |
| Raman | E. coli, Pseudomonas aeruginosa (P. aeruginosa), Monilia albican and 11 other kinds of fungi and bacteria | Species | (95.64±546)% | — | CNN LDA |
[18] |
| Raman | E. coli, P. aeruginosa, Klebsiella pneumoniae (K. pneumoniae), S. aureus, etc. | Species | Gram-positive bacteria: 98.8% Gram-negative bacteria: 94.3% |
— | PCA LDA |
[14] |
| Raman | 11 types of bacteria that cause UTI: E. faecalis, 2 strains of E. coli, S. aureus, etc. | Species and strain | 92% | <2 h | SVM | [25] |
| Raman | 30 types of common fungi and bacteria: Methicillin-resistant and methicillin-susceptible S. aureus | Species and strain | 82% 98% |
Few hours | PCA SVM LR CNN |
[26] |
| Raman | Burkholderia mallei, Burkholderia pseudomallei, other species of Burkholderia | Species | 90% | — | PCA SVM |
[17] |
| Raman | 26 types of Mycobacterium | Strain | 94% | — | SVM | [27] |
| Raman | Elizabethkingia spp. | Strain | LDA | [19] | ||
| SERS | E. faecalis, Streptococcus pyogenes, K. pneumoniae, Acinetobacter baumannii | Species | — | — | [13] | |
| SERS | 7 types of bacteria that cause UTI: E. coli, E. faecalis, S. aureus, Staphylococcus saprophyticus, K. pneumoniae, etc. | Species | — | 1 h | PCA | [16] |
| SERS | Bacteria in urine samples: E. coli, K. pneumoniae, E. faecalis | Species | Sensitivity: 95.8%, Specificity: 99.3% |
<1 h | PLS-DA | [22] |
| SERS | 9 strains of E. coli | Strain | 92% | — | SVM | [20] |
| SERS | 3 strains of E. coli, S. epidermidis | Species and strain | — | 10 min | HCA | [28] |
| SERS | Bacteria in blood samples: vancomycin-susceptible and vancomycin-resistant E. faecalis, E. coli, Lactobacillus plantarum | Species and strain | — | — | SNIP-based self-development program | [23] [29] |
| SERS | E. coli, Proteus mirabilis Pathogenic and non-pathogenic Escherichia coli | Species and strain | — | Few hours |
PCA | [30] |
| SERS | S. aureus, Legionella pneumophila Methicillin-resistant and methicillin-susceptible S. aureus |
Species and strain | 97.8% | — | PCA HCA k-NN |
[31] |
| SERS | E. coli, Klebsiella oxytoca, K. pneumoniae, etc. Multiple strains of E. coli |
Species and strain | — | — | DFA HCA |
[32] |
| SERS: Surface-enhanced Raman scattering; UTI: Urinary tract infection; PCA: Principal component analysis; LDA: Linear discriminant analysis; SVM: Support vector machine; CNN: Convolutional neural networks; LR: Logistic regression; PLS-DA: Partial least squares discriminant analysis; HCA: Hierarchical cluster analysis; SNIP: Scale normalization for image pyramids; k-NN: k-nearest neighbor; DFA: Deterministic finite automaton; “—” indicates that the study did not show relevant data. | ||||||
在病原菌感染的诊断过程中,对革兰细菌进行分类至关重要,可为后续诊断及抗生素的选择提供参考。传统的革兰染色鉴定须进行细菌培养,但细菌形态和过度脱色等问题容易导致误判。Paret等[12]应用类胡萝卜素的共振拉曼带来区分几种革兰阴性菌和阳性菌。Prucek等[13]采用表面增强拉曼光谱技术区分了2种革兰阴性菌和2种革兰阳性菌。Oliveira等[10]采用代表肽聚糖的拉曼特征峰来进行革兰菌分类,发现革兰阳性菌在540 cm-1和1 380 cm-1的峰值明显高于革兰阴性菌,这2个峰值代表了N-乙酰葡萄糖胺(N-acetylglucosamine,NAG)和N-乙酰胞壁酸(N-acetylmuramic acid,NAMA),二者是肽聚糖的主要成分。Yi等[14]结合自发拉曼光谱与机器学习模型,建立了一个包含4株革兰阴性菌和4株革兰阳性菌的拉曼谱库,分类准确率达到100%,对真实血液和尿液标本中未被数据库覆盖的菌种也实现了准确的革兰菌分类。
2.2 菌种和菌株水平的识别Sundaram等[15]采用表面增强拉曼光谱技术对2种革兰阳性菌和2种革兰阴性菌进行鉴别,发现1 200~1 700 cm-1为识别4种细菌的指纹区域,准确率为97%。Avci等[16]应用表面增强拉曼光谱技术对引起尿路感染的大肠埃希菌(Escherichia coli,E. coli)、粪肠球菌(Enterococcus faecalis,E. faecalis)、金黄色葡萄球菌(Staphylococcus aureus,S. aureus)、腐生葡萄球菌(Staphylococcus saprophyticus,S. saprophyticus)、肺炎克雷伯菌(Klebsiella pneumoniae,K. pneumoniae)等7种病原菌进行识别,在1 h内成功获得结果。Moawad等[17]结合主成分分析(principal component analysis,PCA)与支持向量机(support vector machine,SVM)方法,成功区分了难区分的鼻疽伯克霍尔德菌(Burkholderia mallei,B. mallei)和类鼻疽伯克霍尔德菌(Burkholderia pseudomallei,B. pseudomallei),识别率高于90%。Lu等[18]采用卷积神经网络(convolutional neural networks,CNN)对14种细菌和真菌进行了鉴定。
同种细菌之间光谱差异很小,因此菌株水平的识别难度更高。机器学习技术的不断发展,使其对光谱信息有了更准确的分析。Yuan等[19]对拉曼光谱进行线性判别分析(linear discriminant analysis,LDA),成功区分了一类导致脑膜炎的伊丽莎白金菌(Elizabethkingia spp.)与其他常见革兰阴性菌,且实现了不同来源伊丽莎白金菌的溯源。Walter等[20]采用SVM算法处理细菌的表面增强拉曼光谱数据,成功区分了9种大肠埃希菌,准确率为92%,实现了大肠埃希菌在菌株水平的识别。
2.3 临床样本中的细菌识别直接识别体液中的细菌是拉曼光谱技术的一个重大进步,加快了其在临床应用的步伐。近年来,大量研究者尝试利用拉曼光谱技术对临床样本进行分析并开展临床转化研究。Schröder等[21]将拉曼光谱技术与双电泳芯片结合,从尿液样本中捕获并采集细菌的拉曼光谱,成功识别了尿液中的大肠埃希菌和粪肠球菌。Premasiri等[22]采用表面增强拉曼光谱技术直接检测尿液中的细菌,不到1 h即成功识别了尿液中的大肠埃希菌、肺炎克雷伯菌、粪肠球菌等,敏感度为95.8%,特异度为99.3%。Liu等[23]在血液中用万古霉素涂层银纳米颗粒列阵捕获细菌,进而分离细菌和血细胞等其他杂质,使细菌捕获量增加了1 000倍,对人类血液中细菌实现了免培养的分析和识别。表 1对单细胞拉曼光谱技术在病原菌鉴定中的研究进展进行了总结。
3 单细胞拉曼光谱技术在AST中的应用AST可测定抗生素在体外对病原菌的抑制效果,并确定抗生素的最低抑菌浓度(minimum inhibitory concentration,MIC)。目前,基于单细胞拉曼光谱技术的AST主要有2种,即非标记拉曼光谱特征峰识别法和重水标记拉曼技术(Raman-deuterium isotope probing,Raman-DIP)。
3.1 非标记拉曼光谱特征峰识别法抗生素作用于细菌时,会引起易感菌株内部发生一系列生物化学反应。因此,可通过观察细菌某些特征峰峰值的变化,来判断抗生素是否起作用及其疗效,还可区分易感与耐药菌株并确定MIC值。Uysal Ciloglu等[31]采用PCA、分级聚类算法(hierarchical cluster analysis,HCA)、k最近邻(k-nearest neighbor,KNN)算法等方法,分析了光谱极其相似的甲氧西林敏感金黄色葡萄球菌与耐甲氧西林金黄色葡萄球菌,准确率达到97.8%。Ho等[26]采用深度学习方式中的CNN算法,成功区分了甲氧西林敏感金黄色葡萄球菌与耐甲氧西林金黄色葡萄球菌。Liu等[33]发现,在抗生素作用期间,金黄色葡萄球菌和大肠埃希菌位于730 cm-1和1 095 cm-1的代表细胞壁成分的表面增强拉曼光谱特征峰,可用于区分易感与耐药菌株。Liu等[34]发现,金黄色葡萄球菌等在甲氧西林等抗生素的作用下,表面增强拉曼光谱中的特定生物标记强度在2 h内明显下降,以此可对金黄色葡萄球菌和野生大肠埃希菌进行AST并确定MIC值,所得结果与“金标准”方法一致。Kirchhoff等[35]检测从血液感染者血培养标本中分离的大肠埃希菌,发现代表C—H振动的1 458 cm-1光谱峰峰值随着环丙沙星浓度的增高而增高,而代表核酸的1 485 cm-1峰值随之降低,两个振动带的强度比值反映了环丙沙星的抑菌效果,进而可确定其MIC值。该方法不到2 h即完成了AST并确定了MIC值,所得结果与标准方法一致。以上通过观察光谱特征峰变化的方法须针对不同种类的细菌和抗生素进行逐案评估,因此其在AST中的通用性尚待进一步评估。
3.2 Raman-DIPBerry等[36]首次报道了在含有重水(D2O)的培养条件下培养大肠埃希菌,仅20 min后于其单细胞拉曼光谱中检测到碳-氘(C—D)峰(见图 1),由此认为C—D峰的出现完全是由细菌代谢引起的。细菌在含有重水的培养基中培养时,氘原子会参与细胞脂质合成过程,形成C—D键替换原有的C—H键,新形成的C—D键的特征峰出现在2 000~2 300 cm-1区域,而该区域通常不存在其他标志性峰,因此被称为静默区。基于此项研究,Song等学者提出了Raman-DIP方法,以C—D峰强度代表抗生素作用下的细菌代谢活性,并依此识别耐药菌,从而判断抗生素的敏感性[37-38]。由于细菌代谢不以细菌分裂增殖为先决条件,且单细胞拉曼光谱技术可在单细胞水平分析样品的拉曼散射特征,因此可进行免培养的AST。Song等[37]应用Raman-DIP在数小时内即成功识别了未培养的环境样品中的抗生素敏感菌和耐药菌。Tao等[38]将其用于识别口腔微生物变形链球菌(Streptococcus mutans,S. mutans)。Ueno等[39]用该方法发现,耻垢分枝杆菌(M. smegmatis)耐药菌在利福平的作用下表现出一定的代谢活性。Hong等[40]和Zhang等[41]将Raman-DIP与受激拉曼散射成像结合,通过检测活菌中的葡萄糖代谢来进行AST,在一个细胞周期内就确定了耐万古霉素粪肠球菌的MIC值,并用于8种细菌和14种抗生素的检测。Yi等[14]发现,将抗生素与重水在不同时间点加入菌液或标本,可减小氨苄西林等抗生素对细菌生长和代谢活性抑制效果的差异,提高AST的准确率,基于此建立了快速拉曼AST(fast Raman-assisted antibiotic susceptibility test, FRAST)方法,此法适用于38种抗生素。他们采用FRAST方法检测了9份泌尿系统感染患者的尿液样本和3份败血症患者的血液样本,结果与常规自动化药敏结果一致,但将检测时间分别缩短至3 h和21 h,进一步证实了其在临床应用的可行性。Yuan等[19]在对伊丽莎白属细菌的快速药敏研究中进一步发现,FRAST方法可以对细菌浓度达到106CFU/mL以上的血液标本直接进行药敏检测。
4 结语拉曼光谱技术在生物领域应用广泛,尤其在病原微生物检测方面优点突出,可直接对细菌感染临床样品进行非接触快速检测。相较于传统方法,拉曼光谱技术具有无须制备样品、无须进行微生物培养的优势,是微生物感染的重要诊断方法。单细胞拉曼光谱技术将临床标准药敏检测时间从48 h缩短至2.5 h内,为临床快速诊断与精准选择抗生素提供了依据,但其临床应用仍面临诸多挑战。首先,拉曼光谱技术与样品前处理的结合至关重要。为了更高效地收集高密度的细菌,或分离出单细胞水平的细菌,其可结合微流控器件等方式,实现高通量、智能化的细菌细胞收集、分离和分选。其次,目前商业化飞行时间质谱鉴定系统具有包含上万株菌株的数据库,与其相比,利用拉曼光谱技术建立病原微生物物种的拉曼统一数据库及其校准方法已迫在眉睫。高质量的数据库可为后续数据处理提供丰富且准确的信息,进而提高辨识菌种的精确度。有些实验室已做出努力,建立了适合自己研究方向的小型数据库,如鉴定多种分枝杆菌的数据库[23]、快速检测尿路感染(urinary tract infection,UTI)细菌的数据库[27]等。然而,这些小型数据库均是针对不同分析目的而特别制定的,且不同实验室拉曼光谱设备不同、参考样本来源不同等因素,导致各谱库之间有细微偏差。Dörfer等[42]应用化学计量技术微调校准参数和例程,进而消除实验记录的拉曼光谱中的设置差异,为拉曼谱库的校准做出了贡献,也进一步为标准数据库的建立提供了实践基础。
综上所述,拉曼光谱技术在感染领域蕴含着巨大潜力。尽管目前其在临床应用方面仍有较大的优化空间,但相信在不久的将来,随着操作进一步规范化、器材更加便捷和智能化、数据处理技术达到更高的精确度,以及全病原微生物物种标准数据库的建立,于临床广泛应用快速、灵敏、自动化、智能化的拉曼光谱技术将成为可能,这项技术将为临床感染领域带来变革。
| [1] |
张景皓, 方毅, 张艳梅, 赵虎. MALDI-TOF MS结合短期培养法快速检测中段尿样本中的病原菌[J]. 检验医学, 2017, 32(4): 326-330. [DOI]
|
| [2] |
顾兵, 李永军. 基质辅助激光解吸电离飞行时间质谱技术在临床微生物鉴定中的应用及价值[J]. 临床检验杂志, 2013, 31(11): 814-817. [DOI]
|
| [3] |
刘玮, 路颖, 于庆潭. 分子生物学技术在生物战病原微生物检测中的应用[J]. 国际检验医学杂志, 2021, 42(23): 2927-2930. [DOI]
|
| [4] |
白菊红, 康建平, 张星灿, 华苗苗, 刘建, 杨健, 钟雪婷. 多重PCR技术在病原微生物检测中的应用[J]. 食品工业科技, 2019, 40(7): 322-325. [DOI]
|
| [5] |
李梦雅, 夏珂, 和海妍, 谢凤欣, 府伟灵. 拉曼光谱技术在检验医学中的研究进展及应用[J]. 中华检验医学杂志, 2021, 44(1): 70-74. [DOI]
|
| [6] |
Raman CV, Krishnan KS. A new type of secondary radiation[J]. Nature, 1928(121): 501-502.
[DOI]
|
| [7] |
Petry R, Schmitt M, Popp J. Raman spectroscopy—A prospective tool in the life sciences[J]. Chemphyschem, 2003, 4(1): 14-30.
[DOI]
|
| [8] |
许永建, 罗荣辉, 郭茂田, 吴俊富, 李明玉. 共聚焦显微拉曼光谱的应用和进展[J]. 激光杂志, 2007, 28(2): 13-14. [DOI]
|
| [9] |
Downes A, Elfick A. Raman spectroscopy and related techniques in biomedicine[J]. Sensors, 2010, 10(3): 1871-1889.
[DOI]
|
| [10] |
de Siqueira E Oliveira FS, da Silva AM, Pacheco MTT, Giana HE, Silveira L Jr. Biochemical characterization of pathogenic bacterial species using Raman spectroscopy and discrimination model based on selected spectral features[J]. Lasers Med Sci, 2020, 36(1): 1-14.
[DOI]
|
| [11] |
Stöckel S, Kirchhoff J, Neugebauer U, Rösch P, Popp J. The application of Raman spectroscopy for the detection and identification of microorganisms[J]. J Raman Spectrosc, 2016, 47(1): 89-109.
[DOI]
|
| [12] |
Paret ML, Sharma SK, Green LM, Alvarez AM. Biochemical characterization of Gram-positive and Gram-negative plant-associated bacteria with micro-Raman spectroscopy[J]. Appl Spectrosc, 2010, 64(4): 433-441.
[DOI]
|
| [13] |
Prucek R, Ranc V, Kvítek L, Panáček A, Zbořil R, Kolář M. Reproducible discrimination between Gram-positive and Gram-negative bacteria using surface enhanced Raman spectroscopy with infrared excitation[J]. Analyst, 2012, 137(12): 2866-2870.
[DOI]
|
| [14] |
Yi X, Song Y, Xu X, Peng D, Wang J, Qie X, Lin K, Yu M, Ge M, Wang Y, Zhang D, Yang Q, Wang M, Huang WE. Development of a fast Raman-assisted antibiotic susceptibility test (FRAST) for the Antibiotic Resistance Analysis of Clinical Urine and Blood Samples[J]. Anal Chem, 2021, 93(12): 5098-5106.
[DOI]
|
| [15] |
Sundaram J, Park B, Kwon Y, Lawrence KC. Surface enhanced Raman scattering (SERS) with biopolymer encapsulated silver nanosubstrates for rapid detection of foodborne pathogens[J]. Int J Food Microbiol, 2013, 167(1): 67-73.
[DOI]
|
| [16] |
Avci E, Kaya NS, Ucankus G, Culha M. Discrimination of urinary tract infection pathogens by means of their growth profiles using surface enhanced Raman scattering[J]. Anal Bioanal Chem, 2015, 407(27): 8233-8241.
[DOI]
|
| [17] |
Moawad AA, Silge A, Bocklitz T, Fischer K, Rösch P, Roesler U, Elschner MC, Popp J, Neubauer H. A machine learning-based Raman spectroscopic assay for the identification of Burkholderia mallei and related species[J]. Molecules, 2019, 24(24): 4516.
[DOI]
|
| [18] |
Lu W, Chen X, Wang L, Li HF, Fu YV. Combination of an artificial intelligence approach and laser tweezers Raman spectroscopy for microbial identification[J]. Anal Chem, 2020, 92(9): 6288-6296.
[DOI]
|
| [19] |
Yuan S, Chen Y, Lin K, Zou L, Lu X, He N, Liu R, Zhang S, Shen D, Song Z, Tong C, Song Y, Zhang W, Chen L, Sun G. Single cell Raman-deuterium isotope probing for drug resistance of Elizabethkingia spp.[J]. Front Microbiol, 2022, 13: 876925.
[DOI]
|
| [20] |
Walter A, März A, Schumacher W, Rösch P, Popp J. Towards a fast, high specific and reliable discrimination of bacteria on strain level by means of SERS in a microfluidic device[J]. Lab Chip, 2011, 11(6): 1013-1021.
[DOI]
|
| [21] |
Schröder UC, Ramoji A, Glaser U, Sachse S, Leiterer C. Combined dielectrophoresis-Raman setup for the classification of pathogens recovered from the urinary tract[J]. Anal Chem, 2013, 85(22): 10717-10724.
[DOI]
|
| [22] |
Premasiri WR, Chen Y, Williamson PM, Bandarage DC, Pyles C, Ziegler LD. Rapid urinary tract infection diagnostics by surface-enhanced Raman spectroscopy (SERS): identification and antibiotic susceptibilities[J]. Anal Bioanal Chem, 2017, 409(11): 3043-3054.
[DOI]
|
| [23] |
Liu TY, Tsai KT, Wang HH, Chen Y, Chen YH, Chao YC, Chang HH, Lin CH, Wang JK, Wang YL. Functionalized arrays of Raman-enhancing nanoparticles for capture and culture-free analysis of bacteria in human blood[J]. Nat Commun, 2011, 2(1): 538.
[DOI]
|
| [24] |
Strola SA, Baritaux JC, Schultz E, Simon AC, Allier C, Espagnon I, Jary D, Dinten JM. Single bacteria identification by Raman spectroscopy[J]. J Biomed Opt, 2014, 19(11): 111610.
[DOI]
|
| [25] |
Kloss S, Kampe B, Sachse S, Rösch P, Straube E, Pfister W, Kiehntopf M, Popp J. Culture independent Raman spectroscopic identification of urinary tract infection pathogens: a proof of principle study[J]. Anal Chem, 2013, 85(20): 9610-9616.
[DOI]
|
| [26] |
Ho CS, Jean N, Hogan CA, Blackmon L, Jeffrey SS, Holodniy M, Banaei N, Saleh AAE, Ermon S, Dionne J. Rapid identification of pathogenic bacteria using Raman spectroscopy and deep learning[J]. Nat Commun, 2019, 10(1): 4927.
[DOI]
|
| [27] |
Stöckel S, Meisel S, Lorenz B, Kloß S, Henk S, Dees S, Richter E, Andres S, Merker M, Labugger I, Rösch P, Popp J. Raman spectroscopic identification of Mycobacterium tuberculosis[J]. J Biophotonics, 2017, 10(5): 727-734.
[DOI]
|
| [28] |
Zhou H, Yang D, Ivleva NP, Mircescu NE, Niessner R, Haisch C. SERS detection of bacteria in water by in situ coating with Ag nanoparticles[J]. Anal Chem, 2014, 86(3): 1525-1533.
[DOI]
|
| [29] |
Yang G, Dai J, Liu X, Chen M, Wu X. Multiple constrained reweighted penalized least squares for spectral baseline correction[J]. Appl Spectrosc, 2020, 74(12): 1443-1451.
[DOI]
|
| [30] |
Mircescu NE, Zhou H, Leopold N, Chiş V, Ivleva NP, Niessner R, Wieser A, Haisch C. Towards a receptor-free immobilization and SERS detection of urinary tract infections causative pathogens[J]. Anal Bioanal Chem, 2014, 406(13): 3051-3058.
[DOI]
|
| [31] |
Uysal Ciloglu F, Saridag AM, Kilic IH, Tokmakci M, Kahraman M, Aydin O. Identification of methicillin-resistant Staphylococcus aureus bacteria using surface-enhanced Raman spectroscopy and machine learning techniques[J]. Analyst, 2020, 145(23): 7559-7570.
[DOI]
|
| [32] |
Jarvis RM, Goodacre R. Discrimination of bacteria using surface-enhanced Raman spectroscopy[J]. Anal Chem, 2004, 76(1): 40-47.
[DOI]
|
| [33] |
Liu TT, Lin YH, Hung CS, Liu TJ, Chen Y, Huang YC, Tsai TH, Wang HH, Wang DW, Wang JK, Wang YL, Lin CH. A high speed detection platform based on surface-enhanced Raman scattering for monitoring antibiotic-induced chemical changes in bacteria cell wall[J]. PLoS One, 2009, 4(5): e5470.
[DOI]
|
| [34] |
Liu CY, Han YY, Shih PH, Lian WN, Wang HH, Lin CH, Hsueh PR, Wang JK, Wang YL. Rapid bacterial antibiotic susceptibility test based on simple surface-enhanced Raman spectroscopic biomarkers[J]. Sci Rep, 2016, 6(1): 23375.
[DOI]
|
| [35] |
Kirchhoff J, Glaser U, Bohnert JA, Pletz MW, Popp J, Neugebauer U. Simple ciprofloxacin resistance test and determination of minimal inhibitory concentration within 2 h using Raman spectroscopy[J]. Anal Chem, 2018, 90(3): 1811-1818.
[DOI]
|
| [36] |
Berry D, Mader E, Lee TK, Woebken D, Wang Y, Zhu D, Palatinszky M, Schintlmeister A, Schmid MC, Hanson BT, Shterzer N, Mizrahi I, Rauch I, Decker T, Bocklitz T, Popp J, Gibson CM, Fowler PW, Huang WE, Wagner M. Tracking heavy water (D2O) incorporation for identifying and sorting active microbial cells[J]. Proc Natl Acad Sci USA, 2015, 112(2): E194-E203.
[DOI]
|
| [37] |
Song Y, Cui L, López JÁS, Xu J, Zhu YG, Thompson LP, Huang W. Raman-deuterium isotope probing for in-situ identification of antimicrobial resistant bacteria in Thames river[J]. Sci Rep, 2017, 7(1): 16648.
[DOI]
|
| [38] |
Tao Y, Wang Y, Huang S, Zhu P, Huang WE, Ling J, Xu J. Metabolic-activity-based assessment of antimicrobial effects by D2O-labeled single-cell Raman microspectroscopy[J]. Anal Chem, 2017, 89(7): 4108-4115.
[DOI]
|
| [39] |
Ueno H, Kato Y, Tabata KV, Noji H. Revealing the metabolic activity of persisters in mycobacteria by single-cell D2O Raman imaging spectroscopy[J]. Anal Chem, 2019, 91(23): 15171-15178.
[DOI]
|
| [40] |
Hong W, Karanja CW, Abutaleb NS, Younis W, Zhang X, Seleem MN, Cheng JX. Antibiotic susceptibility determination within one cell cycle at single-bacterium level by stimulated Raman metabolic imaging[J]. Anal Chem, 2018, 90(6): 3737-3743.
[DOI]
|
| [41] |
Zhang M, Hong W, Abutaleb NS, Li J, Dong PT, Zong C, Wang P, Seleem MN, Cheng JX. Rapid determination of antimicrobial susceptibility by stimulated Raman scattering imaging of D2O metabolic incorporation in a single bacterium[J]. Adv Sci (Weinh), 2020, 7(19): 2001452.
[DOI]
|
| [42] |
Dörfer T, Bocklitz T, Tarcea N, Schmitt M, Popp J. Checking and improving calibration of Raman spectra using chemometric approaches[J]. Z Phys Chem, 2011, 225(6/7): 753-764.
[DOI]
|
 2022, Vol. 17
2022, Vol. 17



