B淋巴细胞免疫应答对于机体控制病毒感染至关重要,且在不同感染情境下,B细胞应答呈现出多样化的特征[1-3]。在急性感染中,B细胞应答快速而强烈,以确保及时清除病原体;但在慢性感染时,B细胞应答可能不完全或受损,无法有效清除病毒感染[4-8]。然而,由于缺乏理想的实验模型,目前尚未明确不同感染状况下B细胞出现应答差异的具体机制。因此,亟须建立适宜的急性与慢性病毒感染模型,为系统性比较B细胞在不同感染情况下的应答规律研究奠定基础。
淋巴细胞脉络丛脑膜炎病毒(lymphocytic choriomeningitis virus,LCMV)是一种带包膜的单股负链RNA病毒,其基因组包含S和L 2个节段,分别编码糖蛋白(glycoprotein,GP)和核衣壳蛋白(nucleoprotein,NP),以及RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase or L protein,Pol或L蛋白)和小锌指蛋白(small matrix protein,Z蛋白)[9]。LCMV以小鼠等啮齿类动物为自然宿主,是病毒学及免疫学相关领域研究中的经典工具,通过调整病毒株、剂量和感染途径LCMV可具备不同感染特点,从而可满足不同研究的要求[10]。
研究中常见的LCMV毒株主要来源于Armstrong、Traub和WE等3个亲本株,不同亲本株来源的病毒之间遗传差异显著且致病性和毒力各异[11-12]。从感染LCMV-Armstrong的小鼠脾脏中进一步分离出的变异株LCMV-Clone13与原始株相比,它们的病毒蛋白仅在Pol和GP处存在3个氨基酸的差异[13-14]。但Armstrong和Clone13在Pol之间的差异将导致它们出现不同的急慢性感染特征,其中Armstrong株在小鼠中可造成急性感染,而Clone13株可导致慢性感染,因此它们是建立急慢性病毒感染模型的理想选择[15-16]。野生型小鼠针对特定抗原内源特异性B细胞极少,研究B细胞应答依赖于过继转移抗原特异B细胞受体(B cell receptor,BCR)转基因B细胞。然而,目前尚无针对Armstrong和Clone13株特异的BCR转基因小鼠。LCMV-WE GP的单克隆抗体转基因小鼠KL25HL是目前唯一与LCMV相关的BCR转基因小鼠,其B细胞对WE株的GP具有特异性反应,而WE株通常导致急性感染[17-18]。因此解决Armstrong和Clone13毒株与KL25HL B细胞的适配性问题,是利用它们建立急性与慢性病毒感染B细胞应答机理研究模型的关键。
为此,本研究利用以质粒载体为基础的反向遗传技术,将野生LCMV-Armstrong(wild type LCMV-Armstrong, wtARM)株或野生LCMV-Clone13(wild type LCMV-Clone13, wtCL13)株的GP替换为WE株的GP,构建了包含WE株GP的重组LCMV-Armstrong(recombinant LCMV-Armstrong,rARM)和重组LCMV-Clone13(recombinant LCMV-Clone13,rCL13) 2种重组病毒。这2种重组病毒表现出与野生株相似的感染特征,分别引发急性和慢性感染,并且它们具有相同的GP抗原,可引起KL25HL B细胞的激活、增殖等免疫应答。因此,由这2种重组LCMV建立的小鼠病毒感染模型,在过继转移KL25HL B细胞后,可为研究B细胞在急性和慢性感染过程中的应答差异提供有效工具,有助于阐明B细胞在慢性感染过程中的功能缺陷机制。
1 材料与方法 1.1 材料 1.1.1 细胞、病毒及载体Vero细胞、BHK-21细胞购自中国科学院细胞库。野生型LCMV-Armstrong及LCMV-Clone13由美国加州大学旧金山分校Jason G. Cyster教授惠赠。pC-NP(表达LCMV NP)、pC-L-CL13(表达LCMV-Clone13 L蛋白)、pC-L-ARM(表达LCMV-Armstrong L蛋白)、pI-SWE-GP(-)(转录LCMV重组S片段,即Armstrong株S片段的GP序列被替换成WE株的GP序列)、pI-L-CL13(-)(转录LCMV-Clone13 L片段)及pI-L-ARM(-)(转录LCMV-Armstrong L片段)由瑞士巴塞尔大学Daniel D.Pinschewer教授惠赠。pLB载体由本实验室保存。
1.1.2 实验小鼠KL25HL小鼠为BCR转基因小鼠,其B细胞在LCMV-WE GP诱导后可发生特异性免疫应答。KL25HL小鼠由圣拉斐尔科学研究所Matteo Iannacone教授惠赠。BoyJ(CD45.1)小鼠购自The Jackson Laboratory。BoyJ(CD45.1)小鼠与KL25HL小鼠交配繁殖获得CD45.1-KL25HL小鼠。野生型C57BL/6J小鼠购自上海杰思捷实验动物有限公司。所有实验设计及操作符合动物福利伦理要求,动物伦理批准函:复旦大学基础医学院伦理审查批准函([2019]伦研第(013)号)。
1.1.3 实验试剂DMEM细胞培养基、RPMI 1640、Medium 199、青霉素/链霉素双抗溶液及胰蛋白酶-EDTA购自Gibco公司。胎牛血清(fetal bovine serum, FBS)购自Biological Industries公司。磷酸盐缓冲液(phosphate buffered saline, PBS)购自Corning公司。高保真聚合酶链反应(polymerase chain reaction, PCR)试剂盒、转染试剂lipofectamine 3000、RNA抽提试剂TRIzol LS、逆转录酶SuperScript Ⅲ、随机引物购及CellTrace Violet细胞增殖试剂盒购自Thermo公司。同源重组试剂盒购自南京诺唯赞公司。一步法逆转录试剂盒、SYBR Green qPCR试剂盒购自上海翌圣生物科技有限公司。流式抗体购自eBioscience公司。KL25HL BCR抗体购自圣拉斐尔科学研究所。
1.2 方法 1.2.1 LCMV病毒拯救BHK-21细胞铺于孔板,细胞会聚度长至80%时根据lipofectamine 3000操作手册进行转染,为拯救重组LCMV-Armstrong共转染pC-NP、pC-L-ARM、pI-SWE-GP(-)、pI-L-ARM(-),为拯救重组LCMV-Clone13共转染pC-NP、pC-L-CL13、pI-SWE-GP(-)、pI-L-CL13(-)。
1.2.2 LCMV病毒扩增按感染复数(multiplicity of infection, MOI) MOI=0.03~0.1取相应LCMV病毒量,于DMEM细胞培养基稀释后加入至BHK-21细胞(例如T75培养瓶,须将病毒液稀释为1 mL),在37 ℃培养箱孵育1.5 h,其间每15 min摇匀一次,再加入DMEM完全培养基培养48 h后收集病毒上清液,1 200 r/min 4 ℃离心10 min弃去细胞碎片,并用0.2 μm孔径过滤筛过滤后分装冻存于-80 ℃冰箱。
1.2.3 蚀斑滴定Vero细胞铺于6孔板,细胞会聚度长至80%时弃去旧培养基,加入1 mL新鲜DMEM完全培养基,再向其加入100 μL的系列梯度稀释的病毒液,在37 ℃培养箱孵育1.5 h,其间每20 min摇匀一次, 弃去培养基。加入4 mL噬斑培养基(由2×Medium199完全培养基和等体积平衡至42 ℃的1%琼脂糖溶液混合制备),37 ℃培养箱培养4 d。加入4 mL 7%甲醛溶液固定1 h后结晶紫染色30 min,弃去琼脂糖胶并用流动水冲洗,待干燥后观察噬斑数量。
1.2.4 病毒序列分析按病毒液∶TRIzol LS=3∶1抽提LCMV基因组RNA,根据逆转录酶SuperScript Ⅲ操作手册进行逆转录获得cDNA,通过PCR扩增获得GP片段并测序分析。扩增引物:5’-CGCACCGGGGATCCTAGGCTT-3’,5’-GTTCTTCAGCGTCTTTTCCAGACGG-3’。
1.2.5 pLB-GP载体构建以pI-SWE-GP(-)为模板,经PCR扩增获得GP的部分序列,经同源重组插入至pLB构建pLB-GP载体。引物序列:5’-GTTTTTCAGCAAGATGAGCCAGTGTAGAA~C-3’,5’-ATCTTCTAGAAAGATGCCTTGTT~G~TAGTCA-3’。
1.2.6 反转录-定量PCR(reverse transcription-quantitative PCR, RT-qPCR)检测病毒核酸载量分别取50 mg脾脏、肝脏及肾脏等组织,向其加入500 μL TRIzol制备成匀浆后提取RNA,根据翌圣一步法逆转录试剂盒操作手册获得cDNA作为模板。用SYBR Green qPCR试剂盒进行扩增,扩增引物[17]:GP-F: 5’-CATTCACCTGGACTTTG~TCAGACTC-3’, GP-R: 5’-GCAACTGCTGTG~TTCCCGAAAC-3’[19]。系列梯度稀释的pLB-GP质粒作为标准品,建立qPCR标准曲线,计算出病毒滴度。
1.2.7 细胞过继转移及小鼠感染颈椎脱臼处死KL25HL小鼠,取全身淋巴结及脾脏研磨过筛制备单细胞悬液,RPMI 1640洗涤、重悬,按3×106细胞/只经小鼠眼眶静脉丛注射,将特异性B细胞过继至野生型受体鼠。野生或重组Armstrong株,经腹腔注射每只小鼠2×105 PFU;野生或重组Clone13株,经尾静脉注射每只小鼠2×106 PFU。
1.2.8 流式细胞术检测B激活及增殖取小鼠脾脏制备单细胞悬液,流式抗体按1∶200稀释,冰上避光孵育40 min,FACS缓冲液(于PBS中加入2%的FBS配制)洗涤一次后重悬细胞,使用流式仪器Attune NxT Flow Cytomter(Thermo Scientific)进行检测。B细胞增殖情况根据过继前预先标记的CellTrace Violet稀释情况进行判断。
1.2.9 统计学方法与作图流式细胞术数据用FlowJ_V10软件处理,并使用GraphPad Prism9软件统计分析。组间比较采用t检验,P<0.05表示有统计学差异。
2 结果 2.1 表达WE株GP的rARM和rCL13的构建LCMV基因组(genome)由两条双义负链RNA组成,包括3.4 kb的小片段S与7.2 kb的大片段L,分别携带GP和NP以及Z蛋白和Pol(L蛋白)的编码信息(见图 1A)[10]。NP包裹S、L片段并与Pol相互作用,形成具备转录和复制活性的最小感染单位核糖核蛋白(ribonucleoprotein, RNP)复合体[20]。在Pol的催化下,以基因组S和L片段为模板,一方面合成相应的mRNA指导NP和Pol的翻译,另一方面转录出与其互补的反义基因组(antigenome)RNA以作为病毒复制模板,反义基因组S和L分别也是GP和Z蛋白mRNA的转录模板(见图 1A)[20]。因此,以质粒载体为基础体外拯救具有感染活性的重组LCMV,不仅须提供病毒核酸等遗传物质,而且须提供病毒转录、复制所必需的NP和Pol等反式激活蛋白[21]。首先,为在细胞内合成L片段,LCMV Armstrong和Clone13株L基因的全长cDNA序列被按照基因组方向分别克隆至RNA聚合酶I转录质粒骨架,从而构建获得pI-L-ARM(-)和pI-L-CL13(-)等载体(见图 1B)[22-23]。其次,为转录出重组S片段,Armstrong株S基因的cDNA的GP开放阅读框(open reading frame, ORF)被替换为WE株的GP序列,该cDNA进一步被克隆至上述转录质粒骨架而构建出pI-SWE-GP(-)载体(见图 1B)。最后,分别包含Armstrong和Clone13株Pol编码序列的pC-L-ARM和pC-L-CL13,以及包含Armstrong株NP编码序列(Clone13株NP编码序列与其一致)的pC-NP等质粒,是通过将相应序列克隆至pC表达载体骨架而构建获得,它们则为体外拯救重组LCMV提供Pol和NP等所需的反式激活蛋白[23]。如流程图 1C所示,为实现体外拯救rARM,本研究将pI-L-ARM(-)和pI-SWE-GP(-)转染至BHK-21细胞,它们可分别转录出Armstrong株基因组L片段及重组S片段,并且通过共转染pC-L-ARM和pC-NP表达出Pol和NP。L和S片段及Pol和NP等产物将在胞内组装成RNP进入病毒生命周期,并复制出子代病毒[24]。类似地,向BHK-21细胞共转染pI-L-CL13(-)、pI-SWE-GP(-)、pC-L-CL13和pC-NP等质粒以拯救rCL13。
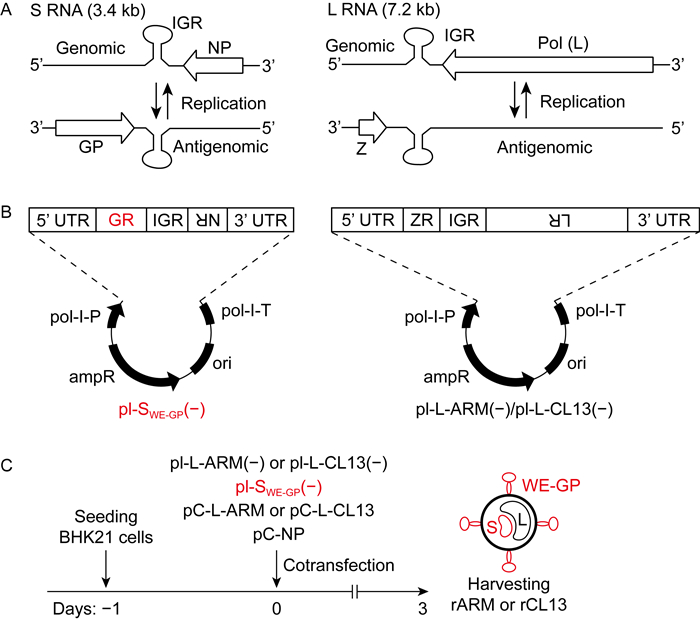
|
| A: Schematic of the bisegmented LCMV genome containing S RNA and L RNA. Each encodes two proteins in the opposite orientation separated by an intergenic region (IGR). S encodes glycoprotein (GP) and nucleoprotein (NP). L encodes Z protein (Z) and polymerase (Pol, or L). Antigenomic RNA transcribed from genomic RNA serves as a template for replication of virus. B: Schematic of the genome expression vectors. cDNA of the L or recombinant S segment were inserted into the pI plasmid backbone. Inverted writing indicates antisense polarity. pol-I-P: pol I promoter; pol-I-T: pol I terminator; UTR: untranslated region.C: Schematic of recovery rARM and rCL13 from plasmids which contain LCMV cDNA in vitro. BHK-21 cells were transfected with pC-L-ARM, pC-NP, pI-L-ARM(-) and pI-SWE-GP(-) to recover rARM. Alternatively, BHK-21 cells were transfected with pC-L-CL13, pC-NP, pI-L-CL13(-) and pI-SWE-GP(-) to recover rCL13. Culture supernatant was collected at the indicated time points. 图 1 rARM及rCL13的构建策略 Fig. 1 Construction strategies of rARM and rCL13 |
为了鉴定通过共转染pI-L-ARM(-)、pI-SWE-GP(-)、pC-L-ARM、pC-NP或pI-L-CL13(-)、pI-SWE-GP(-)、pC-L-CL13、pC-NP等质粒至BHK-21细胞所拯救获得的rARM和rCL13,本研究从收获的病毒液中提取RNA,并通过RT-PCR扩增重组病毒的GP片段(见图 2A)。将该片段测序后与基因库中的LCMV基因信息进行比对分析,结果表明,rARM和rCL13的GP序列与以往报道的LCMV-WE GP序列(AJ297484.1)完全一致(见图 2B)[25]。本研究进一步对rARM及rCL13的蚀斑形成能力进行考察,在BHK-21细胞转染上述质粒72 h后收获培养上清液,并使用该病毒上清液感染Vero细胞进行蚀斑形成实验。结果显示,rARM和rCL13感染Vero细胞后能形成蚀斑(见图 2C),且蚀斑形态、大小与先前报道的野生型LCMV所形成的蚀斑相似[26]。通过系列稀释对该拯救系统72 h后的病毒滴度进行测定,经过计算,rARM和rCL13滴度分别约为2.7×106 PFU/mL和1.6×105 PFU/mL。这一结果与Sánchez和Torre[21]、Flatz等[22]报道的体外拯救野生型LCMV的结果相符。
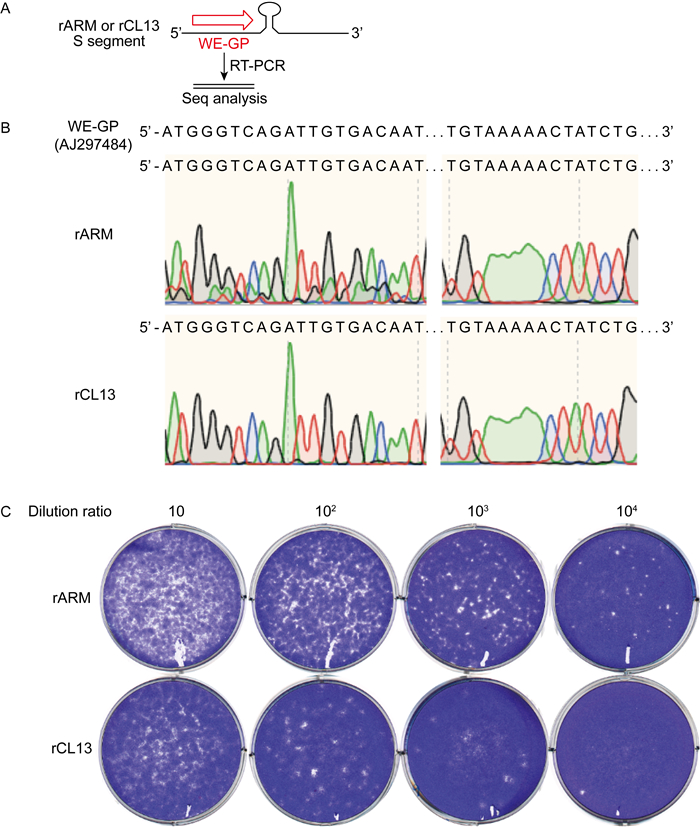
|
| A: Sequence analysis of rARM and rCL13 S segment. Schematic of amplification of the predicted fragments of rARM and rCL13 by RT-PCR (top). The alignment results between RT-PCR products and WE-GP sequence (AJ297484.1) reported previously (bottom). B: Plaque formation assay used to confirm the viral fitness of plaque formation and to quantify the titer of rARM and rCL13. 图 2 rARM及rCL13的鉴定 Fig. 2 Identification of rARM and rCL13 |
为探究rARM和rCL13在感染小鼠后,病毒在小鼠体内的持续时间以及致病情况。本研究对野生型C57BL/6J小鼠进行病毒感染,并监测其体重变化以及各器官中病毒滴度的变化。参考关于LCMV接种剂量影响感染结局的相关研究,rARM以每只鼠1×105 PFU的剂量通过腹腔注射进行感染,而rCL13以每只鼠2×106 PFU通过尾静脉注射进行感染[27]。结果显示,在感染rARM后,小鼠早期体重轻微下降,随后逐渐增加,与未感染的对照组小鼠体重变化一致(见图 3A);然而,感染rCL13后,小鼠体重显著下降,并且随着感染时间推移,其体重未能恢复至初始体重(见图 3A)。这一结果与感染wtARM或wtCL13后引起的小鼠体重变化特征相一致[28]。通过RT-qPCR对感染后小鼠各器官的病毒滴度进行分析,结果显示肝脏、脾脏及肾脏中的rARM和rCL13在感染后第4 d左右达到峰值(见图 3B~D)。随着时间推移,rARM在感染后第7 d基本被清除,而rCL13在感染后第1个月仍维持在高水平滴度,并且持续存在时间超过68 d(见图 3B~D)。综上所述,这些结果提示rARM和rCL13仍然保留了wtARM或wtCL13所具备的急性或慢性感染特点。
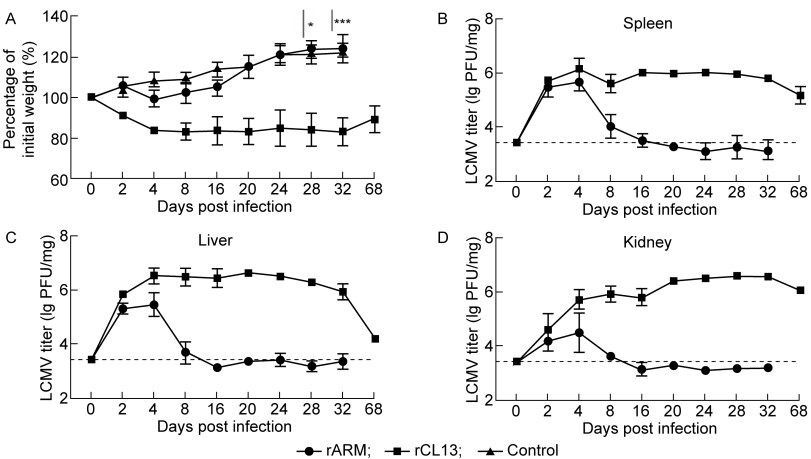
|
| C57BL/6J mice were either infected intraperitoneally with 1×105 PFU of rARM or intravenously with 2×106 PFU of rCL13, or they were left uninfected as control. A: Body weight changes of mice. B-D: Kinetics of viral nucleic acid loads. 50 mg spleen (B), liver (C) or kidney (D) samples were collected at the indicated time points to assess viral nucleic acid loads by RT-qPCR. *P < 0.05, **P < 0.01, ***P < 0.001. 图 3 感染rARM或rCL13后小鼠体重及各器官病毒核酸载量变化情况 Fig. 3 Kinetics of body weight and viral nucleic acid loads in various organs of mice infected with rARM or rCL13 |
为探究rARM和rCL13能否诱导KL25HL B细胞产生免疫应答反应,本研究将KL25HL B细胞过继转移到野生型C57BL/6J小鼠中,分别感染rARM和rCL13,并以感染wtARM和wtCL13作为对照,如图 4A所示。在感染后的第3 d,通过流式细胞术分析过继转移的特异性B细胞的激活和增殖状态。结果表明,在wtARM和wtCL13免疫条件下的KL25HL B细胞,与未免疫条件下的KL25HL B细胞在胞膜KL25HL BCR、CD86和Nur77等激活标志物的表达水平方面未见显著差异(见图 4B~D)。然而,在rARM和rCL13免疫的情况下,特异性B细胞的胞膜KL25HL BCR水平显著下降,且CD86和Nur77的表达显著上升(见图 4B~D)。胞膜BCR下调和激活标志物上调是B细胞激活的重要特征[29-30]。通过分析CellTrace Violet的稀释程度判断KL25HL B细胞的增殖情况。结果显示,尽管rARM诱导KL25HL B细胞增殖的能力不如rCL13,但与wtARM和wtCL13相比,在rARM或rCL13免疫下,KL25HL B细胞的增殖比例都显著增加(见图 4B~D)。上述结果表明,rARM和rCL13可以有效地诱导特异性B细胞发生激活和增殖等免疫应答。
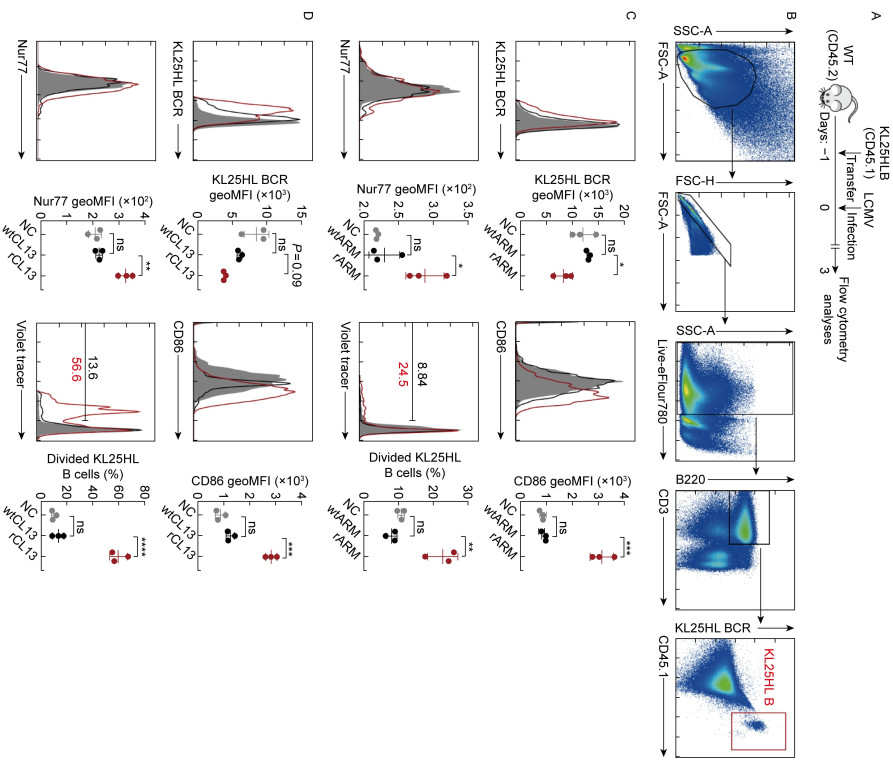
|
| C57BL/6 recipients (CD45.2) were adoptively transferred with 3×106 KL25HL B cells (CD45.1), and infected with wtARM, wtCL13, rARM, rCL13 or left uninfected. A: Schematic view of the experiments. B: Flow cytometry gating strategy for KL25HL B cells. C-D: Analyzing expression of membrane BCR and activation makers (CD86 and Nur77) on KL25HL B cells, as well as proliferation of KL25HL B cells in various infection conditions. C: wtARM and rARM. D: wtCL13 and rCL13. *P < 0.05, **P < 0.01, ***P < 0.001, ****P < 0.0001. 图 4 rARM及rCL13诱导KL25HL B细胞激活、增殖的情况 Fig. 4 The activation and proliferation of KL25HL B cells induced by rARM and rCL13 |
本研究应用反向遗传技术构建了重组LCMV病毒,该病毒可诱导WE株GP特异性BCR转基因细胞KL25HL B细胞发生免疫应答,为研究急性和慢性感染期间宿主体液免疫应答的差异建立了研究模型。通过共转染含有病毒基因序列的质粒载体,成功在体外拯救出rARM和rCL13两种重组病毒。经过测序分析,表明这两种重组病毒的GP序列与以往报道的WE株GP序列完全一致,并且它们感染细胞后具备形成蚀斑的能力。rARM和rCL13表现出与野生毒株相似的急性或慢性感染特征,其中rARM在感染后1周基本被清除,而rCL13在感染后持续时间超过2个月。与wtARM和wtCL13相比,rARM和rCL13能够显著诱导KL25HL B细胞发生激活、增殖等免疫应答。
B细胞介导的体液免疫对于机体控制感染至关重要。以往采用NP-KLH、NP-OVA、HEL-SRBC等免疫原或水泡性口炎病毒(vesicular stomatitis virus,VSV)等模式病毒免疫小鼠,以作为模型对B细胞应答机制进行研究,极大地加深了对于B细胞生发中心反应、浆细胞分化、抗体分泌等机制的认识[31-34]。然而,这些免疫原通常会被机体迅速清除,因此对于研究慢性感染背景下B细胞应答的机制具有一定的局限性。本研究构建的rARM和rCL13,具有相同的WE株GP抗原,并且分别导致急性和慢性感染,为系统性研究不同感染状态下B细胞的应答机制提供了一个良好的模型。Kräutler等[35]的研究表明,在急性或慢性感染的早期阶段,宿主体内中和抗体水平相当。然而,在感染的晚期阶段,慢性感染所诱导产生的中和抗体明显多于急性感染[35]。该现象可能与感染初期的特异性B细胞的命运决定有关,此过程涉及由I型干扰素介导的细胞毒性T淋巴细胞(cytotoxic T lymphocytes, CTLs)对B细胞的杀伤[36]。上述报道部分反映了本研究中观察到的现象,即感染rARM引起的KL25HL B细胞增殖较rCL13感染情况下少。
综上所述,本研究构建了可用于研究急性感染和慢性感染过程中B细胞应答差异的重组LCMV,为研究急性感染时宿主B细胞如何有效应答并清除病毒,以及慢性感染中病毒如何逃避机体免疫系统并持续存在等问题提供了有力的工具,为后续新型疫苗和免疫治疗药物的开发奠定了基础。
| [1] |
Nothelfer K, Sansonetti PJ, Phalipon A. Pathogen manipulation of B cells: the best defence is a good offence[J]. Nat Rev Microbiol, 2015, 13(3): 173-184.
[DOI]
|
| [2] |
Dörner T, Radbruch A. Antibodies and B cell memory in viral immunity[J]. Immunity, 2007, 27(3): 384-392.
[DOI]
|
| [3] |
Angeletti D, Gibbs JS, Angel M, Kosik I, Hickman HD, Frank GM, Das SR, Wheatley AK, Prabhakaran M, Leggat DJ, McDermott AB, Yewdell JW. Defining B cell immunodominance to viruses[J]. Nat Immunol, 2017, 18(4): 456-463.
[DOI]
|
| [4] |
Alsoussi WB, Malladi SK, Zhou JQ, Liu Z, Ying B, Kim W, Schmitz AJ, Lei T, Horvath SC, Sturtz AJ, McIntire KM, Evavold B, Han F, Scheaffer SM, Fox IF, Mirza SF, Parra-Rodriguez L, Nachbagauer R, Nestorova B, Chalkias S, Farnsworth CW, Klebert MK, Pusic I, Strnad BS, Middleton WD, Teefey SA, Whelan SPJ, Diamond MS, Paris R, O'Halloran JA, Presti RM, Turner JS, Ellebedy AH. SARS-CoV-2 Omicron boosting induces de novo B cell response in humans[J]. Nature, 2023, 617(7961): 592-598.
[DOI]
|
| [5] |
Li Y, Yin S, Issa R, Tong X, Wang G, Xia J, Huang R, Chen G, Weng D, Chen C, Wu C, Chen Y. B cell-mediated humoral immunity in chronic hepatitis B infection[J]. J Clin Transl Hepatol, 2021, 9(4): 592-597.
[DOI]
|
| [6] |
Amu S, Ruffin N, Rethi B, Chiodi F. Impairment of B-cell functions during HIV-1 infection[J]. AIDS, 2013, 27(15): 2323-2334.
[DOI]
|
| [7] |
Burton AR, Pallett LJ, McCoy LE, Suveizdyte K, Amin OE, Swadling L, Alberts E, Davidson BR, Kennedy PT, Gill US, Mauri C, Blair PA, Pelletier N, Maini MK. Circulating and intrahepatic antiviral B cells are defective in hepatitis B[J]. J Clin Invest, 2018, 128(10): 4588-4603.
[DOI]
|
| [8] |
闫静静, 仇超, 李亮助, 邱趁丽, 傅卫辉, 孙俊, 徐建青, 张晓燕. 人类免疫缺陷病毒1型慢性感染者B细胞表型分析及抗反转录病毒治疗的修复作用[J]. 微生物与感染, 2013, 8(3): 144-152. [URI]
|
| [9] |
Lapošová K, Pastoreková S, Tomášková J. Lymphocytic choriomeningitis virus: invisible but not innocent[J]. Acta Virol, 2013, 57(2): 160-170.
[DOI]
|
| [10] |
Zhou X, Ramachandran S, Mann M, Popkin DL. Role of lymphocytic choriomeningitis virus (LCMV) in understanding viral immunology: past, present and future[J]. Viruses, 2012, 4(11): 2650-2669.
[DOI]
|
| [11] |
Albariño CG, Palacios G, Khristova ML, Erickson BR, Carroll SA, Comer JA, Hui J, Briese T, St George K, Ksiazek TG, Lipkin WI, Nichol ST. High diversity and ancient common ancestry of lymphocytic choriomeningitis virus[J]. Emerg Infect Dis, 2010, 16(7): 1093-1100.
[DOI]
|
| [12] |
Suprunenko T, Hofer MJ. Complexities of type Ⅰ interferon biology: lessons from LCMV[J]. Viruses, 2019, 11(2): 172.
[DOI]
|
| [13] |
Sullivan BM, Emonet SF, Welch MJ, Lee AM, Campbell KP, de la Torre JC, Oldstone MB. Point mutation in the glycoprotein of lymphocytic choriomeningitis virus is necessary for receptor binding, dendritic cell infection, and long-term persistence[J]. Proc Natl Acad Sci U S A, 2011, 108(7): 2969-2974.
[DOI]
|
| [14] |
Bergthaler A, Flatz L, Hegazy AN, Johnson S, Horvath E, Löhning M, Pinschewer DD. Viral replicative capacity is the primary determinant of lymphocytic choriomeningitis virus persistence and immunosuppression[J]. Proc Natl Acad Sci U S A, 2010, 107(50): 21641-21646.
[DOI]
|
| [15] |
Ahmed R, Salmi A, Butler LD, Chiller JM, Oldstone MB. Selection of genetic variants of lymphocytic choriomeningitis virus in spleens of persistently infected mice. Role in suppression of cytotoxic T lymphocyte response and viral persistence[J]. J Exp Med, 1984, 160(2): 521-540.
[DOI]
|
| [16] |
Dutko FJ, Oldstone MB. Genomic and biological variation among commonly used lymphocytic choriomeningitis virus strains[J]. J Gen Virol, 1983, 64(Pt 8): 1689-1698.
[PubMed]
|
| [17] |
Bruns M, Cihak J, Müller G, Lehmann-Grube F. Lymphocytic choriomeningitis virus. Ⅵ. Isolation of a glycoprotein mediating neutralization[J]. Virology, 1983, 130(1): 247-251.
[DOI]
|
| [18] |
Hangartner L, Senn BM, Ledermann B, Kalinke U, Seiler P, Bucher E, Zellweger RM, Fink K, Odermatt B, Bürki K, Zinkernagel RM, Hengartner H. Antiviral immune responses in gene-targeted mice expressing the immunoglobulin heavy chain of virus-neutralizing antibodies[J]. Proc Natl Acad Sci U S A, 2003, 100(22): 12883-12888.
[DOI]
|
| [19] |
McCausland MM, Crotty S. Quantitative PCR technique for detecting lymphocytic choriomeningitis virus in vivo[J]. J Virol Methods, 2008, 147(1): 167-176.
[DOI]
|
| [20] |
Sarute N, Ross SR. New world arenavirus biology[J]. Annu Rev Virol, 2017, 4(1): 141-158.
[DOI]
|
| [21] |
Sánchez AB, de la Torre JC. Rescue of the prototypic Arenavirus LCMV entirely from plasmid[J]. Virology, 2006, 350(2): 370-380.
[DOI]
|
| [22] |
Flatz L, Bergthaler A, de la Torre JC, Pinschewer DD. Recovery of an arenavirus entirely from RNA polymerase Ⅰ/Ⅱ-driven cDNA[J]. Proc Natl Acad Sci U S A, 2006, 103(12): 4663-4668.
[DOI]
|
| [23] |
Lee KJ, Perez M, Pinschewer DD, de la Torre JC. Identification of the lymphocytic choriomeningitis virus (LCMV) proteins required to rescue LCMV RNA analogs into LCMV-like particles[J]. J Virol, 2002, 76(12): 6393-6397.
[DOI]
|
| [24] |
Lee KJ, Novella IS, Teng MN, Oldstone MB, de La Torre JC. NP and L proteins of lymphocytic choriomeningitis virus (LCMV) are sufficient for efficient transcription and replication of LCMV genomic RNA analogs[J]. J Virol, 2000, 74(8): 3470-3477.
[DOI]
|
| [25] |
Beyer WR, Miletic H, Ostertag W, von Laer D. Recombinant expression of lymphocytic choriomeningitis virus strain WE glycoproteins: a single amino acid makes the difference[J]. J Virol, 2001, 75(2): 1061-1064.
[DOI]
|
| [26] |
Beura LK, Scott MC, Pierson MJ, Joag V, Wijeyesinghe S, Semler MR, Quarnstrom CF, Busman-Sahay K, Estes JD, Hamilton SE, Vezys V, O'Connor DH, Masopust D. Novel lymphocytic choriomeningitis virus strain sustains abundant exhausted progenitor CD8 T cells without systemic viremia[J]. J Immunol, 2022, 209(9): 1691-1702.
[DOI]
|
| [27] |
Stamm A, Valentine L, Potts R, Premenko-Lanier M. An intermediate dose of LCMV clone 13 causes prolonged morbidity that is maintained by CD4+ T cells[J]. Virology, 2012, 425(2): 122-132.
[DOI]
|
| [28] |
Baazim H, Schweiger M, Moschinger M, Xu H, Scherer T, Popa A, Gallage S, Ali A, Khamina K, Kosack L, Vilagos B, Smyth M, Lercher A, Friske J, Merkler D, Aderem A, Helbich TH, Heikenwälder M, Lang PA, Zechner R, Bergthaler A. CD8+ T cells induce cachexia during chronic viral infection[J]. Nat Immunol, 2019, 20(6): 701-710.
[DOI]
|
| [29] |
Chaturvedi A, Martz R, Dorward D, Waisberg M, Pierce SK. Endocytosed BCRs sequentially regulate MAPK and Akt signaling pathways from intracellular compartments[J]. Nat Immunol, 2011, 12(11): 1119-1126.
[DOI]
|
| [30] |
Podojil JR, Sanders VM. CD86 and beta2-adrenergic receptor stimulation regulate B-cell activity cooperatively[J]. Trends Immunol, 2005, 26(4): 180-185.
[DOI]
|
| [31] |
Yeh CH, Finney J, Okada T, Kurosaki T, Kelsoe G. Primary germinal center-resident T follicular helper cells are a physiologically distinct subset of CXCR5hiPD-1hi T follicular helper cells[J]. Immunity, 2022, 55(2): 272-289.
[DOI]
|
| [32] |
Heise N, De Silva NS, Silva K, Carette A, Simonetti G, Pasparakis M, Klein U. Germinal center B cell maintenance and differentiation are controlled by distinct NF-κB transcription factor subunits[J]. J Exp Med, 2014, 211(10): 2103-2118.
[DOI]
|
| [33] |
Osma-Garcia IC, Capitan-Sobrino D, Mouysset M, Bell SE, Lebeurrier M, Turner M, Diaz-Muñoz MD. The RNA-binding protein HuR is required for maintenance of the germinal centre response[J]. Nat Commun, 2021, 12(1): 6556.
[DOI]
|
| [34] |
Fink K, Manjarrez-Orduño N, Schildknecht A, Weber J, Senn BM, Zinkernagel RM, Hengartner H. B cell activation state-governed formation of germinal centers following viral infection[J]. J Immunol, 2007, 179(9): 5877-5885.
[DOI]
|
| [35] |
Kräutler NJ, Yermanos A, Pedrioli A, Welten SPM, Lorgé D, Greczmiel U, Bartsch I, Scheuermann J, Kiefer JD, Eyer K, Menzel U, Greiff V, Neri D, Stadler T, Reddy ST, Oxenius A. Quantitative and qualitative analysis of humoral immunity reveals continued and personalized evolution in chronic viral infection[J]. Cell Rep, 2020, 30(4): 997-1012.
[DOI]
|
| [36] |
Moseman EA, Wu T, de la Torre JC, Schwartzberg PL, McGavern DB. Type Ⅰ interferon suppresses virus-specific B cell responses by modulating CD8+ T cell differentiation[J]. Sci Immunol, 2016, 1(4): eaah3565.
[DOI]
|
 2023, Vol. 18
2023, Vol. 18



