铜绿假单胞菌(Pseudomonas aeruginosa, P. aeruginosa)是一种常见的非发酵型需氧革兰氏阴性菌,广泛地分布在各种环境中,对环境具有强大的适应能力[1]。P. aeruginosa虽然是机会感染病原菌,但是常在医院引发医源性感染,而感染者大多数是需要机械通气的重症患者或者是长期住院的患者,所以治疗困难,合并其他系统疾病后死亡率高[2]。P. aeruginosa不仅可引起一系列下呼吸道感染疾病[3],而且能够在人体内长期定植且难以被清除,P. aeruginosa具有多重耐药菌株,例如耐碳青霉烯类铜绿假单胞菌(carbapenem-resistant P. aeruginosa, CRPA),导致临床抗生素治疗面临着难题[4-5]。
P. aeruginosa等绝大部分革兰氏阴性菌能够分泌直径在20~200 nm范围的外膜囊泡(outer membrane vesicle, OMV)。OMV内有着丰富的内容物,例如源细菌蛋白质、核酸物质(包括DNA、小分子RNA等)、脂质等[6],其中一些物质被称为感染宿主固有免疫系统的特异性抗原成分,即病原体相关分子模式(pathogen-associated molecular patterns, PAMPs)[7]。有研究指出,OMV反映出P. aeruginosa的阶段性活动,例如能够帮助P. aeruginosa入侵宿主黏膜屏障,在细菌之间水平转移耐药性基因[6]。除此之外,P. aeruginosa分泌的OMV也可以帮助维护生物膜的稳定性。
值得关注的是,细菌OMV的成分并不是固定的,与细菌分泌的方式有关系,并且受到细菌培养的环境条件例如温度、氧气、微量元素、抗生素等因素的影响[8-9]。抗生素压力是典型且常见的环境变化,有研究表明,抗生素能够导致细菌的SOS反应[10],产生更多的OMV,帮助改善P. aeruginosa在抗生素暴露情况下的存活率[11],这表明OMV性质的动态变化也反映着P. aeruginosa对环境变化的适应性改变。美洛培南是治疗P. aeruginosa感染的常用碳青霉烯类药物,但是近年来其耐药株CRPA层出不穷,呈现耐药程度高且致死率高的特点[12]。开展P. aeruginosa在美洛培南压力下产生OMV组分的研究以及对固有免疫系统刺激能力的研究,有助于了解P. aeruginosa在临床常用抗生素治疗下的适应性机制,进而了解其感染机制以及与宿主的免疫关联。
本研究以P. aeruginosa为研究对象,通过多种手段表征正常培养条件以及美洛培南抗生素压力下P. aeruginosa产生的OMV,分析其内含蛋白质的差异;观察P. aeruginosa在两种条件下产生的OMV对固有免疫细胞中的巨噬细胞的免疫刺激能力,以期发现OMV的潜在免疫原效应。
1 材料与方法 1.1 材料 1.1.1 菌株与细胞本研究采用的P. aeruginosa ATCC27853标准菌株来自华山医院临床微生物实验室。小鼠巨噬细胞系RAW264.7 (ATCC)系华山医院检验科课题组馈赠。
1.1.2 主要试剂Luria-Bertani(LB)培养基(生工科技,中国);美洛培南(meropenem)干粉(Abmole,美国);DMEM高糖培养基、青霉素-链霉素双抗溶液(Hyclone,美国);澳洲胎牛血清(fetal bovine serum, FBS)、0.25%含EDTA胰酶(Gibco,美国);IL-6, TNF-α, IL-8和TNF-γ ELISA试剂盒(复申科技, 中国);SteadyPure通用RNA提取试剂盒,Evo M-MLV RT Premix for qPCR,SYBR Green Premix Pro Taq HS qPCR Kit(艾科瑞生物,中国);BCA蛋白浓度测定试剂盒(增强型)(碧云天,中国)。
1.1.3 主要仪器37 ℃温箱、37℃摇床(Bluepart,中国);电子分析天平(METTLER TOLEDO,瑞士);光吸收酶标仪(Biotek,美国);超速离心机(Beckman,美国);透射电子显微镜(transmission electron microscopy, TEM)、纳升液相色谱四级杆轨道阱质谱仪、细胞培养箱、Nanodrop 2000分光光度计(Thermo Fisher,美国);纳米颗粒跟踪分析仪(NTA)(ParticleMetrix,德国);7500型快速实时荧光定量PCR仪、PCR扩增仪(ABI,美国)。
1.2 方法 1.2.1 P. aeruginosa 的培养P. aeruginosa ATCC27853最开始保存在含有40%甘油的Luria-Bertani (LB)肉汤中,并冻存在-80 ℃的冰箱中,实验之前,用无菌接种环接种至哥伦比亚血琼脂平板上,放置于37 ℃恒温培养箱中过夜。次日选取单个菌落接种于10 mL LB肉汤中,在37 ℃恒温摇床(200 r/min)中过夜以活化细菌。吸取等量上述增菌培养过的LB肉汤培养物,接种至2支新的含有40 mL LB肉汤培养基的50 mL无菌离心管中,放至250 r/min, 37 ℃恒温摇床振荡培养增菌备用。
1.2.2 P. aeruginosa 最低抑菌浓度(minimal inhibitory concentration, MIC)的测定遵循美国临床和实验室标准协会(Clinical and Laboratory Standards Institute, CLSI) M100-Ed32推荐的微量肉汤稀释法步骤,测定美洛培南对P. aeruginosa ATCC27853的MIC[13]。
1.2.3 P. aeruginosa OMV 的提取正常培养的P. aeruginosa(Normal)的增菌处理: 在生物安全柜中无菌操作,将ATCC27853的增菌LB肉汤培养基在200 r/min,37 ℃恒温摇床培养18~24 h,增菌处理至细菌悬液的OD600大约为1.0。美洛培南处理的P. aeruginosa (MEM):待菌悬液OD600达到0.5~0.6(对数生长期)时,于室温条件下8 000×g离心10 min。再向细菌中加入含有4倍MIC(4 μg/mL)的美洛培南-LB培养基。37 ℃恒温摇床培养18~24 h。以上菌悬液进行8 000×g离心10 min,收集上清液,滤菌后在100 KDa无菌超滤离心管中进行浓缩后150 000×g超高速离心法离心3 h,然后将沉淀重悬于100 μL无菌PBS中并储存于-20 ℃中备用。
1.2.4 P. aeruginosa OMV 的表征形态:所得细菌OMV样品用1.5%醋酸铀水溶液负染后在300目碳涂层铜透射电子显微镜(transmission electron microscopy, TEM)栅格上观察。
粒子浓度与大小:OMV样品稀释后利用纳米颗粒跟踪分析仪(nanoparticle tracking analyzer, NTA)对每一份样品进行浓度和粒径分布的分析,重复3次。根据各条件的稀释系数计算OMV的总浓度。
1.2.5 P. aeruginosa OMV 蛋白质提取与液相色谱-质谱联用分析向P. aeruginosa OMV样品中加入配制好的蛋白裂解液(专用细菌蛋白裂解液:蛋白酶抑制剂=100∶1),充分裂解后,将其置于4 ℃,12 000 r/min离心20 min。离心结束后吸取上清液即为细菌囊泡蛋白,放置于-80 ℃冰箱备用。使用BCA法检测蛋白浓度,采用胶分离-脱色-酶解的方法,将样品蛋白质在纳升液相色谱四极杆轨道阱质谱仪中上样,利用液相色谱-质谱(liquid chromatography tandem mass spectrometry,LC-MS/MS)技术分离OMV蛋白质,并利用Proteome Discoverer软件在P. aeruginosa蛋白数据库中进行比对。
1.2.6 生物信息学分析蛋白质谱数据来源于Uniprot网站(https://www.uniprot.org),用R 3.3.4 (https://www.r-project.org/)进行分析。对蛋白质数据在Gene Ontology数据库网站 http://www.geneontology.org中进行GO分析,在KEGG在线服务工具 http://www.genome.jp/kegg/pathway.html中进行KEGG通路注释。
1.2.7 小鼠巨噬细胞RAW264.7细胞的培养RAW264.7细胞培养基为含有10% FBS及2% 青霉素-链霉素双抗的DMEM高糖培养基,在含5%二氧化碳的37 ℃培养箱中进行培养。当细胞密度达到90%以上时即可传代,由于RAW264.7细胞对外界环境刺激变化敏感,为保证不分化,传代过程中,不适用胰酶以及离心操作,仅用巴氏吸管进行轻柔地吹打。
1.2.8 IL-6, TNF-α, IL-8,IFN-γ的浓度检测收集生长对数期的RAW264.7细胞,以5×105 cells/mL的密度每孔400 μL接种于24孔板中,每个组别设置3个复孔。贴壁之后,弃去培养基替换成最终浓度为1 mg/mL的正常OMV与MEM-OMV的完全培养基进行培养,同时设置空白对照组(等量PBS)。细胞在37 ℃、5% CO2条件下培养,24 h后,收集细胞上清液去除细胞碎片后利用酶联免疫吸附试验(enzyme-linked immunosorbent assay, ELISA),检测IL-6, TNF-α, IL-8,TNF-γ在细胞培养液中的浓度。
1.3 数据处理与分析实验数据使用GraphPad Prism 9.0软件进行统计,两两比较的数据使用t检验进行分析,多组间数据比较使用one-way ANOVA检验分析。用P值表示统计学差异,若P < 0.05则证明差异具有统计学意义,具体在图中的展示形式为*,P < 0.05;* * * *,P < 0.000 1。
2 结果 2.1 P. aeruginosa 的MIC值测量P. aeruginosa ATCC27853是用于药敏试验的质控菌株。用微量肉汤稀释法检测菌株的MIC值,根据美国临床和实验室标准协会(Clinical and Laboratory Standards Institute, CLSI) M100-ED32, 菌株有耐药(resistant, R)、敏感(sensitive, S) 和中介(intermediate, I)3种类型。P. aeruginosa的折点数据如表 1所示。美洛培南对P. aeruginosa ATCC27853的MIC为1 μg/mL,根据CLSI的参考折点,该菌株符合标准。
| 抗生素 | CLSI参考折点(μg/mL) | ATCC27853 | ||||
| S | I | R | MIC (μg/mL) | 结果 | ||
| 美洛培南 | ≤2 | 4 | ≥8 | 1 | S | |
| S: sensitive (敏感);I: intermediate (中介); R: resistant (耐药)。 | ||||||
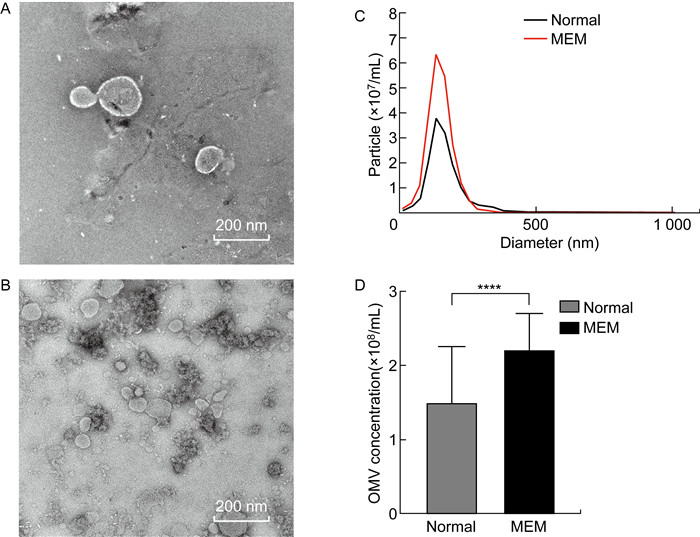
本研究中对P. aeruginosa生长所需要的LB肉汤培养基进行了处理,除了正常培养之外,还在培养基中加入了一定浓度的美洛培南,在该条件以及正常培养的P. aeruginosa培养基上清液中分离出OMV,然后用透射电子显微镜观察并拍摄不同条件下产生的OMV形态,利用纳米粒径分析系统对几种不同的OMV进行颗粒大小和浓度的表征,结果如图 1所示。
|
| A: TEM images of OMV generated by normal culture conditions (Normal). B: TEM image of OMV produced after the addition of meropenem (MEM). C: Particle concentration-size distribution of OMV in 2 groups. D: Original concentration of OMV in 2 groups.* * * *, P < 0.000 1. 图 1 P. aeruginosa OMV的表征 Fig. 1 Characterization of OMV of P. aeruginosa |
图 1A,B显示在2种条件下产生的OMV都具有明显双层膜结构。图 1C,D结果显示,在2种条件下分离的OMV峰值直径相似,但颗粒浓度数量级上有显著差距,美洛培南刺激组培养的OMV (MEM-OMV)的颗粒浓度明显高于正常LB肉汤培养组的OMV(Normal-OMV),MEM-OMV的颗粒浓度是(2.20±0.53)×108/mL,Normal-OMV的颗粒浓度是(1.50±0.76)×108/mL(P < 0.000 1)。如表 2所示,两组的OMV大小的差异并不显著,Normal-OMV平均直径为169±75.5 nm,MEM-OMV的平均直径为150.2±50.3 nm。
| 处理条件 | OMV颗粒平均浓度(/mL) | 粒子直径(nm) | 峰值直径(nm) |
| 正常组(Normal) | (1.50±0.76)×108 | 169±75.5 | 162.4 |
| 美洛培南(MEM) | (2.20±0.53)×108 | 150.2±50.3 | 157.4 |
| Data are shown as mean ± SD of three independent experiments. | |||
从在不同处理的培养基中生长的P. aeruginosa ATCC27853菌株中分离得到OMV并提取蛋白质,然后消化成肽段并使用液相色谱-质谱联用技术进行分析,与数据库比对,采用一定的生物信息学方法对不同的OMV蛋白组分进行分析和功能预测。
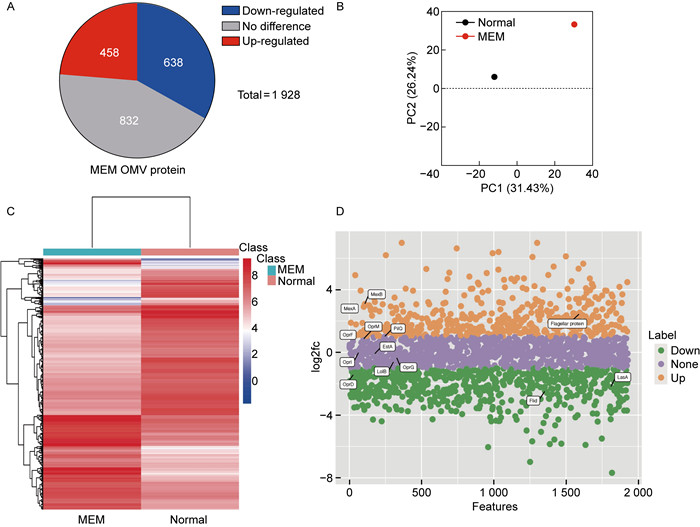
2.3.1 P. aeruginosa OMV 的蛋白质种类总览从P. aeruginosa ATCC27853菌株的OMV中提取蛋白质消化成肽段,使用LC-MS/MS技术进行分析,并比较这些在不同生长环境下P. aeruginosa的OMV蛋白质,以期发现不同OMV中蛋白质的选择性富集。如图 2A所示,在P. aeruginosa蛋白质数据库检索到的1 928种蛋白质中,与Normal组相比,MEM-OMV表达上调的蛋白有458种,表达下调的蛋白有638种,但某些蛋白质的含量没有区别,共有832种。图 2B的主成分分析(principal component analysis,PCA)以及图 2C的蛋白质丰度热图显示,MEM-OMV和PMB-OMV的蛋白质组成与正常组相比差异较大,美洛培南在培养基中对P. aeruginosa的某种作用,使得部分蛋白质被选择性更多或更少地进入OMV。此外,MEM-OMV中外排泵蛋白MexA、MexB和鞭毛蛋白的含量显著增多,外膜蛋白OprD、Flid以及毒力因子LasA的含量显著减少(见图 2D)。
|
| A: Distribution of number of protein expression differences in MEM-OMV compared with Normal-OMV. B: Principal component analysis (PCA) of Normal-OMV and MEM-OMV. C: Protein identification and abundance heatmap of differentially expressed proteins in Normal-OMV and MEM-OMV (P < 0.05, one-way ANOVA). The figure shows the combined data for 3 biological replicates. D: Volcano diagram of MEM-OMV protein compared with Normal-OMV. P < 0.05 & log2 fold change < -0.5 (green); P < 0.05 & log2 fold change > 0.5(orange); P > 0.05 (purple). 图 2 MEM-OMV与Normal-OMV的蛋白质比较 Fig. 2 The proteins in MEM-OMV compared with the proteins in Normal-OMV |
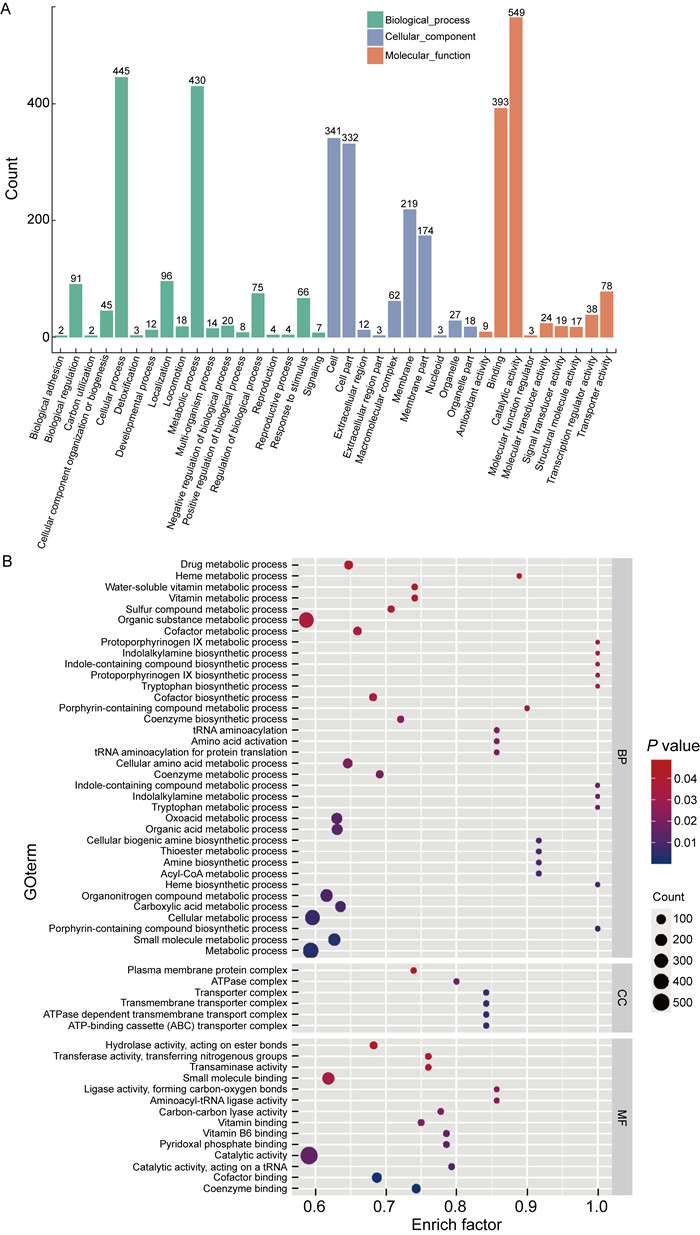
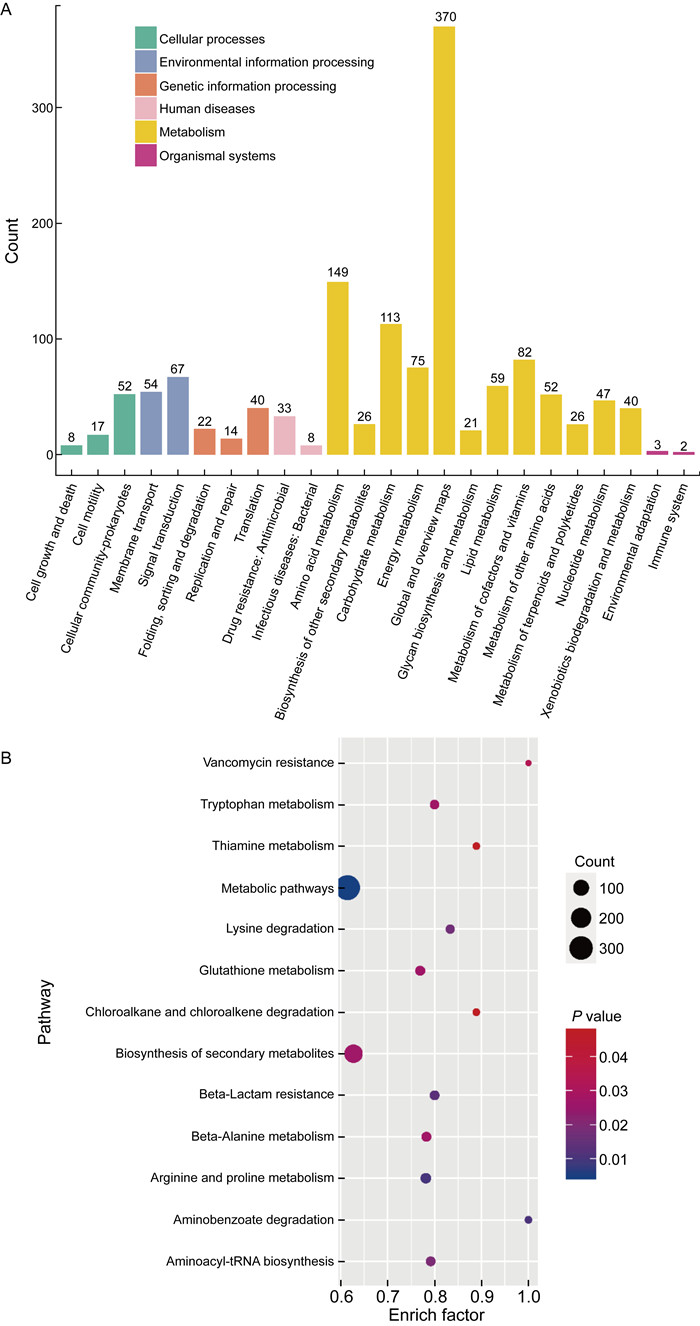
通过基因本体(gene ontology, GO)分析和京都基因和基因组百科(Kyoto encyclopedia of genes and genomes, KEGG)通路富集分析对不同组别的OMV蛋白表达谱进行进一步表征,详见图 3A、图 4A。根据GO分析的二级分类,将蛋白质的功能分为生物学过程(biological process, BP)、细胞组成(cellular component, CC)和分子功能(molecular function, MF)。
|
| A: Species distribution of proteins in MEM-OMV; B: Bubble plots of GO analysis on differential proteins between MEM-OMV and Normal-OMV. 图 3 两组P. aeruginosa OMV差异蛋白的GO分析 Fig. 3 GO analysis of differential proteins of P. aeruginosa OMV in two groups |
|
| A: Distribution of functional pathways of proteins in MEM-OMV; B: Bubble plots of KEGG pathway analysis on differential proteins between MEM-OMV and Normal-OMV. 图 4 两组P. aeruginosa OMV差异蛋白的KEGG分析 Fig. 4 KEGG analysis of differential proteins of P. aeruginosa OMV in two groups |
图 3B的气泡图显示:与Normal-OMV相比,MEM-OMV中富集了与代谢过程相关的一系列蛋白质,例如质膜蛋白复合体、ATP酶复合体、跨膜转运体复合体、ATP-结合盒(ABC)转运体复合体、催化活性、辅因子结合、辅酶结合的通路相关蛋白质;图 4B的KEGG分析气泡图显示:MEM-OMV的蛋白质在万古霉素耐药途径、代谢途径、氨基酸代谢与降解途径、β-内酰胺耐药途径、氨基酰-tRNA生物合成途径中富集。
2.4 P. aeruginosa OMV 刺激固有免疫细胞分泌细胞因子将小鼠的巨噬细胞RAW264.7细胞分别与1 mg/mL Normal-OMV和MEM-OMV共孵育48 h,并通过ELISA分析细胞因子IL-6、TNF-α、IL-8和TNF-γ的表达水平。如图 5所示,与空白对照相比,Normal-OMV刺激RAW 264.7细胞分泌IL-6、TNF-α和TNF-γ的浓度分别为62.21±0.858 pg/mL,352.93±40.632 pg/mL,549.20±15.421 pg/mL, 空白对照组IL-6、TNF-α和TNF-γ的浓度分别为39.20±1.067 pg/mL,278.77±3.796 pg/mL,379.87±13.467 pg/mL,Normal-OMV显著促进了RAW 264.7细胞分泌以上几种细胞因子(P < 0.05),IL-8的表达水平无显著差异(空白:58.13±3.512 pg/mL;MEM-OMV:65.40±3.051 pg/mL)。同样,结果显示MEM-OMV刺激RAW 264.7细胞分泌IL-6、TNF-α、IL-8和TNF-γ的浓度分别为115.99±1.689 pg/mL,635.03±13.505 pg/mL,127.93±5.267 pg/mL和906.80±16.549 pg/mL,显著高于Normal-OMV刺激RAW 264.7细胞时分泌的细胞因子水平(P < 0.000 1)。
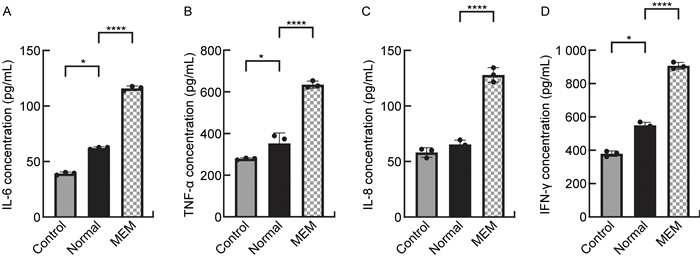
|
| Data are presented as mean±SD of three independent experiments. *, P < 0.05, * * * *, P < 0.000 1. 图 5 RAW264.7巨噬细胞与Normal-OMV、MEM-OMV共孵育48 h后细胞因子的表达 Fig. 5 Cytokine expression in RAW264.7 macrophages incubated with Normal-OMV versus MEM-OMV for 48 h |
P. aeruginosa是常见的机会性感染病原体之一,也是用来研究细菌分泌OMV的模式菌,能够在医院广泛传播与流行,对杀菌剂以及抗生素的抵抗性、生物膜的存在使得P. aeruginosa一旦定植就难以被清除,如何实现完全清除细菌负荷、恢复患者肺功能的目标,是一个棘手的临床难题。目前的治疗思路中,抗生素的联合治疗除了会造成肝肾毒性以及肠道菌群失调等不良反应之外,有时还会使得感染情况加重[14]。因此在存在抗生素的情况下,研究P. aeruginosa如何生存、加重宿主感染,甚至逃脱免疫系统的监管至关重要。OMV是P. aeruginosa重要的分泌物,具有多种生物学功能。而且据以往的文献报道,OMV的产生受到细菌生长环境的控制,其中也包括抗生素的压力[15],产生了能够帮助P. aeruginosa适应环境的OMV群体[8, 16]。因此,研究不同培养条件尤其是抗生素压力对细菌OMV的异质性影响,是一项重要的课题。美洛培南在临床治疗P. aeruginosa感染中起到重要作用,同时也能分离出相应的耐药株,故其适合作为典型抗生素进行研究。本研究分别提取了相同时间内P. aeruginosa在正常培养基和添加了一定浓度的美洛培南培养基中分泌的OMV,表征其尺寸以及浓度,同时提取蛋白质进行质谱分析。结果显示,OMV的释放受美洛培南的影响,浓度显著提升。原因在于:①美洛培南破坏了P. aeruginosa细胞壁的完整性,可以促进OMV从细菌体中释放;②有研究证实,P. aeruginosa在某些抗生素的作用下,会形成SOS应答[17]。SOS反应是一种诱导性DNA修复途径,主要被细菌DNA的损伤所触发,也有其他的响应机制,例如抑制细菌生长或者活性氧(reactive oxygen species, ROS)调节[10]。包括抗生素药物在内的外界因素可以诱发ROS调节和提升细菌的ROS水平,从而损害DNA、蛋白质和脂质,诱导SOS反应[18],释放更多的OMV就是细菌通过该反应完成环境适应的机制之一。
此外,除了物理表征,OMV蛋白质表达水平也因为美洛培南的加入出现上调与下调,MEM-OMV中与P. aeruginosa外排泵系统相关的MexA&B上调[19],除此之外,蛋白Flid和毒力因子LasA表达下调,这2种蛋白与囊性纤维化患者呼吸道中P. aeruginosa的定植有关[20]。美洛培南压力下的P. aeruginosa分泌的OMV毒力降低,在某种程度上削弱了P. aeruginosa对气道和肺部的入侵能力。美洛培南在P. aeruginosa生长期间改变了细菌释放OMV过程中蛋白质的包装,这些差异蛋白的功能定位也集中在耐药以及代谢物生物合成方面,进一步验证了SOS反应中细菌的营养物质合成会受到影响,后续的变化也通过OMV展现出来。
有文献报道,P. aeruginosa的OMV以TLR4依赖的方式入侵人类上皮细胞和巨噬细胞,导致TLR4的表达以正反馈机制上调,从而驱动进一步的炎症反应[21]。在培养基中加入美洛培南后P. aeruginosa产生的OMV也提高了巨噬细胞中炎性细胞因子IL-6、TNF-α、IL-8、TNF-γ的分泌水平。根据文献报道,P. aeruginosa产生囊泡的机制主要有两大类,一类是由于细胞壁和细胞外膜发生结构变化,在自然状态下产生细菌外膜囊泡;另一类是由外部的因素导致一部分细菌发生了裂解并产生OMV,称为爆炸性细胞裂解OMV(explosive cell lysis OMV,E-OMV)[22]。MEM-OMV的GO分析结果也显示,差异蛋白集中在与代谢相关的胞内蛋白,综合美洛培南有破坏细菌细胞壁结构的特点,提示MEM-OMV属于E-OMV。综合文献报道与蛋白质组学结果,我们推测,与自然状态下产生的OMV相比,MEM-OMV富含了更多的致病蛋白与营养获取蛋白,这些蛋白质通过OMV接触到巨噬细胞表面的Toll样受体(Toll-like receptors) 2和4(TLR2,TLR4),或者在巨噬细胞内由线粒体功能障碍激活NLRP3炎症小体,进一步促进细胞因子的分泌[23-24]。因此,巨噬细胞分泌水平提高的原因与MEM-OMV中富含的某些蛋白质有关。IL-6、TNF-α和TNF-γ在抗感染作用中与巨噬细胞的激活和启动下一级免疫反应密切相关,能够诱导形成M1型巨噬细胞,并吞噬破坏微生物;IL-8则能够趋化和激活中性粒细胞和一部分淋巴细胞[25]。这些细胞因子分泌水平的显著提高,证实了OMV具有被巨噬细胞识别与内化的能力,而且在特定条件下被改变的OMV能够使巨噬细胞分泌更多在抵御病原体入侵过程中起着重要作用的细胞因子。最近的研究指出,这种细胞因子的表达增加可能有利于先天免疫细胞的“免疫训练”[26],外来免疫原性物质持续的刺激可以使宿主免疫系统对下一次病原体入侵做出更快的反应[27]。
综上所述,在美洛培南的环境压力下,P. aeruginosa分泌OMV的量增加,蛋白质表达量改变,OMV的这些变化间接调节了其随后对巨噬细胞的作用。关注OMV中差异显著的蛋白以及影响免疫细胞的途径,有助于了解不同外部条件影响下P. aeruginosa的致病机制。更多的环境变化因素以及OMV内容物值得进一步探索,这些研究有助于实现在不同的环境中制定相应的除菌以及抗感染治疗的策略。
| [1] |
Crone S, Vives-Flórez M, Kvich L, Saunders AM, Malone M, Nicolaisen MH, Martínez-García E, Rojas-Acosta C, Catalina Gomez-Puerto M, Calum H, Whiteley M, Kolter R, Bjarnsholt T. The environmental occurrence of Pseudomonas Aeruginosa[J]. Apmis, 2020, 128(3): 220-231.
[DOI]
|
| [2] |
Wang K, Chen YQ, Salido MM, Kohli GS, Kong JL, Liang HJ, Yao ZT, Xie YT, Wu HY, Cai SQ, Drautz-Moses DI, Darling AE, Schuster SC, Yang L, Ding Y. The rapid in vivo evolution of Pseudomonas Aeruginosa in ventilator-associated pneumonia patients leads to attenuated virulence[J]. Open Biol, 2017, 7(9): 170029.
[DOI]
|
| [3] |
Rossi E, La Rosa R, Bartell JA, Marvig RL, Haagensen JAJ, Sommer LM, Molin S, Johansen HK. Pseudomonas Aeruginosa adaptation and evolution in patients with cystic fibrosis[J]. Nat Rev Microbiol, 2021, 19(5): 331-342.
[DOI]
|
| [4] |
Jurado-Martín I, Sainz-Mejías M, Mcclean S. Pseudomonas Aeruginosa: an audacious pathogen with an adaptable arsenal of virulence factors[J]. International Journal of Molecular Sciences, 2021, 22(6): 3128.
[DOI]
|
| [5] |
Tenover FC, Nicolau DP, Gill CM. Carbapenemase-producing Pseudomonas Aeruginosa-An emerging challenge[J]. Emerg Microbes Infect, 2022, 11(1): 811-814.
[DOI]
|
| [6] |
Juodeikis R, Carding Simon R. Outer membrane vesicles: biogenesis, functions, and issues[J]. Microbiol Mol Biol Rev, 2022, 86(4): e0003222.
[DOI]
|
| [7] |
Bitto NJ, Baker PJ, Dowling JK, Wray-Mccann G, De Paoli A, Tran LS, Leung PL, Stacey KJ, Mansell A, Masters SL, Ferrero RL. Membrane vesicles from pseudomonas aeruginosa activate the noncanonical inflammasome through caspase-5 in human monocytes[J]. Immunol Cell Biol, 2018, 96(10): 1120-1130.
[DOI]
|
| [8] |
Orench-Rivera N, Kuehn MJ. Environmentally controlled bacterial vesicle-mediated export[J]. Cell Microbiol, 2016, 18(11): 1525-1536.
[DOI]
|
| [9] |
Metruccio MM, Evans DJ, Gabriel MM, Kadurugamuwa JL, Fleiszig SM. Pseudomonas Aeruginosa outer membrane vesicles triggered by human mucosal fluid and lysozyme can prime host tissue surfaces for bacterial adhesion[J]. Front Microbiol, 2016, 7: 871.
[DOI]
|
| [10] |
Baggett NS, Bronson AS, Cabeen MT. Sos-independent pyocin production in P. Aeruginosa is induced by xerc recombinase deficiency[J]. Mbio, 2021, 12(6): e0289321.
[DOI]
|
| [11] |
Macdonald IA, Kuehn MJ. Stress-induced outer membrane vesicle production by Pseudomonas Aeruginosa[J]. J Bacteriol, 2013, 195(13): 2971-2981.
[DOI]
|
| [12] |
Reyes J, Komarow L, Chen L, Ge L, Hanson BM, Cober E, Herc E, Alenazi T, Kaye KS, Garcia-Diaz J, Li L, Kanj SS, Liu Z, Oñate JM, Salata RA, Marimuthu K, Gao H, Zong Z, Valderrama-Beltrán SL, Yu Y, Tambyah P, Weston G, Salcedo S, Abbo L M, Xie Q, Ordoñez K, Wang M, Stryjewski ME, Munita JM, Paterson DL, Evans S, Hill C, Baum K, Bonomo RA, Kreiswirth BN, Villegas MV, Patel R, Arias CA, Chambers HF, Fowler VG, J r., Doi Y, Van Duin D, Satlin MJ. Global epidemiology and clinical outcomes of carbapenem-resistant Pseudomonas Aeruginosa and associated carbapenemases (pop): a prospective cohort study[J]. Lancet Microbe, 2023, 4(3): e159-e170.
[DOI]
|
| [13] |
The Clinical And Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing[S]. 32nd Ed. Malvern, Pennsylvania: The Clinical And Laboratory Standards Institute, 2022.
|
| [14] |
中华医学会呼吸病学分会感染学组. 中国铜绿假单胞菌下呼吸道感染诊治专家共识(2022年版)[J]. 中华结核和呼吸杂志, 2022(8): 739-752. |
| [15] |
Li Q, Li J, He T, Ji X, Wei R, Yu M, Wang R. Sub-Mic antibiotics modulate productions of outer membrane vesicles in tigecycline-resistant Escherichia Coli[J]. Antibiotics (Basel), 2024, 13(3): 276.
[DOI]
|
| [16] |
Sartorio MG, Pardue EJ, Feldman MF, Haurat MF. Bacterial outer membrane vesicles: from discovery to applications[J]. Annu Rev Microbiol, 2021, 75: 609-630.
[DOI]
|
| [17] |
Maredia R, Devineni N, Lentz P, Dallo SF, Yu J, Guentzel N, Chambers J, Arulanandam B, Haskins WE, Weitao T. Vesiculation from Pseudomonas Aeruginosa under sos[J]. Scientific World Journal, 2012, 2012: 402919.
[DOI]
|
| [18] |
Podlesek Z, Žgur Bertok D. The DNA damage inducible sos response is a key player in the generation of bacterial persister cells and population wide tolerance[J]. Front Microbiol, 2020, 11: 1785.
[DOI]
|
| [19] |
Pan YP, Xu YH, Wang ZX, Fang YP, Shen JL. Overexpression of mexab-oprm efflux pump in carbapenem-resistant Pseudomonas Aeruginosa[J]. Arch Microbiol, 2016, 198(6): 565-571.
[DOI]
|
| [20] |
Arora SK, Ritchings BW, Almira EC, Lory S, Ramphal R. The Pseudomonas Aeruginosa flagellar cap protein, flid, is responsible for mucin adhesion[J]. Infect Immun, 1998, 66(3): 1000-1007.
[DOI]
|
| [21] |
Wang X, Eagen WJ, Lee JC. Orchestration of human macrophage Nlrp3 inflammasome activation by staphylococcus aureus extracellular vesicles[J]. Proc Natl Acad Sci U S A, 2020, 117(6): 3174-3184.
[DOI]
|
| [22] |
Zavan L, Fang H, Johnston EL, Whitchurch C, Greening DW, Hill AF, Kaparakis-Liaskos M. The mechanism of Pseudomonas Aeruginosa outer membrane vesicle biogenesis determines their protein composition[J]. Proteomics, 2023, 23(10): e2200464.
[DOI]
|
| [23] |
Park KS, Lee J, Jang SC, Kim SR, Jang MH, Lötvall J, Kim YK, Gho YS. Pulmonary inflammation induced by bacteria-free outer membrane vesicles from Pseudomonas Aeruginosa[J]. Am J Respir Cell Mol Biol, 2013, 49(4): 637-645.
[DOI]
|
| [24] |
Deo P, Chow SH, Han ML, Speir M, Huang C, Schittenhelm RB, Dhital S, Emery J, Li J, Kile BT, Vince JE, Lawlor KE, Naderer T. Mitochondrial dysfunction caused by outer membrane vesicles from gram-negative bacteria activates intrinsic apoptosis and inflammation[J]. Nat Microbiol, 2020, 5(11): 1418-1427.
[DOI]
|
| [25] |
Mcelvaney OJ, Mcelvaney NG. Targeting Il-8 in cystic fibrosis: enough but not too much[J]. Am J Respir Cell Mol Biol, 2018, 59(4): 401-402.
[DOI]
|
| [26] |
Priem B, Van Leent MMT, Teunissen AJP, Sofias AM, Mourits VP, Willemsen L, Klein ED, Oosterwijk RS, Meerwaldt AE, Munitz J, Prévot G, Vera Verschuur A, Nauta SA, Van Leeuwen EM, Fisher EL, De Jong KAM, Zhao Y, Toner YC, Soultanidis G, Calcagno C, Bomans PHH, Friedrich H, Sommerdijk N, Reiner T, Duivenvoorden R, Zupančič E, Di Martino JS, Kluza E, Rashidian M, Ploegh HL, Dijkhuizen RM, Hak S, Pérez-Medina C, Bravo-Cordero JJ, De Winther MPJ, Joosten LAB, Van Elsas A, Fayad ZA, Rialdi A, Torre D, Guccione E, Ochando J, Netea MG, Griffioen AW, Mulder WJM. Trained immunity-promoting nanobiologic therapy suppresses tumor growth and potentiates checkpoint inhibition[J]. Cell, 2020, 183(3): 786-801.
[DOI]
|
| [27] |
Liu G, Ma N, Cheng K, Feng Q, Ma X, Yue Y, Li Y, Zhang T, Gao X, Liang J, Zhang L, Wang X, Ren Z, Fu YX, Zhao X, Nie G. Bacteria-derived nanovesicles enhance tumour vaccination by trained immunity[J]. Nat Nanotechnol, 2024, 19(3): 387-398.
[DOI]
|
 2024, Vol. 19
2024, Vol. 19


