2. 复旦大学基础医学院病原生物学系,教育部/卫健委医学分子病毒学重点实验室,上海 200032;
3. 上海市肝病研究所,上海 200032
2. Department of Medical Microbiology and Parasitology, School of Basic Medical Sciences, Fudan University, Shanghai 200032, China;
3. Shanghai Institute of Liver Diseases, Shanghai 200032, China
乙型肝炎病毒(hepatitis B virus,HBV) 感染迄今仍然对全球公共卫生产生巨大影响,并会造成巨大的社会经济负担。虽然大多数急性HBV感染病例会自发消退,但仍有大约2.96亿人患有慢性感染,并发展为慢性乙型病毒性肝炎(chronic hepatitis B, CHB)。CHB可进一步导致肝硬化(cirrhosis, Crhs)、失代偿性肝硬化(decompensated cirrhosis, DC) 和肝细胞癌(hepatocellular carcinoma,HCC)[1]。在全球范围内的肝硬化患者群体中,有42%的患者存在HBV感染情况。目前,病毒性肝炎在我国仍然是肝硬化和肝癌的主要病因[2]。因此,研究慢性乙型肝炎的病理机制、开发有效的抗病毒治疗策略具有重大的科学意义和社会意义。由于CHB发病机制较为复杂,适宜的HBV感染动物模型是机制研究的必要条件。
HBV属于嗜肝DNA病毒,完整的乙肝病毒呈颗粒状,可分为外壳和核心两部分。外壳由脂质双层和蛋白质组成,脂质双层内含有乙肝表面抗原(hepatitis B surface antigen,HBsAg)。核心分为外部的衣壳和内部双链DNA。其DNA基因组(relaxed circular DNA,rcDNA) 包含4个重叠的开放阅读框(open reading frames,ORFs),即S、C、P、X,这些ORFs分别编码前-S1、前-S2、HBsAg、HBcAg、HBeAg、病毒多聚酶及HBxAg7种蛋白质[3]。HBV因其严格的种属特异性,仅能感染人和黑猩猩等一些灵长类动物。小鼠具有和人类高度相似的遗传背景,且其繁殖力和快速成熟的特性使其成为科学研究中有效且经济的哺乳动物模型。近年来,国内外科学家们在小鼠模型上取得了极大的进展。目前,HBV小鼠模型主要包括HBV转基因小鼠模型、流体动力学尾静脉注射(hydrodynamic tail vein injections,HDI) 小鼠模型、腺相关病毒-乙型肝炎病毒(adeno-associated virus-hepatitis B virus,AAV-HBV) 转染小鼠模型、共价闭合环状DNA(covalently closed circular DNA,cccDNA) 替代小鼠模型以及人鼠嵌合肝脏小鼠模型[4]。然而,这些模型中的HBV持续感染大多类似于临床患者的免疫耐受阶段,难以解释一些涉及HBV相关疾病发生和发展等方面的重要科学问题,尤其是肝纤维化、肝硬化以及肝癌发生的病理过程等。
早期的研究报道,HBV基本核心启动子(basic core promoter, BCP) 区域位置的突变与急性重型肝炎相关[5]。Su等[6]在1.3倍拷贝的HBV基因组5’端BCP区引入C1766T和T1768A两个点突变,构建了HBV突变株转基因小鼠模型。该转基因小鼠的后代表现出慢性肝脏损伤的病理特征,包括慢性肝炎、自发性肝纤维化/硬化以及肿瘤发生。这与人类HBV慢性感染所致的疾病发生发展过程极为相似,是一个非常理想的研究HBV的疾病模型。本研究采用RNA测序技术(RNA-Seq),深入剖析了BCP特定突变的HBV转基因小鼠的基因网络动态变化,旨在为临床HBV感染所引发的慢性肝纤维化的早期诊断、药物筛选及新型生物标志物的发掘提供科学依据。
1 材料和方法 1.1 实验动物基于BCP区突变的HBV转基因小鼠由复旦大学基础医学院邓强教授组赠予,小鼠模型可自发形成肝纤维化以及肝癌。本研究以padw1.3质粒为模板,用重叠延伸聚合酶链式反应(polymerase chain reaction,PCR) 的方法,在基因组5’端的BCP区引入2个突变位点,分别是1 766位的C碱基突变为T碱基,1 768位的T碱基突变为A碱基,构建了padw1.3 mut质粒,具体构建程序参考已发表文献[6]。该转基因小鼠的构建和繁殖由上海南方模式生物科技有限公司协助。所有动物实验均遵循国家实验动物饲养与使用指南。通过PCR鉴定转基因小鼠的基因型,鉴定引物为:BCPF-test-F:5’-CTCGCCTCGCAGACGCAGAT-3’; BCPF-test-R:5’-TGATAAAACGCCGCAGACACAT-3’,扩增所用体系及反应条件具体参照已发表文献[6]。能扩增出1.2 kb条带的转基因小鼠为阳性鼠,归为转基因小鼠组(HBV-Tg组),结果为阴性的为对照组(WT组)。饲养至第12周处死小鼠并收集肝脏组织以及血清样本。
1.2 方法 1.2.1 样本采集用3% (w/w) 戊巴比妥钠溶液腹腔注射麻醉小鼠,摘除眼球留取静脉血,然后打开腹腔, 摘取肝脏并称重记录。切除部分肝脏使用10% (v/v) 福尔马林溶液浸泡固定,剩余肝脏暂存于-80 ℃冰箱。静脉血室温静置2 h后,使用离心机离心(4 ℃,3 000 r/min) 离心20 min并收集上层血清,部分分装放置于4 ℃保存,剩余血清在-80 ℃保存。
1.2.2 乙肝病毒载量(hepatitis B DNA,HBV-DNA) 定量检测使用乙型肝炎病毒核酸测定试剂盒(PCR-荧光探针法)(广州达安基因,DA0030)提取DNA;采用ABI Prism 7500(美国赛默飞)进行PCR扩增检测。
1.2.3 小鼠血清丙氨酸转氨酶(alanine transaminase,ALT) 和天冬氨酸氨基转移酶(aspartate transaminase,AST) 检测获得的小鼠血清使用生理盐水稀释5倍后,送至复旦大学附属中山医院检测中心,采用罗氏Cobas C 702仪器(瑞士)进行检测。
1.2.4 肝脏组织天狼猩红(sirius red)染色① 肝脏组织使用福尔马林固定;②切片常规脱蜡至水;③蒸馏水水洗1 min;④用天狼猩红-苦味酸染色液(上海瑞雨公司,Bry-0013)浸染30~60 min;⑤无水酒精快速洗涤3遍;⑥二甲苯透明;⑦中性树胶封片,通过显微镜(日本Nikon公司)观察肝组织胶原沉积情况。
1.2.5 RNA提取、文库构建和测序① RNA提取:将肝组织加入1 mL Trizol(invitrogen, 15596),并加入3粒钢珠,使用组织破碎仪(上海必横生物)振荡3~5次后移至离心管中;离心管中加入氯仿后室温放置5 min,离心后取上清液转移至新的离心管,加入异丙醇沉淀,最后使用75% (v/v) 乙醇清洗2遍得到总的RNA。②对RNA浓度和纯度进行检测,调整至适合的浓度用于后续检测。③Oligo dT的磁珠富集mRNA。④加入fragmentation buffer(美国New England Biolabs),将mRNA随机断裂成300 bp左右的小片段。⑤使用逆转录试剂盒(南京诺唯赞,RT101-01)反转合成cDNA。⑥加入End Repair Mix (美国New England Biolabs)将双链cDNA补成平末端,随后在3’末端加上一个A碱基,用于连接Y字形的接头。⑦使用T100 Thermal Cycler (美国BIO-RAD)进行扩增,纯化得到最终的文库。⑧使用上海美吉生物公司的Illumina NovaSeq 6000(美国Illumina) 上机测序。⑨利用HiSat2和StringTie软件提取数据并进行质量评估。
1.2.6 差异表达基因筛选差异表达分析使用的软件是DESeq2。显著差异表达基因的筛选标准为FC (Fold Change) > 2或 < 0.5,Padj < 0. 05,当一个基因同时满足这2个条件时,则视该基因为差异表达基因。
1.2.7 功能和通路富集分析及蛋白互作网络分析将筛选出的差异基因导入DAVID数据库进行KEGG (Kyoto Encyclopedia of Genes and Genomes) 通路富集以及GO (Gene Ontology) 富集分析,绘制得到富集分析结果的柱状图。蛋白质-蛋白质相互作用(protein-protein interaction,PPI) 通过Sting数据库以及Cytoscape软件绘制得到。
1.2.8 统计学分析采用GraphPad Prism10软件进行数据分析,数据以实验的平均值±均数标准误(standard error of mean,SEM) 表示,两组数据之间的统计分析采用非配对样本t检验,P < 0. 05表示差异有统计学意义。
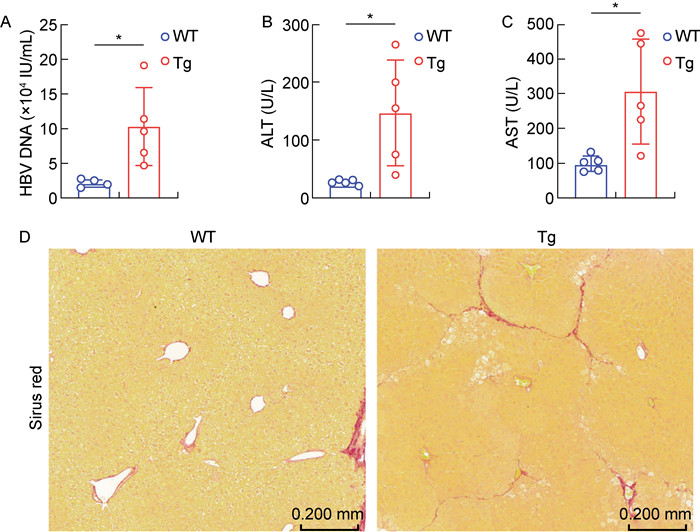
2 结果 2.1 血清学以及肝脏纤维化的比较与WT组小鼠相比,早期的BCP区突变的HBV转基因小鼠(HBV-Tg) HBV-DNA水平明显上升(见图 1A),且血清学的ALT以及AST水平均明显升高(见图 1B、C)。如图 1D的肝脏组织天狼猩红染色显示,WT组未见胶原沉积,HBV-Tg组汇管区及小叶间出现细线状的胶原沉积,但未见明显的桥接和假小叶形成。
|
|
A: Serum HBV-DNA in WT and Tg mice; B: Serum alanine aminotransferase (ALT); C: Serum aspartate transaminase (AST); D: Representative micrograph of sirius red staining of mouse liver tissue. Images were taken at original magnification (100×), scale bars = 0.200 mm. WT (Control, non-Tg WT mice, n=5), Tg (BCP-mutated HBV transgenic mice, n=5). All data were expressed as mean ± SEM. *P < 0.05. 图 1 血清学检测以及肝组织天狼猩红染色 Fig. 1 Serum liver function and sirius red staining of liver tissue |
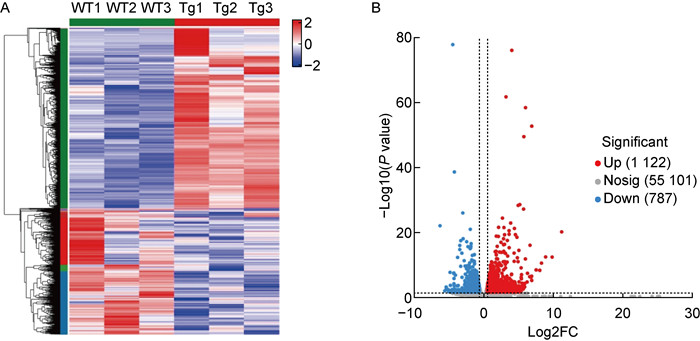
差异基因聚类分析热图显示,HBV-Tg组与WT组在转录水平上有明显的生物学异质性(见图 2A)。差异基因火山图分析显示,两组有1 104个差异基因(FC>2或 < 0.5,Padj < 0. 05),与WT相比,HBV-Tg组有729个基因表达上调,325个基因表达下调(见图 2B)。其中,Vmn2r3、Elfn1、S100 g、Cyp2a4,和Cyp2b9等基因表达明显上调,而Moxd1、Hmgb1-ps2、Dkk4等基因表达明显下调(见表 1)。
|
|
A: Heat map of differentially expressed genes in WT and BCP-mutated HBV transgenic mice; B: Differential gene volcano map of WT and BCP-mutated HBV transgenic mice. Red (up-regulated gene). Blue (down-regulated gene). Grey (no significant differenced genes). 图 2 肝组织的差异表达基因 Fig. 2 Differentially expressed genes in liver tissue |
| Gene name | Log2FC | P value | Regulate | Gene name | Log2FC | P value | Regulate | |
| Kansl2-ps | -5.570 469 511 | 2.48×10-2 | down | Vmn2r3 | 11.262 584 1 | 1.70×10-20 | up | |
| Moxd1 | -5.349 086 28 | 1.55×10-3 | down | Cyp2a4 | 9.898 148 171 | 9.37×10-13 | up | |
| Gpr137b-ps | -5.078 308 536 | 9.98×10-3 | down | S100 g | 8.957 266 289 | 8.39×10-13 | up | |
| Mid1-ps1 | -5.040 112 85 | 2.91×10-3 | down | Elfn1 | 8.361 923 53 | 6.88×10-11 | up | |
| Hmgb1-ps2 | -5.027 505 159 | 1.37×10-2 | down | Cyp2b9 | 8.117 414 135 | 1.11×10-8 | up | |
| Unc93a | -4.977 751 533 | 1.33×10-2 | down | Pappa2 | 7.595 240 451 | 8.39×10-9 | up | |
| Mfrp | -4.972 440 88 | 3.34×10-2 | down | Cyp2a22 | 6.966 004 461 | 4.99×10-53 | up | |
| Hsd3b9 | -4.784 503 542 | 4.65×10-8 | down | Kcna7 | 6.963 951 088 | 1.43×10-5 | up | |
| Kcnk10 | -4.738 727 755 | 4.77×10-4 | down | Sprr1a | 6.945 993 053 | 6.96×10-7 | up | |
| Dkk4 | -4.647 258 973 | 3.81×10-2 | down | Gpnmb | 6.712 358 025 | 2.15×10-16 | up | |
| Dnai1 | -4.500 638 087 | 9.96×10-3 | down | Areg | 6.669 526 834 | 1.05×10-6 | up | |
| Hsd3b5 | -4.402 606 814 | 4.05×10-78 | down | Nupr1 | 6.066 610 945 | 1.00×10-58 | up | |
| Hectd2os | -4.256 529 767 | 1.00×10-7 | down | Fgf21 | 6.051 553 914 | 3.55×10-12 | up | |
| Hsd3b4 | -4.240 788 459 | 7.20×10-8 | down | Ccdc85a | 5.948 693 77 | 1.44×10-3 | up | |
| Hsd3b8 | -4.240 788 459 | 7.20×10-8 | down | Adm2 | 5.948 179 469 | 1.19×10-4 | up | |
| Crybb3 | -4.227 532 801 | 1.49×10-4 | down | Vxn | 5.914 018 154 | 1.85×10-4 | up | |
| Serpina4-ps1 | -4.219 525 221 | 3.71×10-6 | down | Cyp2b13 | 5.828 709 021 | 9.20×10-2 | up | |
| Zfp600 | -4.195 046 265 | 6.08×10-3 | down | Bhlha15 | 5.822 949 523 | 8.34×10-50 | up | |
| Elovl3 | -4.164 465 666 | 6.01×10-39 | down | Hapln1 | 5.784 806 374 | 1.61×10-27 | up | |
| Vmn2r90 | -4.032 804 971 | 4.06×10-2 | down | Pi16 | 5.746 796 088 | 6.27×10-3 | up |
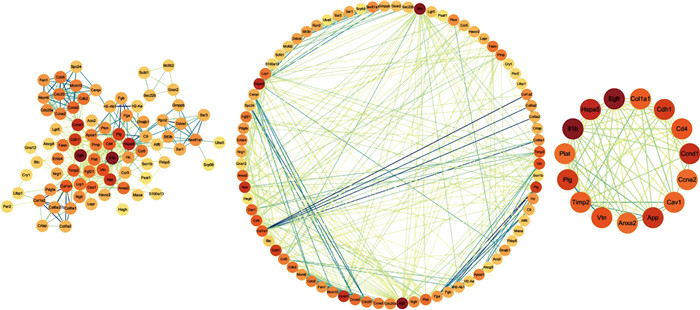
筛选出的差异基因通过String数据库构建蛋白网络互作图,通过MCODE挖掘出最有意义的板块,利用Degree值等综合排序筛选出最有意义的15个枢纽基因,分别是IL1β、Egfr、Hspa5、Ccnd1、App、Cdh1、Plg、Vtn、Cd4、Timp2、Col1a1、Cav1、Ccna2、Plat以及Anxa2 (见图 3)。
|
| 图 3 PPI网络构建及核心靶点的筛选过程 Fig. 3 PPI network construction and core target screening process |
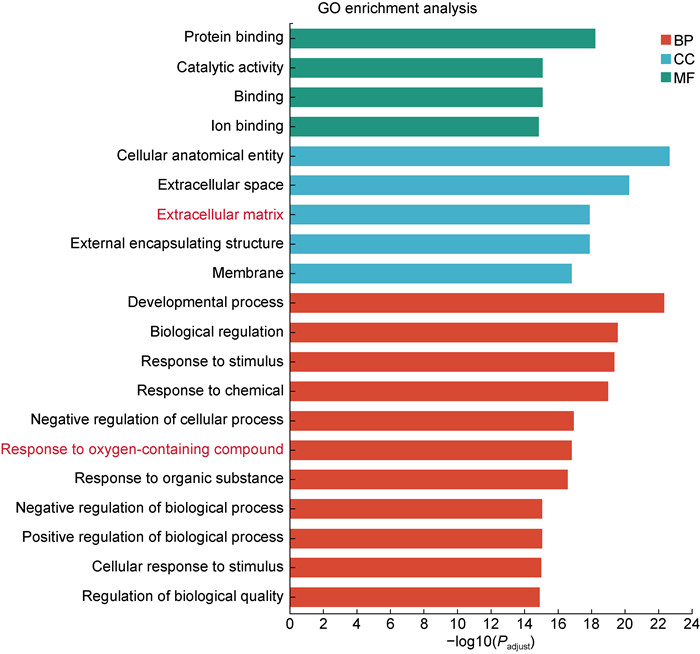
GO富集通路主要分为生物过程(biological process, BP)、细胞组分(cell component, CC) 和分子功能(molecular function, MF)。图 3展示了排名前20的GO富集通路,其中BP包括蛋白质结合、催化活性、离子结合等;CC包括细胞外基质、细胞外空间等;MF包括发育、对含氧化合物的反应等通路(见图 4)。
|
| Note: The ordinate denotes the gene ontology (GO) term, while the abscissa indicates the significance level of enrichment, corresponding to the column height. A smaller Padjust value results in a higher-log10 (Padjust) value, indicating a more significant enrichment of the GO term. BP (biological process), CC (cell component), MF (molecular function). 图 4 差异表达基因GO富集分析 Fig. 4 GO enrichment analysis of differentially expressed genes |
与WT小鼠相比,HBV-Tg小鼠的差异表达基因KEGG富集,排名前20的主要包括化学致癌-DNA加合物、肝癌、急性髓系白血病、化学致癌-受体活化、动脉粥样硬化、慢性髓系白血病、PPAR信号通路、卵巢类固醇生成、脂肪的消化与吸收、类固醇激素生物合成、视黄醇代谢、胆汁酸分泌、细胞色素P450、谷胱甘肽代谢、细胞周期、嘧啶代谢等通路(见图 5)。
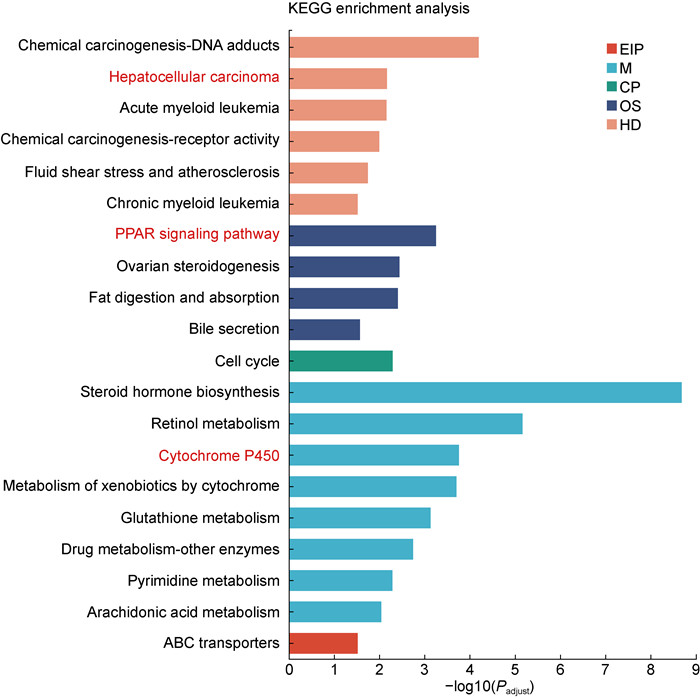
|
| Note: The ordinate denotes the KEGG pathway and the abscissa indicates the significance level of enrichment, corresponding to the height of the column. A smaller Padjustvalue results in a higher-log10 (Padjust) value, thereby indicating a more significant enrichment of the KEGG pathway. Environmental information processing (EIP), metabolism (M), cellular process (CP), biological system (OS), human disease (HD). 图 5 差异表达基因的KEGG分析 Fig. 5 KEGG analysis of differentially expressed genes |
该转基因小鼠可自发形成肝纤维化、肝硬化以及肝癌,因此通过对差异基因的进一步分析,筛选出与慢性肝病进展相关的特征性基因。如图 6A所示,参与细胞外基质合成的基因Col1a1、Col15a1、Col6a3、Col12a1、Acta2、Elfn1、Lum、Anxa2、Tgfb2、Sparcl1、Mfap4在HBV-Tg小鼠肝脏中的表达上调。正向调节细胞死亡相关的基因Gadd45a、Fkbp5、Ddit3、Myc、Slc7a11、Hrk、Ccng1、Eif2ak3、Tnfrsf10b、Chek1、Casp12、Casp6以及Bok在HBV-Tg小鼠中表达增加。热休克蛋白Hsp70家族的2个成员Hspa1a和Hspa1b对应激诱导的细胞凋亡起保护作用,在HBV-Tg小鼠肝脏中表达下调。Bcl2a1b和Bcl2a1d抗凋亡基因表达却上调(见图 6B)。此外,与WT组小鼠相比,HBV-Tg组中参与肝脏炎症调节的相关基因Ccl22、Ccl5、Ccr2、Cd14、Cd63、Cd8a、Il1r、Itgax、Lepr和Lgals3表达上调,而Nfkbia、Nlrp12、Cd4、Cxcl13以及Lbp表达下调(见图 6C)。
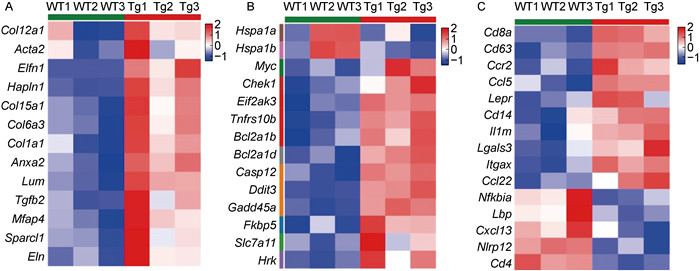
|
| A: The heat map of extracellular matrix differential gene; B: The heat map of cell death; C: The heat map of inflammatory response. 图 6 肝组织差异基因的表达水平 Fig. 6 Expression levels of differential genes in liver tissue |
HBV BCP区突变的转基因小鼠,实现了转基因小鼠中乙型肝炎核心抗原(hepatitis B core antigen,HBc)的高表达及持续性的肝损伤,且随着肝细胞的持续损伤,转基因小鼠积累相应的组织病理改变,最终演化和进展为终末期肝病,引发肝硬化及肝癌[6]。本研究主要观察12周龄的BCP区突变转基因小鼠,天狼猩红染色结果显示该转基因小鼠可自发形成纤维化。本研究首次对BCP区突变的转基因小鼠肝脏样本进行了转录组学分析,通过筛选出差异基因,发现了许多显著变化的GO功能(如细胞外基质)和KEGG通路富集(包括PPAR以及细胞色素P450通路等)。进一步通过PPI网络分析,筛选出前15的关键基因,主要涉及炎症反应(如IL1β、Cd4)、细胞周期(如Ccnd1、Ccna2)、细胞增殖与死亡(如Egfr、Hspa5)以及细胞外基质形成(如Timp2、Col1a1、Anxa2)。
慢性肝病的进展是持续性慢性肝损伤、慢性炎症和进行性纤维化相互关联、恶性循环驱动的结果,并且通常是一个长期的过程[7]。肝纤维化是由各类致病因子引发的肝内结缔组织异常增殖,是慢性肝病进展至肝硬化的中间环节及可逆阶段[7]。肝星状细胞(hepatic stellate cell,HSC) 的活化和转化被认为是肝纤维化发生和发展的2个主要原因[8]。过氧化物酶体增殖物激活受体(peroxisome proliferator-activated receptor,PPAR) 属于配体激活转录因子家族,在肝脏稳态、调节脂肪生成和抑制纤维生成中起关键作用[9]。PPAR家族的所有3种同型,包括α、β/δ和γ,都参与了肝星状细胞活化的调节[10]。研究发现,PPARα激动剂wy-14643、非诺贝特和油基乙醇酰胺可抑制肝纤维化的发展[11-12]。在三氧化二砷诱导的肝纤维化模型中,活化PPARα能够促进自噬,稳定HSC的细胞脂滴,从而抑制其活化[13]。此外,PPARα激活后还能抑制NF-κB的活性,从而降低炎症因子的水平,抑制肝脏炎症反应[14]。在CCl4小鼠模型中,激活PPARβ/δ也会增强肝纤维化表型[15],但其对HSC的作用仍具有争议。PPARγ对HSC活化的抑制可能涉及TGF-β/Smad和SMRT/HIF-1α途径[16-17]。有研究报道,PPARγ激动剂罗格列酮可抑制HBV的复制[18]。CYP450是肝酶系统的重要成员,在肝脏中负责代谢许多药物、毒素和内源性物质。CYP450可通过改变上游因子的转录和表达来调节全身代谢稳态,此外其还参与了活性氧(reactive oxygen species,ROS)生成、类固醇代谢、免疫炎症反应等途径。有研究表明,靶向CYP450可能是治疗调节肝脏脂质代谢等新兴疾病的一种潜在方法[19-20]。细胞色P450 2E1 (CYP2E1) 通过催化底物转化为更极性的代谢物并产生活性氧,在酒精和毒素代谢中起重要作用。活性氧诱导的氧化应激促进了肝细胞的损伤和死亡,进而诱导炎症、肝星状细胞活化和肝纤维化[21]。
近年来大量研究表明,肝纤维化的进展与细胞程序性死亡过程密切相关,如细胞凋亡、自噬、焦亡、坏死、铁死亡和泛凋亡[22]。通过差异基因筛选出与细胞死亡等的相关基因,如Myc、SLC7A11、Hrk、Bcl2a1b、Tnfrsf10b、Bcl2a1d、Casp12、Casp6、Bok以及Ddit3表达上调,而Hspa1b和Hspa1a表达下调。既往研究报道,caspase-12信号通路与胆汁淤积性肝纤维化和非酒精性肝纤维化的发生密切相关[23]。Myc是编码转录因子的调节基因和原癌基因家族,肝细胞中c-myc的过度表达促进了肝星状细胞的活化,从而促进肝纤维化的发生[24]。抑制xCT/SLC7A11可诱导肌成纤维肝星状细胞铁死亡,从而加重慢性肝损伤[25]。Caspase-6在小鼠和人NASH中被激活诱导了肝细胞损伤,进而促进了肝纤维化[26]。小鼠缺失Hspa1a/b导致活化的HSC消失增加,加速了肝纤维化的消退[27]。
慢性肝病(如病毒性肝炎或代谢相关脂肪肝病)的特征是持续炎症和随后的肝纤维化。在本研究中,转基因小鼠模型中与炎症反应相关的许多基因发生了改变,Ccl5、Cd63、Ccl22、Lgals3以及Il1rn等基因表达上调,而Lbp、Nlrp12和Cxcl13等基因表达下调。Ccl5在纤维化患者的肝脏中表达上调,干扰了Ccl5及其受体CCR5,可阻止HSC活化和纤维化[28]。Cd63通过抑制STAT3信号通路,发挥对肝癌发展的负性调控作用[29]。白细胞介素-1受体拮抗剂(IL-1 receptor antagonist, IL-1Rα)通过模型依赖的方式调节小鼠肝脏炎症和纤维化,在肝纤维化过程中表现出促纤维化和抗纤维化的双重作用。IL-1Rα在CCl4模型中能促进肝纤维化,而在BDL模型中具有保护作用[30]。NLRP12通过下调JNk依赖性炎症和肝细胞增殖来负向调控HCC[31]。
综上所述,本研究通过肝脏RNA-Seq检测发现的差异表达基因及其富集的通路与CHB的临床病理生理相符,尤其是涉及细胞外基质、细胞死亡、炎症、PPAR信号通路以及细胞色素P450等通路。因此,该模型可为HBV感染相关肝病的研究提供可靠的研究工具。
| [1] |
Seto WK, Lo YR, Pawlotsky JM, Yuen MF. Chronic hepatitis B virus infection[J]. Lancet, 2018, 392(10161): 2313-2324.
[DOI]
|
| [2] |
Huang DQ, Terrault NA, Tacke F, Gluud LL, Arrese M, Bugianesi E, Loomba R. Global epidemiology of cirrhosis-aetiology, trends and predictions[J]. Nat Rev Gastroenterol Hepatol, 2023, 20(6): 388-398.
[DOI]
|
| [3] |
Rajoriya N, Combet C, Zoulim F, Janssen HLA. How viral genetic variants and genotypes influence disease and treatment outcome of chronic hepatitis B. Time for an individualised approach?[J]. J Hepatol, 2017, 67(6): 1281-1297.
[DOI]
|
| [4] |
Liu Y, Maya S, Ploss A. Animal models of hepatitis B virus infection-success, challenges, and future directions[J]. 2021, 13(5): 777. doi: 10.3390/v13050777.
|
| [5] |
Liang TJ, Hasegawa K, Rimon N, Wands JR, Ben-Porath E. A hepatitis B virus mutant associated with an epidemic of fulminant hepatitis[J]. N Engl J Med, 1991, 324(24): 1705-1709.
[DOI]
|
| [6] |
Su Y, Bu F, Zhu Y, Yang L, Wu Q, Zheng Y, Zhao J, Yu L, Jiang N, Wang Y, Wu J, Xie Y, Zhang X, Gao Y, Lan K, Deng Q. Hepatitis B virus core protein as a Rab-GAP suppressor driving liver disease progression[J]. Sci Bull, 2024, 69(16): 2580-2595.
[DOI]
|
| [7] |
Parola M, Pinzani M. Liver fibrosis: pathophysiology, pathogenetic targets and clinical issues[J]. Mol Aspects Med, 2019, 65: 37-55.
[DOI]
|
| [8] |
Zhang C Y, Yuan WG, He P, Lei JH, Wang CX. Liver fibrosis and hepatic stellate cells: etiology, pathological hallmarks and therapeutic targets[J]. World J Gastroenterol, 2016, 22(48): 10512-10522.
[DOI]
|
| [9] |
Zardi EM, Navarini L, Sambataro G, Piccinni P, Sambataro FM, Spina C, Dobrina A. Hepatic PPARs: their role in liver physiology, fibrosis and treatment[J]. Curr Med Chem, 2013, 20(27): 3370-3396.
[DOI]
|
| [10] |
Pu S, Zhou H, Liu Y, Liu J, Guo Y, Zhou H. Roles of nuclear receptors in hepatic stellate cells[J]. Expert Rev Gastroenterol Hepatol, 2021, 15(8): 879-890.
[DOI]
|
| [11] |
Ip E, Farrell G, Hall P, Robertson G, Leclercq I. Administration of the potent PPARalpha agonist, Wy-14, 643, reverses nutritional fibrosis and steatohepatitis in mice[J]. Hepatology, 2004, 39(5): 1286-1296.
[DOI]
|
| [12] |
Toyama T, Nakamura H, Harano Y, Yamauchi N, Morita A, Kirishima T, Minami M, Itoh Y, Okanoue T. PPARalpha ligands activate antioxidant enzymes and suppress hepatic fibrosis in rats[J]. Biochem Biophys Res Commun, 2004, 324(2): 697-704.
[DOI]
|
| [13] |
Wang Z, Tao Y, Qiu T, Yao X, Jiang L, Wang N, Wei S, Jia X, Pei P, Yang G, Liu X, Liu S, Sun X. Taurine protected As(2)O(3)-induced the activation of hepatic stellate cells through inhibiting PPARα-autophagy pathway[J]. Chem Biol Interact, 2019, 300(2019): 123-130.
|
| [14] |
Li Q, Sun J, Mohammadtursun N, Wu J, Dong J, Li L. Curcumin inhibits cigarette smoke-induced inflammation via modulating the PPARγ-NF-κB signaling pathway[J]. Food Funct, 2019, 10(12): 7983-7994.
[DOI]
|
| [15] |
Kostadinova R, Montagner A, Gouranton E, Fleury S, Guillou H, Dombrowicz D, Desreumaux P, Wahli W. GW501516-activated PPARβ/δ promotes liver fibrosis via p38-JNK MAPK-induced hepatic stellate cell proliferation[J]. Cell Biosci, 2012, 2(1): 34.
[DOI]
|
| [16] |
Choi JH, Jin SW, Choi CY, Kim HG, Lee GH, Kim YA, Chung YC, Jeong HG. Capsaicin inhibits dimethylnitrosamine-induced hepatic fibrosis by inhibiting the TGF-β1/Smad pathway via peroxisome proliferator-activated receptor gamma activation[J]. J Agric Food Chem, 2017, 65(2): 317-326.
[DOI]
|
| [17] |
Zhang F, Lu S, He J, Jin H, Wang F, Wu L, Shao J, Chen A, Zheng S. Ligand activation of PPARγ by ligustrazine suppresses pericyte functions of hepatic stellate cells via SMRT-mediated transrepression of HIF-1α[J]. Theranostics, 2018, 8(3): 610-626.
[DOI]
|
| [18] |
Wakui Y, Inoue J, Ueno Y, Fukushima K, Kondo Y, Kakazu E, Obara N, Kimura O, Shimosegawa T. Inhibitory effect on hepatitis B virus in vitro by a peroxisome proliferator-activated receptor-gamma ligand, rosiglitazone[J]. Biochem Biophys Res Commun, 2010, 396(2): 508-514.
[DOI]
|
| [19] |
Jiang YJ, Cao YM, Cao YB, Yan TH, Jia CL, He P. A review: cytochrome P450 in alcoholic and non-alcoholic fatty liver disease[J]. Diabetes Metab Syndr Obes, 2024, 17: 1511-1521.
[DOI]
|
| [20] |
Zhang X, Li S, Zhou Y, Su W, Ruan X, Wang B, Zheng F, Warner M, Gustafsson J, Guan Y. Ablation of cytochrome P450 omega-hydroxylase 4A14 gene attenuates hepatic steatosis and fibrosis[J]. Proc Natl Acad Sci U S A, 2017, 114(12): 3181-3185.
[DOI]
|
| [21] |
Xu J, Ma HY, Liang S, Sun M, Karin G, Koyama Y, Hu R, Quehenberger O, Davidson NO, Dennis EA, Kisseleva T, Brenner DA. The role of human cytochrome P450 2E1 in liver inflammation and fibrosis[J]. Hepatol Commun, 2017, 1(10): 1043-1057.
[DOI]
|
| [22] |
Lu JL, Yu CX. Programmed cell death in hepatic fibrosis: current and perspectives[J]. Cell Death Discov, 2023, 9(1): 449.
[DOI]
|
| [23] |
Huang Y, Li X, Wang Y, Wang H, Huang C, Li J. Endoplasmic reticulum stress-induced hepatic stellate cell apoptosis through calcium-mediated JNK/P38 MAPK and Calpain/Caspase-12 pathways[J]. Mol Cell Biochem, 2014, 394(1/2): 1-12.
[DOI]
|
| [24] |
Nevzorova YA, Hu W, Cubero FJ, Haas U, Freimuth J, Tacke F, Trautwein C, Liedtke C. Overexpression of c-myc in hepatocytes promotes activation of hepatic stellate cells and facilitates the onset of liver fibrosis[J]. Biochim Biophys Acta, 2013, 1832(10): 1765-1775.
[DOI]
|
| [25] |
Du K, Oh SH, Dutta RK, Sun T, Yang WH, Chi JT, Diehl AM. Inhibiting xCT/SLC7A11 induces ferroptosis of myofibroblastic hepatic stellate cells but exacerbates chronic liver injury[J]. Liver Int, 2021, 41(9): 2214-2227.
[DOI]
|
| [26] |
Zhao P, Sun X, Chaggan C, Liao ZJ, Wong KI, He F, Singh S, Loomba R, Karin M, Karin JL, Saltiel AR. An AMPK-caspase-6 axis controls liver damage in nonalcoholic steatohepatitis[J]. Science, 2020, 367(6478): 652-660.
[DOI]
|
| [27] |
Yenari MA, Liu J, Zheng Z, Vexler ZS, Lee JE, Giffard RG. Antiapoptotic and anti-inflammatory mechanisms of heat-shock protein protection[J]. Ann N Y Acad Sci, 2005, 1053(1): 74-83.
[URI]
|
| [28] |
Kim BM, Abdelfattah AM, Vasan R, Fuchs BC, Choi MY. Hepatic stellate cells secrete Ccl5 to induce hepatocyte steatosis[J]. Sci Rep, 2018, 8(1): 10284.
[DOI]
|
| [29] |
Yu S, Chen J, Quan M, Li L, Li Y, Gao Y. CD63 negatively regulates hepatocellular carcinoma development through suppression of inflammatory cytokine-induced STAT3 activation[J]. J Cell Mol Med, 2021, 25(2): 1024-1034.
[DOI]
|
| [30] |
Meier RPH, Meyer J, Montanari E, Lacotte S, Balaphas A, Muller YD, Clément S, Negro F, Toso C, Morel P, Buhler LH. Interleukin-1 receptor antagonist modulates liver inflammation and fibrosis in mice in a model-dependent manner[J]. Int J Mol Sci, 2019, 20(6): 1295.
[DOI]
|
| [31] |
Udden SN, Kwak YT, Godfrey V, Khan MAW, Khan S, Loof N, Peng L, Zhu H, Zaki H. NLRP12 suppresses hepatocellular carcinoma via downregulation of cJun N-terminal kinase activation in the hepatocyte[J]. Elife, 2019, 16(8): e40396.
[DOI]
|
 2024, Vol. 19
2024, Vol. 19


