2. 上海市重大传染病与生物安全研究院, 上海 200032
2. Shanghai Institute of Infectious Disease and Biosecurity, Shanghai 200032, China
盖塔病毒(Getah virus,GETV)是一种蚊传虫媒病毒(mosquito-borne arbovirus),属于披膜病毒科(Togaviridae)甲病毒属(Alphavirus)。盖塔病毒粒子呈球形,直径为50~70 nm,表面由结构蛋白E1、E2构成的三聚体均匀分布在核衣壳构成的二十面体晶格中。核衣壳被囊膜包裹,内含单股正链RNA,基因组大小为11~12 kb。盖塔病毒基因组具有5’甲基化帽结构与3’Poly(A)尾结构,在5’和3’非编码区中间依次排列2个开放阅读框,分别编码4种非结构蛋白(nsP1、nsP2、nsP3和nsP4)以及5种结构蛋白(Capsid、E3、E2、6K和E1),其中结构蛋白的表达由位于2个阅读框中间的26S亚基因启动子调控[1]。自1955年首株盖塔病毒从库蚊中被分离以来,多个国家已分离得到盖塔病毒并报道相应的动物发病情况。在我国,盖塔病毒主要导致猪的死胎和繁殖障碍性综合征[2],同时也会引起马的发热、皮疹和水肿[3],并且近年来逐渐在牛等动物中流行[4],对养殖业造成了一定的经济损失。此外,在流行地区,未知原因发烧患者血清中亦能检测到盖塔病毒的特异性抗体[5]。因此,开展盖塔病毒的研究对公共健康具有重要意义。
在反向遗传学中,构建病毒感染性克隆通常指将病毒全长cDNA片段克隆到质粒载体中,以利用转染的方式在宿主细胞中扩增出目的病毒。在构建包含病毒全长cDNA序列的质粒时,对于启动子的选择通常有2种:一种是在病毒基因组5’UTR上游插入原核细胞启动子,如T7启动子,从而在试管中体外转录出病毒RNA并将其转染到宿主细胞中,也可以直接将质粒转染到瞬时或者稳定表达T7聚合酶的细胞中,进而直接转录出病毒RNA;另一种是使用真核细胞启动子,如CMV,直接转染质粒并利用细胞中的Ⅱ型RNA聚合酶(Pol Ⅱ)转录出病毒RNA[6]。尽管反向遗传学方法在病毒的拯救过程中得到广泛应用,但对于较长的基因序列,可能隐藏引起有害缺失和突变的转录、剪接或者终止信号。针对这些问题,常用的解决方法包括使用低拷贝质粒作为载体以及预测并突变病毒序列中潜在的有害位点等[7]。目前,通常将BHK-21细胞作为盖塔病毒拯救及后续病毒感染的靶细胞[8]。本研究利用反向遗传学系统拯救盖塔病毒的过程中,使用的模板是病毒cDNA,因此后续实验可以进一步基于盖塔病毒复制子构建盖塔病毒复制型表达载体。
复制子是指在RNA病毒基因组基础上经过改造得到的能够自我复制的RNA,通常在改造过程中仅删除结构蛋白部分,以确保复制子能够在宿主细胞中有效复制,但不会产生感染性病毒颗粒。基于甲病毒复制子结构,在5’非编码区前添加Pol II启动子,如CMV,在26S亚基因组启动子后使用外源基因替代原有结构蛋白基因的位置,最终构建甲病毒复制型表达载体[9]。自20世纪90年代,研究首次证明表达流感病毒核衣壳蛋白的载体能够诱导产生特异性抗体并引起T细胞应答以来,DNA疫苗的相关研究逐渐深入[10]。与经典的DNA疫苗载体相比,甲病毒复制型表达载体具有稳定携带外源基因的优势,并且能够在细胞中高效复制,在一定程度上提高外源基因的表达效率。已有部分研究表明,甲病毒复制型表达载体构建的复制型DNA疫苗具有激活免疫应答以及提供长期免疫保护的能力[11]。然而,目前常用于研究的疫苗载体大多是基于委内瑞拉马脑炎病毒(Venezuelan equine encephalitis virus,VEEV)减毒毒株TC-83株等甲病毒复制子的相关疫苗载体[12],几乎没有聚焦于盖塔病毒复制型表达载体的复制型DNA疫苗载体的研究。同时本研究拯救的盖塔病毒原毒株是从猪繁殖和呼吸障碍综合证减毒活疫苗中分离得到的毒力相对较弱的毒株,具有作为复制型DNA疫苗载体的潜力。除此之外,现有研究通常是在盖塔病毒结构基因的不同位置插入GFP等元件,进而获得携带报告基因的全长盖塔病毒[8, 13],针对盖塔病毒复制型表达载体的研究相对较少,因此本研究在成功构建盖塔病毒复制型表达载体后系统性评价其携带外源基因的表达效率,为构建盖塔病毒复制型DNA疫苗奠定了基础。
1 材料和方法 1.1 材料 1.1.1 细胞和质粒仓鼠肾成纤维细胞系BHK-21以及质粒pFK-ZL1、pFK-T7、pFK-CMV、pcDNA3.1-mCherry、pFK-ZL1-Rluc均由本实验室保存,TransStbl3化学感受态细胞购自北京全式金公司。
1.1.2 主要试剂DMEM培养基(Gibco,C11965500BT),胎牛血清(fetal bovine serum, FBS)(Gibco,A5669701),Lipofectamine 3000转染试剂(Invitrogen,L3000015),Opti-MEM(Gibco,31985070),DNA聚合酶试剂2×Phanta Max Master Mix (Vazyme,P515-02),限制性内切酶EcoRI-HF(NEB,R3101S)、ScaI-HF(NEB,R3122S)、SacI-HF(NEB,R3156S)、SpeI-HF(NEB,R3133S),T4 DNA连接酶(NEB,M0202S),mMESSAGE mMACHINE T7转录试剂盒(Invitrogen,AM1344),TRNzol Universal总RNA提取试剂(TIANGEN,DP424),E1抗体(GenTex,GTX637973),Capsid抗体(GenTex,GTX135183),Goat Anti-Rabbit IgG (H+L) HRP(圣尔,SB-AB0101),ECL化学发光超敏显色试剂盒(Yeasen,36208ES76),荧光素酶检测试剂盒Renilla Luciferase Assay System(Promega,E2810)。
1.2 方法 1.2.1 盖塔病毒序列合成根据GenBank登录号OM363683.1获取盖塔病毒BJ0304株全长序列,利用基因启动子转录起始位点预测在线软件(http://www.fruitfly.org/seq_tools/promoter.html)对序列进行细菌启动子的预测及沉默突变(见表 1),修饰后基于预测的合成难度,将序列分成3段交由上海捷瑞生物工程有限公司合成。
| Virus and E.coli promoter | Start | Virus sequence (5′-3′) | End | Viral protein | Score |
| 1WT | 452 | CAGACTTGGAATCCCCGACTTTTTGCCTGCATACTGACGAGACGTGCCGC | 497 | NS1 | 0.97 |
| Mutant | CAGACTTGGAATCCCCGACTTTTTGCCTGCACACTGACGAGACGTGCCGC | ND | |||
| 2WT | 595 | TATTGGATTGGATTCGACACCACTCCATTCATGTTCGAGGCACTAGCGGG | 640 | NS1 | 0.93 |
| Mutant | TATTGGATCGGATTCGACACCACTCCATTCATGTTCGAGGCACTAGCGGG | ND | |||
| 3WT | 1 340 | CGTTTAAAAGCCACAAAACCCACGCCATGTATAAGCGGCCTGAAACACAA | 1 385 | NS1 | 0.96 |
| Mutant | CGTTTAAAAGCCACAAAACCCACGCCATGTACAAGCGGCCTGAAACACAA | ND | |||
| 4WT | 2 299 | GCCAGTGGAAAGAAGGAGAACTGCCAAGAGATCATGAATGATGTAAAGAG | 2 344 | NS2 | 0.95 |
| Mutant | GCCAGCGGAAAGAAGGAGAACTGCCAAGAGATCATGAATGATGTAAAGAG | ND | |||
| 5WT | 2 537 | TCTTCAACTTGATGCAACTGAAGGTGCACTATAACCACAACATTTGCACA | 2 582 | NS2 | 0.94 |
| Mutant | TCTTCAACTTGATGCAACTGAAGGTGCACTACAACCACAACATTTGCACA | ND | |||
| 6WT | 3 841 | CTGCTGATGAGAGCATATGGTTACGCAGATAGAGTCAGCGAGATGGTGGT | 3 886 | NS2 | 0.95 |
| Mutant | CTGCTGATGAGAGCATATGGTTACGCAGACAGAGTCAGCGAGATGGTGGT | ND | |||
| 7WT | 3 975 | TTCTAACTTTGATAACGGCAGAAGAGCGGTAACCTTGCACCAAGCTAACC | 4 020 | NS2 | 0.93 |
| Mutant | TTCTAACTTTGATAACGGCAGAAGAGCGGTCACCTTGCACCAAGCTAACC | ND | |||
| 8WT | 5 868 | ATTAATTGTAGAAAGATTACGCGAAGGAGCAAAGTTGTACCTCCATGAGC | 5 913 | NS4 | 0.97 |
| Mutant | ATTAATCGTAGAAAGATTACGCGAAGGAGCAAAGTTGTACCTCCATGAGC | ND | |||
| 9WT | 7 296 | AAAAGATGAAACAGTTAAGTGGTCCCGCGTAGGATTGAGAGAAGAATTAG | 7 341 | NS4 | 0.90 |
| Mutant | AAAAGACGAAACAGTTAAGTGGTCCCGCGTAGGATTGAGAGAAGAATTAG | ND | |||
| 10WT | 7 944 | AAAGTGATGAAGCCGGCACACGTCAAAGGTGTGATCGACAACCCCGACCT | 7 989 | Capsid | 0.91 |
| Mutant | AAAGTGATGAAGCCGGCACACGTCAAAGGCGTGATCGACAACCCCGACCT | ND | |||
| 11WT | 8 337 | GCCTTGATGATGTGCGTCTTAGCCAACGTTACATTCCCATGCTCAGAGCC | 8 382 | E3 | 0.96 |
| Mutant | GCCTTGATGATGTGCGTCTTAGCCAACGTCACATTCCCATGCTCAGAGCC | ND | |||
| 12WT | 10 067 | AGTACTTGGAACCAGCTTGGAACCCACGCTAAACTTAGAGTACATAACCT | 10 112 | E1 | 0.97 |
| Mutant | AGTACTCGGAACCAGCTTGGAACCCACGCTAAACTTAGAGTACATAACCT | ND | |||
| 注:红色碱基标识沉默突变的碱基位置;WT:wild type;ND:not detectable。 | |||||
参照优化后的盖塔病毒序列及质粒pFK-T7、pFK-CMV、pcDNA3.1-mCherry、pFK-ZL1-Rluc序列,利用SnapGene软件设计引物(见表 2),引物由北京擎科生物科技股份有限公司合成。
| Primers | Sequences (5′-3′) | Amplified fragments | Product size (bp) |
| F1 | AAAAACGCCAGCAACGCGAGCTTAATACGACTCACTATAGATGGCGG | Fragment (T7) | 5 092 |
| R1 | CTGTATGTACTTTCTGGGACTTACTAGTGACGGGACGGTC | ||
| F1′ | GCAGAGCTCTCTGGCTAACTATGGCGGACGTGTGAC | Fragment 1′(CMV) | 5 072 |
| R1′ | CTGTATGTACTTTCTGGGACTTACTAGTGACGGGACGGTC | ||
| F2 | GACCGTCCCGTCACTAGTAAGTCCCAGAAAGTACATACAG | Fragment 2 | 5 417 |
| R2 | GTGTGCTCCCCGTTGAC | ||
| F3 | CTCAATCAGACAACAACGGCG | Fragment 3 | 1 333 |
| R3 | CTCTTTTTTTTTTTTTTTTTTTTTTTTTTTTTGTAAAATATTAAAAAAACAAATTAGACGCCTACG | ||
| F4 | GAGGGGAATTCCTCGAGG | pFK Vector 1(T7) | 1 833 |
| R4 | CTATAGTGAGTCGTATTAAGCTCGC | ||
| F4′ | GAGGGGAATTCCTCGAGG | pFK Vector 1′ (CMV) | 2 568 |
| R4′ | TGATGTCACACGTCCGCCATAGTTAGCCAGAGA~GCTCTGC | ||
| F5 | GGAAGTAAAGATCATCGATGCCAC | Fragment 4 | 423 |
| R5 | CATGGGCCCGGTGTCTGTAATACGTGGTCTTTAG | ||
| F6 | TTACAGACACCGGGCCCATGGTGAGCAAGGGCG | mCherry | 745 |
| R6 | AAGCCTCCCGGACGCGTTTACTTGTACAGCTCGT~CCATGC | ||
| F6′ | TTACAGACACCGGGCCCATGACGTCAAAAGTTTACGATCCAG | Rluc | 970 |
| R6′ | AAGCCTCCCGGACGCGTTTATTGTTCATTTTTGAGAACTCGCTC | ||
| F7 | TAAACGCGTCCGGGAGGCTTGACATAATG | Fragment 5 | 479 |
| R7 | TTCGTCTTCAAGAATTAATTCCCCTCGAGGAATTCCCCTC | ||
| F8 | GCAGAGCTCTCTGGCTAACTATGGTGAGCAAGGGCG | mCherry | 768 |
| R8 | TTCCCCTCTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTACTTGTACAGCTCGTCCATG | ||
| F8′ | GCAGAGCTCTCTGGCTAACTATGACGTCAAAAGTTTACGATCCAG | Rluc | 988 |
| R8′ | CTCTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTATTGTTCATTTTTGAGAACTCGCTC | ||
| F9 | AAAAAAAAGAGGGGAATTCCTCGAGG | pFK Vector2 | 2 556 |
| R9 | GCAGAGCTCTCTGGCTAACT |
以合成的3段盖塔病毒序列以及相关质粒为模版,通过1.2.2设计引物对相应模版设置聚合酶链式反应(polymerase chain reaction,PCR)程序(见表 3),扩增产物在1%(w/w)琼脂糖凝胶中120 V电泳30 min后胶回收。
| Steps | Temperature (℃) | Time | Cycles |
| Initial denaturation | 95 | 3 min | 1 |
| Denaturation | 95 | 15 s | |
| Annealing | 56 | 15 s | |
| Extension | 72 | 30 s/kb | 30 |
| Final extension | 72 | 5 min | 1 |
按照比例将1.2.3中回收的盖塔病毒3个片段克隆到对应的含有T7或者CMV启动子的载体上,转化TransStbl3化学感受态细胞,涂板后30 ℃培养18 h,挑菌提质粒酶切鉴定,鉴定正确的样品送北京擎科生物科技股份有限公司测序,最终质粒分别命名为T7-GETV和CMV-GETV。
1.2.5 盖塔病毒复制型表达载体构建在1.2.4构建的CMV-GETV基础上,删除病毒结构基因部分,利用ClaI和AbsI酶切位点获得载体骨架,通过1.2.2设计引物扩增Fragment4、Fragment5以及报告基因mCherry/Rluc片段,将扩增的3片段利用引物F5、R7进行overlap PCR,再用ClaI和AbsI内切酶切出末端,分别将含有2种报告基因的酶切片段与载体骨架用T4 DNA连接酶16 ℃过夜连接,后续鉴定操作与1.2.4相同, 最终质粒命名为pFK-GETV-mCherry和pFK-GETV-Rluc。
1.2.6 体外转录获得盖塔病毒RNA利用AbsI酶切位点将1.2.4构建的T7-GETV线性化,通过酚氯仿法回收线性化质粒,根据mMESSAGE mMACHINE T7转录试剂盒说明书进行体外转录。
1.2.7 盖塔病毒的拯救24孔板每孔铺1.5×105BHK-21细胞过夜培养,第2天待汇合度80%左右按照Lipofectamine3000转染试剂说明书进行转染,4 h后换含3%血清的培养基继续培养,每天观察转染细胞的形态变化,分别收集24、48、72、96、120 h的上清液,3000 r/min离心2 min,并过0.22 μm滤膜后于-80 ℃冻存,每个时间点设置4个重复。
1.2.8 TCID50法测定病毒滴度96孔板每孔铺2×104 BHK-21细胞过夜培养,第2天待汇合度70%左右,将1.2.7收集到的每个时间点的3个重复分别用含3%血清的培养基10倍稀释,96孔板细胞用1×PBS洗涤1遍,选择合适的稀释区间,每孔加入100 μL稀释液,每个稀释度设置8个重复,置于37 ℃,5%(v/v)CO2培养箱90 min,用1×PBS洗涤一遍,更换新鲜的含3%血清培养基。待48~72 h后,光镜下观察每个稀释度BHK-21细胞CPE(细胞皱缩、变圆、聚团),根据Reed-Muench方法对不同时间点上清液中的病毒滴度进行计算。将72 h时间点第4个重复样置于装有汇合度70%左右的BHK-21细胞的10 cm皿中扩大培养,待90%左右细胞出现CPE(感染后48~72 h),收取细胞上清液,3 000 r/min离心2 min,过0.22 μm滤膜后-80 ℃分装冻存,取其中一管冻融一次后的病毒液按照上述TCID50法测定病毒滴度。
1.2.9 盖塔病毒感染24孔板每孔铺1×105 BHK-21细胞过夜培养,第2天待汇合度70%左右,用1×PBS洗涤一遍,每孔按照感染复数(multi~plicity of infection,MOI)为0.1加入200 μL无血清培养基稀释的病毒液,置于37 ℃,5%CO2培养箱90 min,用1×PBS洗涤一遍,更换新鲜的含3%血清培养基继续培养。
1.2.10 一步生长曲线绘制按照1.2.9进行盖塔病毒感染,设置6、12、24、36、48、60 h 6个时间点,根据1.2.8,分别收取各个时间点上清液进行离心过滤处理并冻融一次后,用TCID50法测定病毒滴度,绘制一步生长曲线,每个时间点设置3个重复。
1.2.11 实时荧光定量PCR对于提取细胞中的RNA,24孔板每孔加500 μL TRNzol Universal总RNA提取试剂;对于提取上清液中的RNA,每孔吸取上清液3 000 r/min离心2 min,取200 μL上清加入600 μL TRNzol Universal总RNA提取试剂。对提取的RNA进行基因组DNA去除以及反转录,之后利用PCR引物(见表 4)进行实时荧光定量PCR,其中96孔板中每孔用20 μL体系,384孔板每孔用10 μL体系。
| GETV-nsp2 | GETV-E2 | GAPDH | |
| Forward primer | CGGCAACACCACTACCAACAATG | AGCGACAAGACTATCAATTCGT | GAAGGCCGGGGCCCACTTGA |
| Reverse primer | CACCACCATCTCGCTGACTCTG | TGCACTTTACCTTTGCGAGAC | CTGGGTGGCAG~ TGATGGCATGG |
24孔板中每孔细胞样品加入200 μL 1×loading buffer,静置5 min后收集到1.5 mL EP管中,短暂离心,95 ℃金属浴变性30 min,每孔上样10 μL,90 V电泳。PVDF膜浸在甲醇溶液中5 min,配制1×转膜液并4 ℃预冷,组装三明治夹,90 V湿转80 min。将膜标记后浸泡在5%脱脂奶粉中并置于55 r/min左右摇床上封闭1 h,1×PBST洗涤一次后裁剪合适大小区间的条带,加入一抗稀释液4 ℃过夜孵育。次日回收抗体并置于120 r/min左右摇床用1×PBST洗涤3次,每次10 min。根据种属置于55 r/min左右摇床上室温孵育二抗1 h,之后用相同的方法洗涤3次,配制显色液曝光。
1.2.13 荧光素酶活性检测24孔板每孔铺2×105 293T细胞过夜培养,第2天待汇合度80%左右按照Lipofectamine 3000转染试剂说明书进行转染;对照组中按照每孔与实验组相同摩尔量的DNA进行转染,同时P3000的量按照每500 ng DNA加入1 μL的比例进行缩减,其他试剂用量与操作均同1.2.7一致。于不同时间点收样,每个时间点设置3个重复。24孔板每孔加入100 μL 1×Passive Lysis Buffer,震荡充分裂解15 min后12 000 r/min离心15 s,收集上清液。根据荧光素酶检测试剂盒Renilla Luciferase Assay System说明书,将100×底物用Renilla Luciferase Assay Buffer稀释后,每个样品取10 μL加入50 μL稀释的底物中,混匀后置于测量仪器中读取相应数值并换算成实际荧光数值。
1.3 统计学分析使用GraphpadPrism8软件进行统计学分析和绘图,利用one-way ANOVA分析进行组间比较,P < 0.05被认为具有统计学差异。
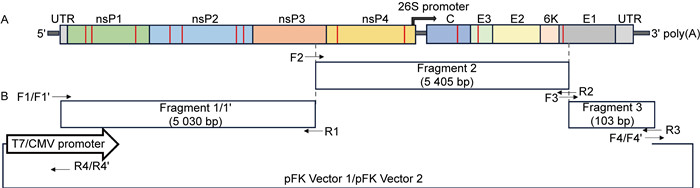
2 结果 2.1 成功构建携带T7和CMV启动子的GETV全长cDNA克隆根据GenBank登录号(OM363683.1)获取盖塔病毒BJ0304株全长序列。由于病毒基因组序列较长(11 689 bp),为降低细菌毒性(bacterial toxicity)对克隆的影响[14],参考本课题组之前拯救寨卡病毒的经验[15],对病毒全长序列进行原核启动子预测并合理修改。同时,为了提高合成效率,并考虑到合成的难度,将基因序列分为3段合成(见图 1A)。随后采用同源重组的方法直接将扩增的病毒3个片段克隆到实验室保存的分别携带T7和CMV启动子的2种pFK质粒pFK-T7和pFK-CMV上(见图 1B),构建成功后分别命名为T7-GETV和CMV-GETV。由于所构建的质粒是低拷贝质粒,这在一定程度上降低了病毒碱基突变的概率。
|
|
A: Genome structure of GETV BJ0304. The red line indicates the positions of the bases modified in the sequence to eliminate the prokaryotic promoters. B: Strategy for invitro assembly of an infectious cDNA clone of GETV BJ0304. 图 1 盖塔病毒毒株BJ0304全长感染性克隆的构建框架 Fig. 1 Schematic of the construction of full-length infectious clone of GETV BJ0304 strain |
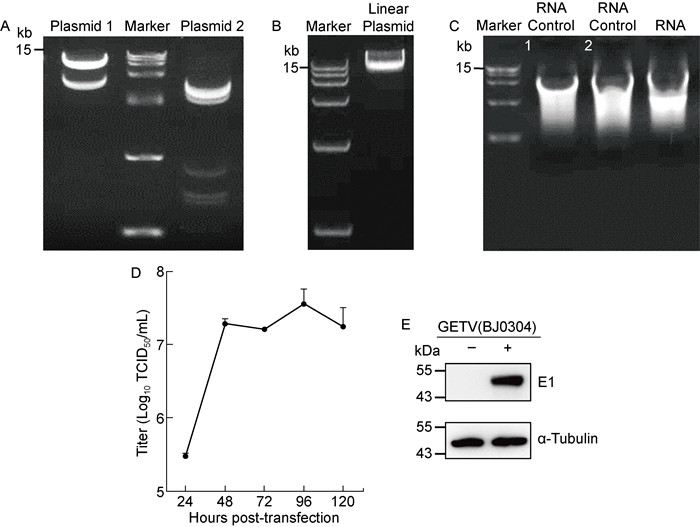
成功构建了分别携带CMV(Plasmid1)及T7(Plasmid2)启动子的T7-GETV和CMV-GETV质粒后,分别用EcoRI、ScaI内切酶进行酶切验证(见图 2A),并且送样测序以确保质粒的准确性。在确认质粒鉴定正确后,将10 μg的T7-GETV质粒进行线性化处理(见图 2B),并取其中1 μg进行体外转录,以寨卡病毒体外转录RNA为对照(见图 2C),成功得到盖塔病毒RNA。待24孔板BHK-21细胞汇合度达80%左右,利用lipo3000试剂转染,每孔转染500 ng体外转录得到的盖塔病毒RNA,在转染4 h后更换新鲜培养基,并连续5天每天收取上清液做TCID50检测(见图 2D)。结果显示,转染后24~48 h内盖塔病毒大量扩增,转染后48~96 h内病毒感染力水平相对稳定。此外,收集转染后72 h细胞样品进行WB实验,可以检测到病毒蛋白E1表达(见图 2E),综上说明盖塔病毒被成功拯救。
|
|
A: Analysis of restriction enzyme digestion on a 1% agarose gel. Plasmid 1: pFK-GETV plasmid carrying the T7 promoter digested with EcoRI; Plasmid 2: pFK-GETV plasmid carrying the CMV promoter digested with ScaI; Marker: DL15 000 DNA Marker. B: Analysis of linearized GETV infectious clone BJ0304 on a 1% agarose gel. C: Analysis of in vitro transcript on a 1% agarose gel. RNA Control 1: in vitro transcript from ZIKV infectious clone pZL1 stored at-80 ℃ as a control; RNA Control 2: in vitro transcript from ZIKV infectious clone pZL1; RNA: in vitro transcript from GETV infectious clone BJ0304. D: The titers of rescued BJ0304 at different time points determined using TCID50 assay. The results are presented as the mean ± SD (n=3). E: Western blotting analysis of expression of rescued BJ0304 E1 protein. Viral E1 protein was detected at 3 days post transfection. 图 2 盖塔病毒BJ0304毒株的拯救 Fig. 2 Rescue of the GETV BJ0304 strain |
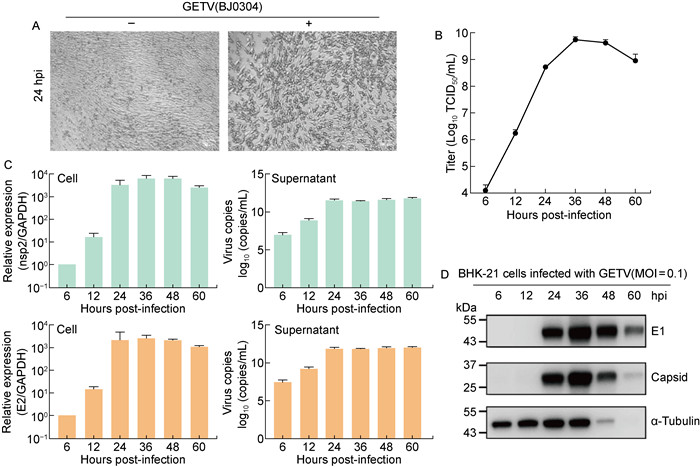
对转染72 h后的病毒液进行扩增,收取子代病毒液测定冻融一次后的TCID50,按照MOI=0.1感染24孔板中汇合度70%左右的BHK-21细胞。感染24 h后观察到细胞有明显的皱缩、变圆及聚集现象(见图 3A)。取感染后6、12、24、36、48和60 h 6个时间点绘制病毒的一步生长曲线(见图 3B)。设计针对非结构蛋白nsP2和结构蛋白E2的特异性引物,通过相对定量和绝对定量检测感染后病毒组分的转录水平变化(见图 3C)。结果显示,在病毒感染宿主细胞的过程中,病毒感染力和转录水平随时间的变化趋势大致相同,在24~48 h内盖塔病毒的滴度以及病毒RNA转录水平达到最高,同时在对应时间点检测到病毒蛋白E1、Capsid的表达(见图 3D)。
|
|
A: Cytopathic effect (CPE) in BHK-21 cells infected with the rescued GETV BJ0304 strain (MOI=0.1) at 24 hpi (hours post-infection). B: The titers of BJ0304 at different time points when BHK-21 cells were infected with GETV (MOI=0.1). The titer of BJ0304 was determined using TCID50 assay and the results are presented as the mean ± SD (n=3). C: The RNA replication of BJ0304 at different time points when BHK-21 cells were infected with GETV (MOI=0.1). The culture supernatants or cells were harvested and the RNA replication was detected by examining the transcription level of nsp2 or E2 using RT-qPCR. The results are presented as the mean ± SD (n=3). D: Western blotting analysis of expression of BJ0304 Capsid and E1 protein at different time points when BHK-21 cells were infected with GETV (MOI=0.1). 图 3 盖塔病毒BJ0304毒株在BHK-21细胞中生长特性的检测 Fig. 3 Identification of GETV BJ0304 strain grown in BHK-21 cells |
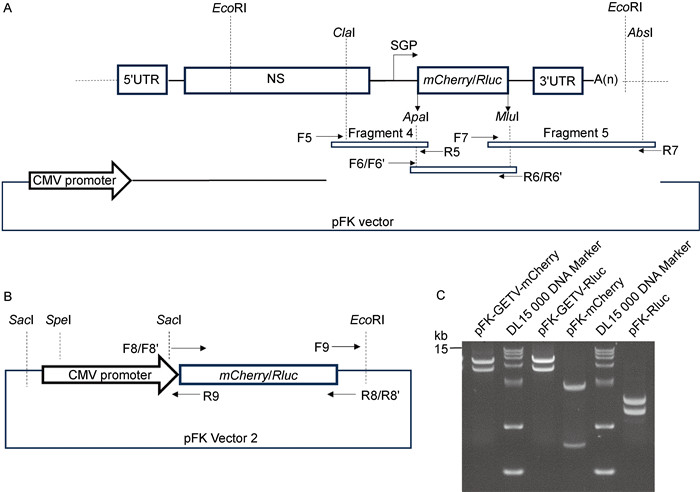
盖塔病毒拯救成功后,尝试在CMV-GETV质粒基础上构建携带报告基因mCherry或Rluc的GETV复制型表达载体(见图 4A)。利用酶切位点ClaI和AbsI删除结构蛋白部分,构建质粒载体。随后分别用引物对F5/R5和F7/R7扩增结构蛋白基因前后4、5片段,再分别利用引物对F6/R6和F6’/R6’扩增mCherry或Rluc片段,通过Overlap PCR将4、5片段分别与mCherry和Rluc片段连接, 形成一个完整的片段。随后用ClaI、AbsI内切酶对片段进行双酶切,最后与质粒载体进行T4连接,构建携带报告基因的复制型表达载体,分别命名为pFK-GETV-mCherry和pFK-GETV-Rluc。同时,删除盖塔病毒全序列,并在CMV-pFK载体上直接插入mCherry或者Rluc片段,以期作为验证复制子效率的对照质粒,分别命名为pFK-mCherry和pFK-Rluc (见图 4B)。最后,分别利用EcoRI、SacI和SpeI内切酶对构建的4种重组质粒进行酶切验证(见图 4C)并且送样测序。结果显示,成功构建了基于盖塔病毒复制子的复制型表达载体以及对照体系。
|
|
A: Strategy for in vitro assembly of the pFK-GETV-mCherry or pFK-GETV-Rluc plasmid. The entire structure gene is replaced by mCherry or Rluc gene. B: Strategy for in vitro assembly of the pFK-mCherry or pFK-Rluc plasmid. C: Analysis of restriction enzyme digestion on a 1% agarose gel. 图 4 盖塔病毒复制型表达载体和对照质粒构建示意图 Fig. 4 Schematic of the construction of GETV replicative expression vectors and control plasmids |
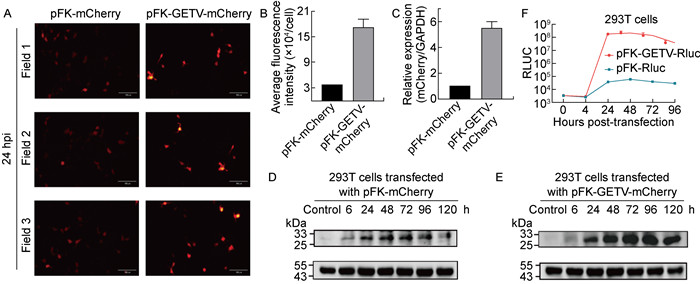
待24孔板293T细胞汇合度达到80%左右时,利用lipo3000等摩尔转染对照质粒和携带相应报告基因的复制型表达载体质粒。相同曝光条件下的结果显示,由于质粒大小等因素的影响,在相同时间点,对照组转染效率明显更高(见图 5A)。然而,转染效率的差异并不能充分反映基因表达水平的差异。进一步的单细胞平均荧光强度分析表明,转染24 h后,转染pFK-GETV-mCherry细胞的单细胞平均荧光强度大约是转染pFK-mCherry细胞的5倍(见图 5B),这与RT-qPCR测定的两组在相同时间点mCherry基因转录水平的差异基本一致(见图 5C)。进一步比较对照组和实验组mCherry蛋白表达水平随时间的变化,结果显示与对照组相比,在相同时间点转染pFK-GETV-mCherry组的细胞mCherry蛋白表达水平更高。而且一直持续到120 h,实验组仍有较高的蛋白表达水平(见图 5D~5E)。同时,荧光素酶的检测发现,在24 h时,盖塔病毒复制型表达载体表达报告基因Rluc的效率超过对照体系近1 000倍(见图 5F)。综上所述,复制型表达载体中外源基因的表达效率相对较高。
|
|
A: Fluorescence levels of pFK-mCherry and pFK-GETV-mCherry-transfected 293T cells at 24 hpi (hours post-infection). The results are demonstrated by selecting three different fields of view. B: Average fluorescence intensity per cell of pFK-mCherry and pFK-GETV-mCherry-transfected 293T cells at 24 hpi. The average fluorescence intensity per cell of cells transfected with the two plasmids was analyzed with software Image J and the results are presented as the mean ± SD (n=3). C: Expression level of heterologous gene mCherry in pFK-GETV-mCherry. The RNA replication was detected by examining the transcription level of mCherry using RT-qPCR. The results are presented as the mean±SD (n=3). D-E: Western blotting analysis of expression of mCherry at different time points when 293T cells were transfected with pFK-mCherry or pFK-GETV-mCherry plasmids. F: Detection of renilla luciferase signals produced from pFK-Rluc and pFK-GETV-Rluc-transfected 293T cells at different time points. The results are presented as the mean ±SD (n=3). 图 5 盖塔病毒复制型表达载体与对照质粒表达报告基因水平的比较 Fig. 5 Comparison of reporter gene expression levels between GETV replicative expression vectors and control plasmids |
蚊传虫媒病毒是指通过蚊虫传播的病毒。蚊虫不仅为病毒提供扩增的场所,而且可以通过吸血行为将病毒传播到宿主动物体内。病毒在宿主中扩增后,可以通过蚊虫的吸血行为进一步传播,从而维持其在自然界中的生存[16]。在我国,由于蚊虫种类繁多且分布广泛,蚊传虫媒病毒对公共卫生安全构成了巨大威胁。目前,我国已经对寨卡病毒[17]、登革病毒[18]等黄病毒属蚊传虫媒病毒给予了足够重视,但是对于盖塔病毒的研究仍然相对较少。自1964年科学家在海南从库蚊中分离出中国首株盖塔病毒以来,该病毒毒株逐渐在多个省份被分离,目前盖塔病毒分布范围已经蔓延到我国15个省份[19]。2017年,研究人员首次从山东某蓝狐养殖场中检测到盖塔病毒,这也是全球范围内首次从蓝狐中分离得到盖塔病毒[20],盖塔病毒的宿主谱不断扩大的趋势应引起广泛的关注。因此,本研究利用反向遗传学的方法拯救盖塔病毒,为深入研究其结构、功能及致病机制等提供了有力的工具。
在拯救盖塔病毒时,本文选择低拷贝质粒pFK作为盖塔病毒全长cDNA序列的插入骨架,以提高感染性克隆的稳定性。同时,选择原核启动子T7作为盖塔病毒感染性克隆的启动子。虽然与使用真核启动子(如CMV启动子)相比,构建后须先经过体外转录获得病毒RNA才能继续将病毒RNA转染到宿主细胞中,步骤相对烦琐,但质粒DNA只有进入细胞核后才能转录生成病毒RNA[21],因此直接转染病毒RNA可以提高盖塔病毒拯救的成功率。
根据序列特征和血清学交叉反应的特点,甲病毒主要分为8个抗原复合组,其中盖塔病毒属于塞姆利基森林病毒复合组,该组中还包括基孔肯雅病毒、塞姆利基森林病毒和罗斯河病毒等病毒[22]。因此,在拯救盖塔病毒以及盖塔病毒感染BHK-21细胞的过程中,本研究尝试使用实验室已有的用于检测基孔肯雅病毒Capsid和E1蛋白表达的特异性抗体,作为检测盖塔病毒Capsid和E1蛋白表达的通用抗体。首先,利用SnapGene软件中的氨基酸序列比对工具进行比较,结果显示基孔肯雅病毒抗体对应病毒毒株的Capsid和E1蛋白与本研究中拯救盖塔病毒毒株的Capsid、E1蛋白的氨基酸相似度分别为79.55%和76.89%。随后的WB实验结果显示,在盖塔病毒Capsid和E1蛋白对应位置有相应的蛋白条带。与对照组相比,相应位置蛋白条带具有特异性,而且其条带的变化趋势与在盖塔病毒感染BHK-21细胞的过程中,细胞内病毒蛋白表达量随时间变化的趋势基本符合。因此,实验室已有的用于检测基孔肯雅病毒Capsid和E1蛋白表达的特异性抗体可作为检测盖塔病毒Capsid和E1蛋白表达的通用抗体。
本研究在构建盖塔病毒感染性克隆的同时,成功构建了基于盖塔病毒复制子的盖塔病毒复制型表达载体。常规的DNA疫苗主要存在抗原基因表达量低的问题,针对此瓶颈的一个重要解决方法就是构建复制型DNA疫苗。利用甲病毒复制子的结构和复制机制可以显著增加抗原基因mRNA拷贝量,进而提高抗原的表达效率[21],因此基于甲病毒复制子的复制型表达载体是目前较为理想的复制型DNA疫苗载体。除了DNA疫苗,基于甲病毒复制子的自我复制型mRNA疫苗(self-amplifying mRNA vaccine,SAM)也可以增加抗原基因的表达。本实验没有选择在含有T7启动子的盖塔病毒感染性克隆基础上进行改造从而获得自我复制型mRNA疫苗载体,一方面是因为基于甲病毒复制型载体构建的质粒型DNA疫苗的免疫效果已经得到验证[11];另一方面这样可以避免体外转录的操作,从而降低由RNA的降解可能导致的抗原表达效率下降的问题,并降低成本。本研究基于研究较为成熟的委内瑞拉马脑炎病毒减毒毒株TC-83株等甲病毒复制子的相关疫苗载体,从猪繁殖和呼吸障碍综合证减毒活疫苗中分离得到毒力相对较弱的盖塔病毒毒株,探索其是否具有作为复制型DNA疫苗载体的潜力。因此,评估盖塔病毒复制型表达载体表达外源基因的能力具有重要的意义。通过报告基因mCherry的荧光水平表征外源基因的表达能力,具有操作方便和通过荧光显微镜直接观察的优势;而利用报告基因Rluc的表达来进一步表征外源基因的表达能力,则使得检测更为灵敏,可以放大实验体系与对照体系之间的差异,从而能够较为直观地分析盖塔病毒复制型表达载体对外源基因的表达能力。通过分析报告基因mCherry的平均荧光强度和转录水平变化,并利用相关试剂盒测定报告基因Rluc的表达水平,结果显示盖塔病毒复制型表达载体的基因表达能力显著高于对照体系。
4 结论本研究成功构建了拯救盖塔病毒的反向遗传学系统,该系统允许在基因水平对盖塔病毒进行改造,为后续实验室探究盖塔病毒的结构、功能及致病机制等提供了有力的工具。同时,基于该系统拯救的盖塔病毒,能够较为完整地表征盖塔病毒在BHK-21细胞中的生长特征。在此过程中,还成功验证了实验室已有的基孔肯雅病毒Capsid和E1蛋白的特异性抗体作为检测盖塔病毒Capsid和E1蛋白表达通用抗体的可行性。更为重要的是,本研究成功构建了基于盖塔病毒复制子的盖塔病毒复制型表达载体。与对照体系报告基因mCherry或者Rluc的表达水平进行比较,结果表明盖塔病毒复制型表达载体的基因表达能力显著高于对照体系,这为研究盖塔病毒复制型DNA疫苗奠定了基础。
| [1] |
Gould EA, Coutard B, Malet H, Morin B, Jamal S, Weaver S, Gorbalenya A, Moureau G, Baronti C, Delogu I, Forrester N, Khasnatinov M, Gritsun T, De Lamballerie X, Canard B. Understanding the alphaviruses: recent research on important emerging pathogens and progress towards their control[J]. Antiviral Res, 2010, 87(2): 111-124.
[DOI]
|
| [2] |
Yang T, Li R, Hu Y, Yang L, Zhao D, Du L, Li J, Ge M, Yu X. An outbreak of Getah virus infection among pigs in China, 2017[J]. Transbound Emerg Dis, 2018, 65(3): 632-637.
[DOI]
|
| [3] |
Lu G, Ou J, Ji J, Ren Z, Hu X, Wang C, Li S. Emergence of Getah virus infection in horse with fever in China, 2018[J]. Front Microbiol, 2019, 10(1): 2601.
[DOI]
|
| [4] |
Liu H, Zhang X, Li LX, Shi N, Sun XT, Liu Q, Jin NY, Si XK. First isolation and characterization of Getah virus from cattle in northeastern China[J]. BMC Vet Res, 2019, 15(1): 320.
[DOI]
|
| [5] |
Li X, Qiu F, Yang H, Rao N, Calisher CH. Isolation of Getah virus from mosquitos collected on Hainan Island, China, and results of a serosurvey[J]. Southeast Asian J Trop Med Public Health, 1992, 23(4): 730-734.
[URI]
|
| [6] |
Almazan F, Sola I, Zuniga S, Marquez-Jurado S, Morales L, Becares M, Enjuanes L. Coronavirus reverse genetic systems: infectious clones and replicons[J]. Virus Res, 2014, 189(11): 262-270.
|
| [7] |
Aubry F, Nougairede A, Gould EA, De Lamballerie X. Flavivirus reverse genetic systems, construction techniques and applications: a historical perspective[J]. Antiviral Res, 2015, 114(2): 67-85.
[URI]
|
| [8] |
Ren TW, Min XL, Mo QR, Wang YX, Wang H, Chen Y, Ouyang K, Huang WJ, Wei ZZ. Construction and characterization of a full-length infectious clone of Getah virus in vivo[J]. Virologica Sinica, 2022, 37(3): 348-357.
[DOI]
|
| [9] |
Lundstrom K. Application of DNA replicons in gene therapy and vaccine development[J]. Pharmaceutics, 2023, 15(3): 947.
[DOI]
|
| [10] |
Ulmer JB, Donnelly JJ, Parker SE, Rhodes GH, Felgner PL, Dwarki VJ, Gromkowski SH, Deck RR, DeWitt CM, Friedman A, Hawe LA, Leander KR, Martinez D, Perry HC, Shiver JW, Montgomery DL, Liu MA. Heterologous protection against influenza by injection of DNA encoding a viral protein[J]. Science, 1933, 259(5102): 1745-1749.
[URI]
|
| [11] |
Tang J, Bi Z, Ding M, Yin D, Zhu J, Zhang L, Miao Q, Zhu Y, Wang G, Liu G. Immunization with a suicidal DNA vaccine expressing the E glycoprotein protects ducklings against duck Tembusu virus[J]. Virol J, 2018, 15(1): 140.
[DOI]
|
| [12] |
Johnson DM, Jokinen JD, Wang M, Pfeffer T, Tretyakova I, Carrion Jr. R, Griffiths A, Pushko P, Lukashevich IS. Bivalent Junin & Machupo experimental vaccine based on alphavirus RNA replicon vector[J]. Vaccine, 2020, 38(14): 2949-2959.
[DOI]
|
| [13] |
Ren TW, Zhou LS, Min XL, Sui MQ, Zhi XC, Mo YF, Huang J, Zhang K, Liu WB, Wang H, Wang XD, Ouyang K, Chen Y, Huang WJ, Wei ZZ. Development of a recombinant reporter Getah virus for antiviral drug screening assays[J]. Vet Microbiol, 2023, 281(6): 109742.
[DOI]
|
| [14] |
Pu SY, Wu RH, Yang CC, Jao TM, Tsai MH, Wang JC, Lin HM, Chao YS, Yueh A. Successful propagation of flavivirus infectious cDNAs by a novel method to reduce the cryptic bacterial promoter activity of virus genomes[J]. J Virol, 2011, 85(6): 2927-2941.
[DOI]
|
| [15] |
Zhao F, Xu Y, Lavillette D, Zhong J, Zou G, Long G. Negligible contribution of M2634V substitution to ZIKV pathogenesis in AG6 mice revealed by a bacterial promoter activity reduced infectious clone[J]. Sci Rep, 2018, 8(1): 10491.
[DOI]
|
| [16] |
Ruckert C, Ebel GD. How do virus-mosquito interactions lead to viral emergence?[J]. Trends Parasitol, 2018, 34(4): 310-321.
[DOI]
|
| [17] |
Shen Z, Li X, Lu Y, Li J, Xiao H, Yuan W, Zhang Z, Zhou Y, Feng Y, Qin W, Xia X. Nonindigenous case of Asian Zika virus lineage in Yunnan, China, 2019[J]. J Infect, 2019, 79(6): 612-625.
[DOI]
|
| [18] |
Sang S, Chen B, Wu H, Yang Z, Di B, Wang L, Tao X, Liu X, Liu Q. Dengue is still an imported disease in China: a case study in Guangzhou[J]. Infect Genet Evol, 2015, 32(6): 178-190.
[DOI]
|
| [19] |
Lu G, Chen R, Shao R, Dong N, Liu W, Li S. Getah virus: an increasing threat in China[J]. J Infect, 2020, 80(3): 350-371.
[DOI]
|
| [20] |
Netherton C, Rouiller I, Wileman T. Highly pathogenic swine Getah virus in blue foxes, Eastern China, 2017[J]. Emerg Infect Dis, 2019, 78(7): 3710-3721.
[URI]
|
| [21] |
Lundstrom K. Plasmid DNA-based Alphavirus vaccines[J]. Vaccines (Basel), 2019, 7(1): 29.
[DOI]
|
| [22] |
Powers AM, Brault AC, Shirako Y, Strauss EG, Kang W, Strauss JH, Weaver SC. Evolutionary relationships and systematics of the alphaviruses[J]. J Virol, 2001, 75(21): 10118-10131.
[DOI]
|
 2024, Vol. 19
2024, Vol. 19


