2. 上海交通大学医学院全球健康学院,上海 200025
2. Institute for Global Health, Shanghai Jiao Tong University School of Medicine, Shanghai 200025, China
以细菌为代表的原核生物,在地球上的存在可追溯到30多亿年前。以细菌为感染对象的噬菌体是最早的病毒形态,其历史被认为同在30亿年以上[1]。在地球生物圈里,大约有超过1031个噬菌体颗粒,每个细菌细胞内平均约有10个噬菌体[2]。人体细胞、噬菌体、细菌及其他微生物共同组成了人体内环境。因此,噬菌体与细菌的相互作用也持续影响着人体健康。
噬菌体自1915年被描述、1940年被电镜观察到开始,一直被认为是应对细菌感染的重要候选制剂。同时,研究噬菌体所获的成果在生命科学的多项重大发现中发挥了重要的支撑作用,如DNA为遗传物质的论证、基因组测序、噬菌体展示技术等[3]。21世纪以来,由于细菌对抗生素耐药情况不断加剧,传统抗生素之外的抗菌制剂研究受到越来越多的关注。噬菌体可随细菌分裂增殖,也可完全裂解细菌。与此同时,细菌也通过基因和表型的改变形成对抗噬菌体的机制,噬菌体针对细菌的对抗机制又形成自己的“抗细菌抗噬菌体机制”,循环往复,绵延不绝。本文从阻止噬菌体吸附、超感染排除(superinfection exclusion,Sie)、限制性修饰(restriction modification,RM)系统、CRISPR-Cas、流产感染(abortive infection,Abi)等方面对细菌抗噬菌体的机制以及噬菌体针对细菌抗性机制的应变进行综述,期望能为细菌耐药性及抗菌制剂的研究提供参考。
1 细菌抗噬菌体的机制细菌抗噬菌体的机制涉及噬菌体的感染、增殖、裂解过程(即噬菌体的生活周期)。主要机制为阻止噬菌体吸附、阻止噬菌体DNA进入细胞质、降解噬菌体的核酸、干扰噬菌体生活周期及阻止噬菌体颗粒释放、干扰噬菌体的组装等。
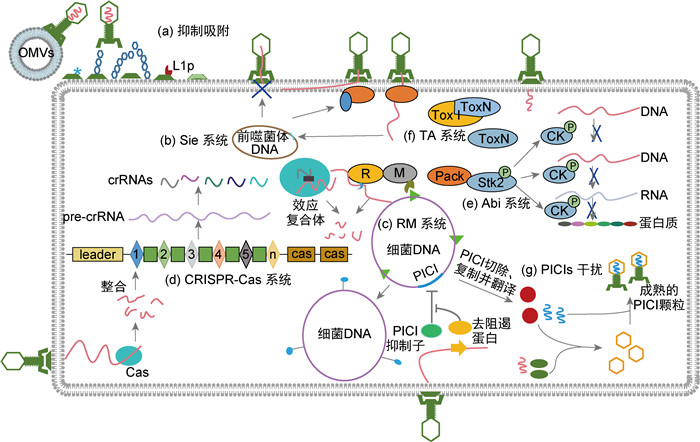
1.1 阻止噬菌体吸附噬菌体感染宿主的第一步是对宿主的吸附。一些细菌(如铜绿假单胞菌)通过阻止噬菌体吸附来对抗其感染(图 1a)[4]。细菌阻止噬菌体吸附的情况包括:①革兰阴性细菌分泌外膜囊泡(outer membrane vesicle,OMV),其含有可充当噬菌体受体的外膜蛋白,竞争性隔离细胞外噬菌体颗粒; ②噬菌体受体(如铜绿假单胞菌Ⅵ型菌毛)糖基化而隐藏,使噬菌体无法识别; ③噬菌体受体被表面结构(如多糖荚膜)屏蔽而无法被噬菌体吸附; ④噬菌体自身编码蛋白与其受体结合,阻止同类型噬菌体与细菌受体结合; ⑤噬菌体受体基因突变使噬菌体不能结合; ⑥噬菌体受体基因不表达,导致相应噬菌体无法感染。
|
| 图 1 细菌抗噬菌体的主要机制 Fig. 1 The main mechanism of bacteriophage resistance of bacteria |
Sie源自一种蛋白,它可阻止噬菌体DNA进入宿主细胞,从而赋予后者针对特定噬菌体的免疫力(图 1b)。这些蛋白多为膜锚定蛋白或与膜成分相关。编码这些蛋白质的基因通常存在于噬菌体中,因此Sie在噬菌体-噬菌体相互作用而非噬菌体-宿主相互作用中发挥重要作用。
以大肠埃希菌T4噬菌体为例,T4噬菌体具有2个Sie系统,分别由imm和sp编码。正常情况下,T4噬菌体感染大肠埃希菌时,细菌的肽聚糖层被其gp5编码的溶菌酶降解,内膜蛋白易位,DNA进入细胞质中。Sie的情况包括:①已进入裂解周期的T4噬菌体编码Imm蛋白,与另一种膜蛋白结合并改变注入部位的构象,阻断其他T4样噬菌体的DNA进入细胞质; ②T4噬菌体编码的Sp蛋白通过抑制噬菌体gp5编码的溶菌酶活性而阻止肽聚糖降解,使DNA停留在肽聚糖层与外膜之间[5]。
1.3 RM系统和CRISPR-Cas系统——降解噬菌体的核酸RM系统是一种普遍存在且极其多样化的抗噬菌体防御模式。它们通常由限制性核酸内切酶和甲基转移酶组成。限制性核酸内切酶识别短的DNA序列并切割噬菌体DNA。这些序列既存在于细菌宿主中,也存在于入侵的噬菌体中,宿主使用甲基转移酶修饰自己的DNA来避免其被限制性酶识别,从而保护自身基因组。入侵的噬菌体通常不会被甲基化,因此在侵入时会被切割。RM系统根据其作用机制和亚基组成分为4型。
以Ⅰ型RM系统为例(图 1c),Ⅰ型RM系统的组分为杂合寡聚复合物,通常包含2个DNA裂解所需的核酸酶亚基(R),以及1个指定识别DNA序列的特异性亚基和2个催化甲基化反应的甲基转移酶亚基(M)。这些酶识别其特定靶序列的甲基化状态。完全甲基化的DNA被认为是细菌基因组的一部分。半甲基化的DNA被识别为新复制的细菌DNA,甲基转移酶(M)会在辅因子的帮助下进一步甲基化修饰另一条链。入侵的噬菌体DNA通常缺乏特异性修饰,因此会被核酸酶(R)识别为外源DNA并被切割[6]。
CRISPR-Cas系统是目前广泛使用的基因编辑技术的最初来源(图 1d)。噬菌体初次入侵时,Cas1-Cas2复合体选择一部分外源DNA(原间隔序列)整合到宿主的CRISPR阵列中,成为间隔序列。间隔序列随后被转录为长的pre-crRNA,并被Cas蛋白或细胞RNase进一步加工为成熟的crRNA。当具有相同或非常相似的原间隔序列的噬菌体感染时,成熟的crRNA与Cas核酸酶组合成效应复合体,识别紧随原间隔序列后的PAM序列,然后Cas蛋白对与crRNA互补的入侵dsDNA进行切割[7]。
1.4 Abi系统和毒素-抗毒素系统——干扰噬菌体生活周期并阻止噬菌体颗粒释放Abi系统是细菌细胞为阻止噬菌体释放而自我牺牲的过程。它是一种“程序性细胞死亡”,是通过干扰细胞DNA生物合成的基本过程(如复制、转录和翻译)或诱导膜渗漏来实现的(图 1e)。以表皮葡萄球菌Stk2 Abi系统为例,在溶原性噬菌体进入裂解周期后,其表达的蛋白PacK会触发表皮葡萄球菌丝氨酸/苏氨酸激酶Stk2磷酸化。后者进而引起一系列涉及转录、翻译、复制和代谢的细胞因子磷酸化。这种广泛的磷酸化可能会破坏细菌基因组的复制、转录和翻译途径,最终导致噬菌体感染周期的流产和细胞死亡[8]。
毒素-抗毒素(toxin-antitoxin,TA)系统(图 1f)广泛存在于细菌、古细菌的染色体、质粒、噬菌体的基因组上,依靠一个毒素和一个对毒素有拮抗作用的抗毒素组成的二元系统行使防御功能。TA系统的基本特征是抗毒素的不稳定性,必须连续表达才能与毒素保持适当的化学计量比并中和毒素。TA系统中的毒素可作用于中心法则的多个过程,最终导致细菌生长停滞或休眠。因此,TA系统既可以作为休眠诱导系统也可以作为Abi系统,其结果取决于一系列因素,包括毒素的作用机制、毒素活性的持续时间及噬菌体的生命周期。以植物病原体黑胫病菌的质粒编码的Ⅲ型TA系统为例,毒素ToxN为内切RNase,抗毒素ToxI为RNA,毒素、抗毒素形成动态平衡。噬菌体感染可通过抑制ToxIN转录、防止ToxI折叠和破坏ToxI-ToxN结合而抑制抗毒素能力,最终导致游离ToxN释放。大量ToxN降解细胞RNA,导致细菌细胞及噬菌体死亡[9]。
1.5 干扰噬菌体组装噬菌体诱导性染色体岛(phage-inducible chromosomal islands,PICI)广泛存在于革兰阳性和革兰阴性细菌中,是高度可移动的遗传元件家族,对基因水平转移、宿主适应性和毒力产生具有重要贡献(图 1g)[10]。
以金黄色葡萄球菌致病岛(Staphylococcus aureus pathogenic island,SaPI)为例。在无辅助噬菌体裂解生长情况下,PICI的表达被PICI抑制子抑制而处于一种静止的前噬菌体状态。在辅助噬菌体处于裂解生长周期时,早期会表达一种去阻遏蛋白,导致PICI从宿主染色体上切除,其基因组得以复制并表达。PICI的表达产物可通过不同机制干扰辅助噬菌体的组装,如抑制辅助噬菌体晚期基因的表达,调节辅助噬菌体衣壳的装配,使其产生只能与较小的SaPI基因组包装在一起的衣壳。最终导致PICI基因组被优先包装并防止形成辅助噬菌体颗粒。与革兰阳性细菌不同,革兰阴性细菌的PICI由PICI编码的激活剂诱导,但激活剂的表达需要辅助噬菌体[10-11]。
2 噬菌体针对细菌抗性的应变在细菌产生针对噬菌体抗性机制的同时,噬菌体也相应发生一系列改变。目前发现噬菌体针对细菌抗性的改变有:适应新受体、应对RM系统、应对Abi系统、应对CRISPR-Cas系统以及溶原决策机制等。
2.1 适应新受体细菌通过改变噬菌体结合受体来抵御噬菌体的结合,噬菌体也可做出针对性的改变,从而结合新的受体(图 2a)。噬菌体依赖其尾部的受体结合蛋白(receptor binding protein,RBP)识别并结合细菌细胞膜上受体以完成吸附。当细菌细胞膜上受体改变时,噬菌体尾部RBP也发生相应改变,识别并结合新受体,再次吸附; 当细菌细胞膜外形成包膜而掩盖受体时,噬菌体产生水解酶降解包膜,使受体暴露从而再次吸附。细菌在不同时期、不同条件下可随机表达受体,相应地,噬菌体可修饰尾部受体结合蛋白,与不同时期和条件下表达的受体结合,以提高感染率[12]。

|
| 图 2 噬菌体针对细菌抗性的应变 Fig. 2 Bacteriophage responses on the bacteriophage-resistance of bacteria |
细菌产生RM系统来降解噬菌体的核酸,噬菌体则通过减少限制性位点以及抑制修饰酶等途径实现对细菌降解作用的逃避(图 2b)。一些噬菌体的基因中只有少量限制位点,或者相距太远而不被宿主的限制性核酸内切酶识别; 部分噬菌体则通过宿主或自身对基因进行甲基化,此时新合成的DNA将受到保护; 噬菌体P1的DarA和DarB蛋白可结合到噬菌体DNA上遮盖住限制位点,使限制性核酸内切酶无法识别到位点; 噬菌体T7的Ocr蛋白模仿靶DNA并隔离限制酶与甲基化酶和限制性核酸内切酶结合而抑制其活性[13]; 噬菌体λ的Ral蛋白激活甲基化酶活性并促进对噬菌体DNA的保护[14]; 某些噬菌体能编码水解酶,水解限制性核酸内切酶的辅因子从而抑制后者活性。
2.3 应对Abi和TA系统一些被感染的细菌通过“Abi”,以牺牲自我的方式限制噬菌体对邻近细菌的感染,噬菌体则可以通过基因突变绕过Abi(图 2c)。例如,乳球菌属某些噬菌体参与核苷酸代谢的基因发生突变,使得作用于代谢过程的磷酸化细胞因子失去活性,从而绕过Abi系统; 噬菌体可编码在功能上替代细菌抗毒素的分子,抵消毒素活性; 噬菌体ϕTE产生假抗毒素RNA或劫持天然抗毒素ToxI,以在噬菌体感染期间中和毒素ToxN[15],游离的ToxN减少将抑制Abi过程。
2.4 应对CRISPR-Cas系统细菌可通过CRISPR-Cas系统实现对噬菌体DNA的降解,而噬菌体则针对这一机制的一些关键点(如前间隔序列突变),抑制CRISPR-Cas效应复合物形成,从而保护自己免受杀灭(图 2d)[14]。例如,噬菌体原间隔序列或PAM序列突变,使得crRNA和靶DNA无法配对,起到干扰CRISPR-Cas系统的作用; 铜绿假单胞菌的溶原性噬菌体能编码抑制CRISPR-Cas复合物形成或抑制其活性的蛋白质,进而抑制CRISPR-Cas系统[16]。霍乱[11]弧菌中,噬菌体基因组进入细胞后表达病毒源性的crRNA和CRISPR-Cas复合物,以霍乱弧菌的PICI样因子(PICI-like elements,PLE)为靶点,噬菌体CRISPR基因座与PLE序列互补,能特异地使CRISPR-Cas系统失活[17]。
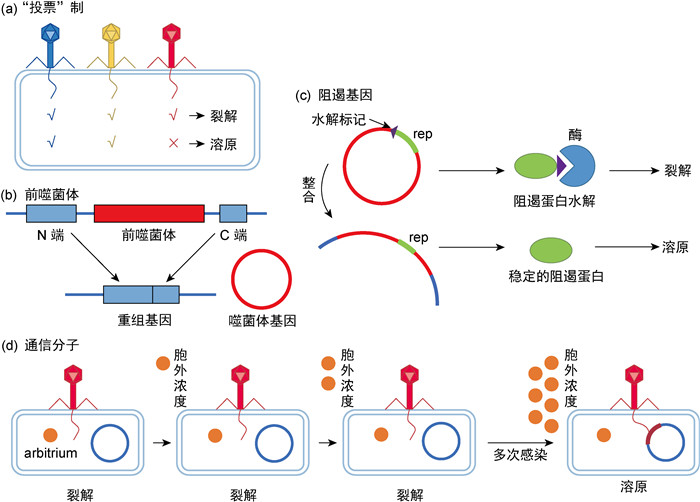
2.5 溶原决策机制噬菌体在感染宿主菌后是否进入溶原周期将受到许多因素的影响[18]。如图 3所示,①当多个噬菌体感染单个宿主菌时,所有感染的噬菌体溶原性的一致“投票(vote)”对最终建立溶原性十分必要(图 3a)[19]; ②前噬菌体可为细菌的基因开关。噬菌体与细菌基因组融合,从而使后者失活; 而当需要噬菌体基因时,便从细菌基因组中切除,重组为功能基因(图 3b)[21]; ③噬菌体阻遏基因(repressor,rep)对溶原态的建立有重要意义。噬菌体整合前,rep包含一个水解标记致使阻遏蛋白水解,促进裂解; 噬菌体整合后,水解标记分离,阻遏蛋白稳定表达,维持溶原态(图 3c); ④噬菌体感染宿主可释放arbitrium肽,胞外arbitrium肽浓度不断上升,导致噬菌体溶原态(图 3d)[20]。
|
| 图 3 溶原决策机制 Fig. 3 The lysogen decision mechanism |
噬菌体的研究为生命科学尤其是基于基因组测序的宏基因组研究奠定了重要基础。噬菌体与细菌的相互作用与它们的共同宿主——人体的健康息息相关。充分利用组学理论和实验技术,进一步探究噬菌体这一古老个体的结构、进化、功能等特点,不仅可为应对抗生素耐药细菌感染提供有力支持,还有望在噬菌体与细菌相互作用的人工干预、助力生物制品研制和生产进程等方面为促进人类健康开辟新的路径。
| [1] |
Hatfull GF, Hendrix RW. Bacteriophages and their genomes[J]. Curr Opin Virol, 2011, 1(4): 298-303.
[DOI]
|
| [2] |
Stone E, Campbell K, Grant I, McAuliffe O. Understanding and exploiting phage-host interactions[J]. Viruses, 2019, 11(6): 567.
[DOI]
|
| [3] |
Chen Y, Batra H, Dong J, Chen C, Rao VB, Tao P. Genetic engineering of bacteriophages against infectious diseases[J]. Front Microbiol, 2019, 10: 954.
[DOI]
|
| [4] |
Rostøl JT, Marraffini L. (Ph)ighting phages: how bacteria resist their parasites[J]. Cell Host Microbe, 2019, 25(2): 184-194.
[DOI]
|
| [5] |
Labrie SJ, Samson JE, Moineau S. Bacteriophage resistance mechanisms[J]. Nat Rev Microbiol, 2010, 8(5): 317-327.
[DOI]
|
| [6] |
Ofir G, Melamed S, Sberro H, Mukamel Z, Silverman S, Yaakov G, Doron S, Sorek R. DISARM is a widespread bacterial defence system with broad anti-phage activities[J]. Nat Microbiol, 2018, 3(1): 90-98.
[DOI]
|
| [7] |
Hille F, Richter H, Wong SP, Bratovi Ač M, Ressel S, Charpentier E. The Biology of CRISPR-Cas: backward and forward[J]. Cell, 2018, 172(6): 1239-1259.
[DOI]
|
| [8] |
Depardieu F, Didier JP, Bernheim A, Sherlock A, Molina H, Duclos B, Bikard D. A eukaryotic-like serine/threonine kinase protects staphylococci against phages[J]. Cell Host Microbe, 2016, 20(4): 471-481.
[DOI]
|
| [9] |
Fineran PC, Blower TR, Foulds IJ, Humphreys DP, Lilley KS, Salmond GP. The phage abortive infection system, ToxIN, functions as a protein-RNA toxin-antitoxin pair[J]. Proc Natl Acad Sci USA, 2009, 106(3): 894-899.
[DOI]
|
| [10] |
Tormo-Más MA, Mir I, Shrestha A, Tallent SM, Campoy S, Lasa I, Barbé J, Novick RP, Christie GE, Penadés JR. Moonlighting bacteriophage proteins derepress staphylococcal pathogenicity islands[J]. Nature, 2010, 465(7299): 779-782.
[URI]
|
| [11] |
Penadés JR, Christie GE. The phage-inducible chromosomal islands: a family of highly evolved molecular parasites[J]. Annu Rev Virol, 2015, 2(1): 181-201.
[DOI]
|
| [12] |
Dunne M, Rupf B, Tala M, Qabrati X, Ernst P, Shen Y, Sumrall E, Heeb L, Plückthun A, Loessner MJ, Kilcher S. Reprogramming bacteriophage host range through structure-guided design of chimeric receptor binding proteins[J]. Cell Rep, 2019, 29(5): 1336-1350.
[DOI]
|
| [13] |
Vasu K, Nagaraja V. Diverse functions of restriction-modification systems in addition to cellular defense[J]. Microbiol Mol Biol Rev, 2013, 77(1): 53-72.
[URI]
|
| [14] |
Labrie SJ, Samson JE, Moineau S. Bacteriophage resistance mechanisms[J]. Nat Rev Microbiol, 2010, 8(5): 317-327.
[DOI]
|
| [15] |
Samson JE, Magadán AH, Sabri M, Moineau S. Revenge of the phages: defeating bacterial defences[J]. Nat Rev Microbiol, 2013, 11(10): 675-687.
[DOI]
|
| [16] |
Thavalingam A, Cheng Z, Garcia B, Huang X, Shah M, Sun W, Wang M, Harrington L, Hwang S, Hidalgo-Reyes Y, Sontheimer EJ, Doudna J, Davidson AR, Moraes TF, Wang Y, Maxwell KL. Inhibition of CRISPR-Cas9 ribonucleoprotein complex assembly by anti-CRISPR AcrⅡC2[J]. Nat Commun, 2019, 10(1): 2806.
[DOI]
|
| [17] |
Naser IB, Hoque MM, Nahid MA, Tareq TM, Rocky MK, Faruque SM. Analysis of the CRISPR-Cas system in bacteriophages active on epidemic strains of Vibrio cholerae in Bangladesh[J]. Sci Rep, 2017, 7(1): 14880.
[DOI]
|
| [18] |
Howard-Varona C, Hargreaves KR, Abedon ST, Sullivan MB. Lysogeny in nature: mechanisms, impact and ecology of temperate phages[J]. ISME J, 2017, 11(7): 1511-1520.
[DOI]
|
| [19] |
Zeng L, Skinner SO, Zong C, Sippy J, Feiss M, Golding I. Decision making at a subcellular level determines the outcome of bacteriophage infection[J]. Cell, 2010, 141(4): 682-691.
[DOI]
|
| [20] |
Erez Z, Steinberger-Levy I, Shamir M1, Doron S, Stokar-Avihail A, Peleg Y, Melamed S, Leavitt A, Savidor A, Albeck S, Amitai G1, Sorek R. Communication between viruses guides lysis-lysogeny decisions[J]. Nature, 2017, 541(7638): 488-493.
[DOI]
|
| [21] |
Feiner R, Argov T, Rabinovich L, Sigal N, Borovok I, Herskovits AA. A new perspective on lysogeny: prophages as active regulatory switches of bacteria[J]. Nat Rev Microbiol, 2015, 13(10): 641-650.
[DOI]
|
 2020, Vol. 15
2020, Vol. 15


